Importar Localidades
8 minute read
OpenDataBio é distribuído com um conjunto de dados de localidades para o Brasil, que inclui estados, municípios, unidades de conservação federais, terras indígenas e os biomas.
Trabalhar com dados espaciais é uma área delicada, por isso tentamos tornar o fluxo de trabalho para inserir Localidades o mais fácil possível.
Se você deseja fazer upload dos limites administrativos de um país, você também pode baixar um arquivo geojson em OSM-Boundaries e carregue-o diretamente através da interface da web. Ou use o repositório GADM exemplificado abaixo.
A importação é direta, mas os principais problemas a serem considerados:
- OpenDataBio armazena as geometrias de localidades usando representação de texto conhecido (WKT).
- As localidades são hierárquicas, portanto, uma localidade DEVE estar completamente dentro de sua localidade pai. O método de importação tentará detectar as localidades pai com base em sua geometria. Portanto, você não precisa informar um pai. No entanto, às vezes a localidade pai e a localidade filho compartilham uma borda ou têm pequenas erros que evitam a detecção. Portanto, se a importação não colocar o local onde você esperava, pode-se atualizar ou importar informando o pai correto. Quando você informar a localidade pai, uma segunda verificação será realizada adicionando um buffer à localidade pai e deverá resolver o problema.
- Os polígonos de países podem ser importados sem detecção ou definição dos pais, e registros marítimos podem ser vinculados a um pai, mesmo que não estejam contidos no polígono pai. Isso requer informar um campo específico (
ismarine) e deve ser usado nestes casos. - Padronizar a geometria para uma projeção comum de uso no sistema. Fortemente recomendado o uso de EPSG:4326 WGS84. Padronize antes de importar.
- Considere enviar seus polígonos político-administrativos antes de adicionar PONTOS, PLOTS ou TRANSECTOS específicos;
- Unidades de Conservação, Territórios Indígenas e Camadas Ambientais podem ser adicionados como locais e serão tratados como casos especiais, pois alguns desses locais abrangem diferentes unidades administrativas. Portanto, uma localidade de tipo POINT, PLOT ou TRANSECTpode pertencer a umA UC, umA TI e muitas camadas ambientais se estas estiverem armazenadas no banco de dados. Essas localidades relacionadas são detectadas automaticamente a partir da geometria da localidade.
Verifique a POST API de localidades para entender quais colunas podem ser declaradas ao importar localidades.
Adm_level define o tipo de localidade
O nível administrativo (adm_level) de um local é um número:
2para país;3à10como outras como ‘áreas administrativas’, seguindo a convenção OpenStreeMap para facilitar a importação de dados externos e traduções locais (À SER IMPLEMENTADO) . Portanto, para o Brasil, os códigos são (Estados = 4, Municípios = 8);999para locais de ‘POINT’ como waypoints GPS;101para transectos100é o código para PARCELAS e SUBPARCELAS;99é o código para Unidades de Conservação98para Territórios Indígenas97para polígonos ambientais (por exemplo, Floresta Ombrofila Densa ou Bioma Amazônia)
Importando polígonos espaciais
Limites administrativos do GADM
Limites administrativos também podem ser importados sem sair de R, obtendo dados de GDAM e usando as funções odb_import*
library(raster)
library(opendatabio)
#download áreas administrativas do GADM para um país
#get country codes
crtcodes = getData('ISO3')
bra = crtcodes[crtcodes$NAME%in%"Brazil",]
#define a path where to save the downloaded spatial data
path = "GADMS"
dir.create(path,showWarnings = F)
#o número de admin_levels em cada país varia
#obter todos os níveis existentes em seu computador
runit =T
level = 0
while(runit) {
ocrt <- try(getData('GADM', country=bra, level=level,path=path),silent=T)
if (class(ocrt)=="try-error") {
runit = FALSE
}
level = level+1
}
#read downloaded data and format to odb
files = list.files(path, full.name=T)
locations.to.odb = NULL
for(f in 1:length(files)) {
ocrt <- readRDS(files[f])
#class(ocrt)
#convert the SpatialPolygonsDataFrame to OpenDataBio format
ocrt.odb = opendatabio:::sp_to_df(ocrt) #only for GADM data
locations.to.odb = rbind(locations.to.odb,ocrt.odb)
}
#see without geometry
head(locations.to.odb[,-ncol(locations.to.odb)])
#you may add a note to location
locations.to.odb$notes = paste("Source gdam.org via raster::get_data()",Sys.Date())
#adjust the adm_level to fit the OpenStreeMap categories
ff = as.factor(locations.to.odb$adm_level)
(lv = levels(ff))
levels(ff) = c(2,4,8,9)
locations.to.odb$adm_level = as.vector(ff)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=locations.to.odb,odb_cfg=cfg)
Atenção: você pode querer verificar se há exclusividade de nome + pai em vez de apenas nome, já que nome + pai é uma combinação única. Você não pode salvar dois locais com o mesmo nome dentro do mesmo pai.
Example usando um shapefile
library(rgdal)
#read your shape file
path = 'mymaps'
file = 'myshapefile.shp'
layer = gsub(".shp","",file,ignore.case=TRUE)
data = readOGR(dsn=path, layer= layer)
#you may reproject the geometry to standard of your system if needed
data = spTransform(data,CRS=CRS("+proj=longlat +datum=WGS84"))
#convert polygons to WKT geometry representation
library(rgeos)
geom = rgeos::writeWKT(data,byid=TRUE)
#prep import
names = data@data$name #or the column name of the data
shape.to.odb = data.frame(name=names,geom=geom,stringsAsFactors = F)
#need to add the admin_level of these locations
shape.to.odb$admin_level = 2
#and may add parent and note if your want
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=shape.to.odb,odb_cfg=cfg)
Example importando de um KML
#read file as SpatialPolygonDataFrame
file = "myfile.kml"
file.exists(file)
mykml = readOGR(file)
geom = rgeos::writeWKT(mykml,byid=TRUE)
#prep import
names = mykml@data$name #or the column name of the data
to.odb = data.frame(name=names,geom=geom,stringsAsFactors = F)
#need to add the admin_level of these locations
to.odb$admin_level = 2
#and may add parent or any other valid field
#import
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=to.odb,odb_cfg=cfg)
Importar Parcelas e SubParcelas
Parcelas e transectos são casos especiais no OpenDataBio:
- Eles podem ser definidas com uma geometria do tipo Polygon ou LineString, respectivamente;
- Ou eles podem ser registrados apenas como localidaes de tipo POINT. Nesse caso, o OpenDataBio criará o polígono ou linestring para você;
- Dimensões (x e y) são armazenadas em metros para PARCELAS. Portanto elas devem ser quadradas ou retangulares.
- SubParcelas são localidades do tipo PARCELA tendo outra localidade do tipo PARCELA como pai e também devem ter posições cartesianas (startX, startY) dentro da localidade pai além das dimensões. A posição cartesiana refere-se às posições X e Y dentro da PARCELA pai e, portanto, DEVE ser menor do que o pai X e Y.
- SubParcela é o único tipo de localidade que pode ser registrado sem uma coordenada geográfica ou geometria, que será calculada a partir da geometria da PARCELA pai usando os valores startx e starty.
Parcela e SubParcela - exemplo 01
Você precisa de pelo menos uma coordenada geográfica para registrar uma localidade do tipo PLOT. A geometria (ou latitude e longitude) não pode estar vazia.
Este exemplo registra uma parcela em Manaus de 100x100m, informando sua geometria e depois importa algumas subparcelas sem especificação de geometria.
#geometry of a plot in Manaus
southWestCorner = c(-59.987747, -3.095764)
northWestCorner = c(-59.987747, -3.094822)
northEastCorner = c(-59.986835,-3.094822)
southEastCorner = c(-59.986835,-3.095764)
geom = rbind(southWestCorner,northWestCorner,northEastCorner,southEastCorner)
library(sp)
geom = Polygon(geom)
geom = Polygons(list(geom), ID = 1)
geom = SpatialPolygons(list(geom))
library(rgeos)
geom = writeWKT(geom)
to.odb = data.frame(name='A 1ha example plot',x=100,y=100,notes='a fake plot',geom=geom, adm_level = 100,stringsAsFactors=F)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=to.odb,odb_cfg=cfg)
Aguarde alguns segundos e, em seguida, importe subtramas para esta plotagem.
#importar subparcelas de 20x20m para a PARCELA acima sem indicar uma geometria.
(parent = odb_get_locations(params = list(name='A 1ha example plot',fields='id,name',adm_level=100),odb_cfg = cfg))
sub1 = data.frame(name='sub plot 40x40',parent=parent$id,x=20,y=20,adm_level=100,startx=40,starty=40,stringsAsFactors=F)
sub2 = data.frame(name='sub plot 0x0',parent=parent$id,x=20,y=20,adm_level=100,startx=0,starty=0,stringsAsFactors=F)
sub3 = data.frame(name='sub plot 80x80',parent=parent$id,x=20,y=20,adm_level=100,startx=80,starty=80,stringsAsFactors=F)
dt = rbind(sub1,sub2,sub3)
#import
odb_import_locations(data=dt,odb_cfg=cfg)
Capturas de tela das parcelas importadas
Abaixo capturas de tela para as parcelas importadas com o código acima
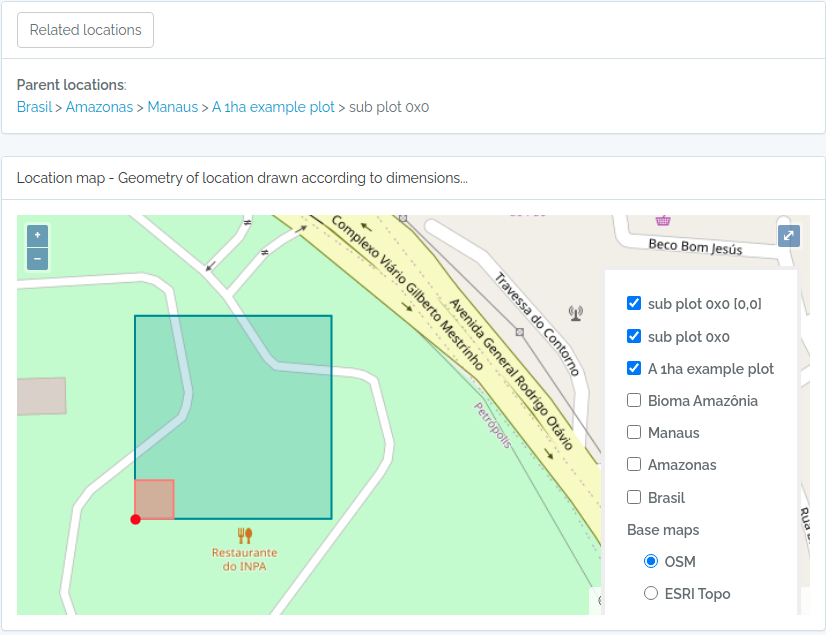
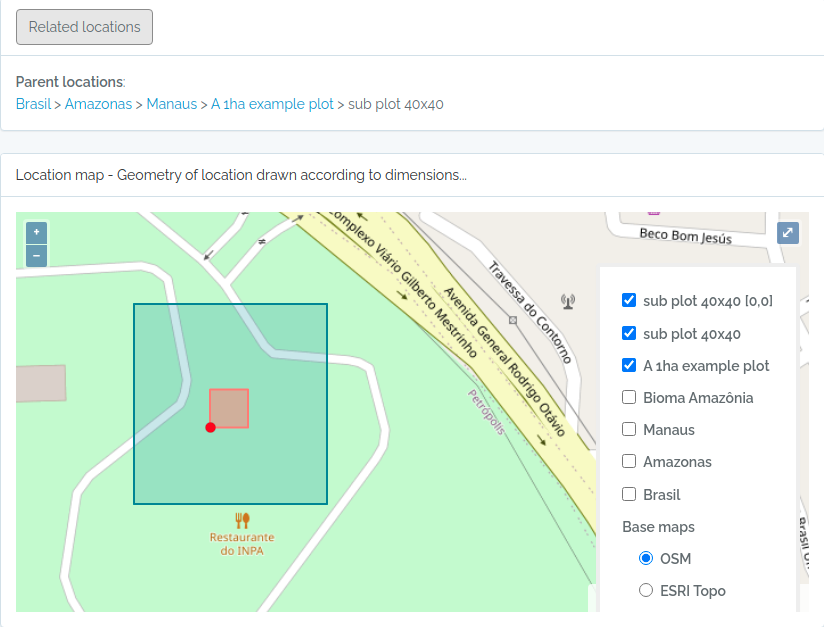
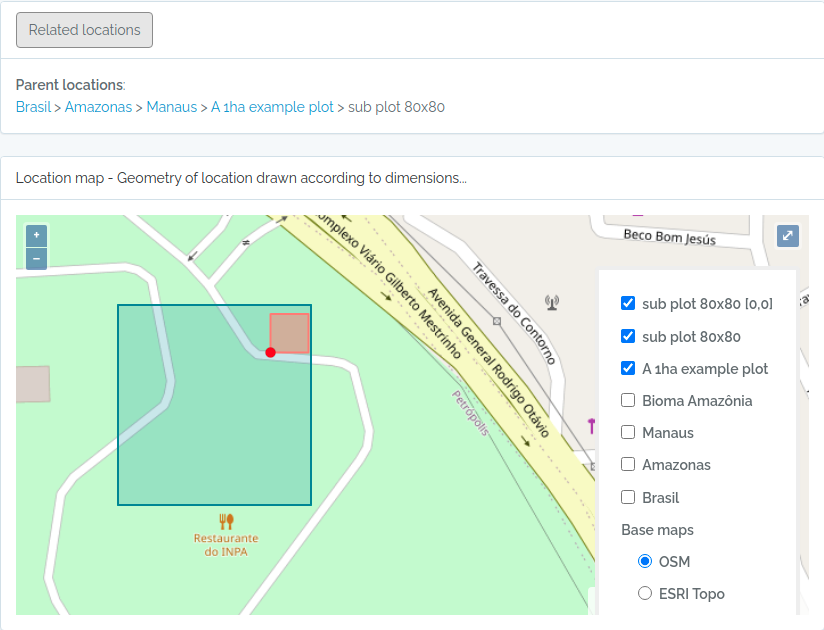
Parcela e SubParcela - exemplo 02
Importe uma parcela e suas subparcelas tendo apenas:
- a coordenada geográfica de um único ponto, representando a coordenada [0,0] da parcela.
- um azimute ou ângulo da direção da parcela (se não informar, Norte será usado
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
#the plot
geom = "POINT(-59.973841 -2.929822)"
to.odb = data.frame(name='Example Point PLOT',x=100, y=100, azimuth=45,notes='OpenDataBio point plot example',geom=geom, adm_level = 100,stringsAsFactors=F)
odb_import_locations(data=to.odb,odb_cfg=cfg)
#define 20x20 subplots cartesian coordinates
x = seq(0,80,by=20)
xx = rep(x,length(x))
yy = rep(x,each=length(x))
names = paste(xx,yy,sep="x")
#importar esses subplots sem ter uma geometria, mas especificando a localidade pai
parent = odb_get_locations(params = list(name='Example Point PLOT',adm_level=100),odb_cfg = cfg)
to.odb = data.frame(name=names,startx=xx,starty=yy,x=20,y=20,notes="OpenDataBio 20x20 subplots example",adm_level=100,parent=parent$id)
odb_import_locations(data=to.odb,odb_cfg=cfg)
#obter os locais importados usando o parâmetro root
locais = odb_get_locations(params=list(root=parent$id),odb_cfg = cfg)
locais[,c('id','locationName','parentName')]
colnames(locais)
for(i in 1:nrow(locais)) {
geom = readWKT(locais$footprintWKT[i])
if (i==1) {
plot(geom,main=locais$locationName[i],cex.main=0.8,col='yellow')
axis(side=1,cex.axis=0.7)
axis(side=2,cex.axis=0.7,las=2)
} else {
plot(geom,add=T,border='red')
}
}
A figura gerada acima:
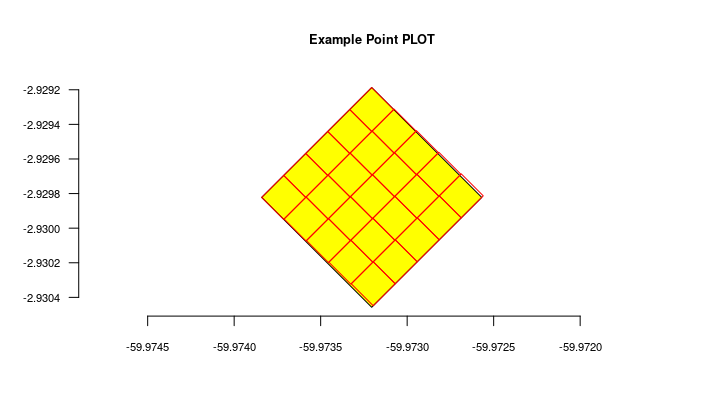
Importar transectos
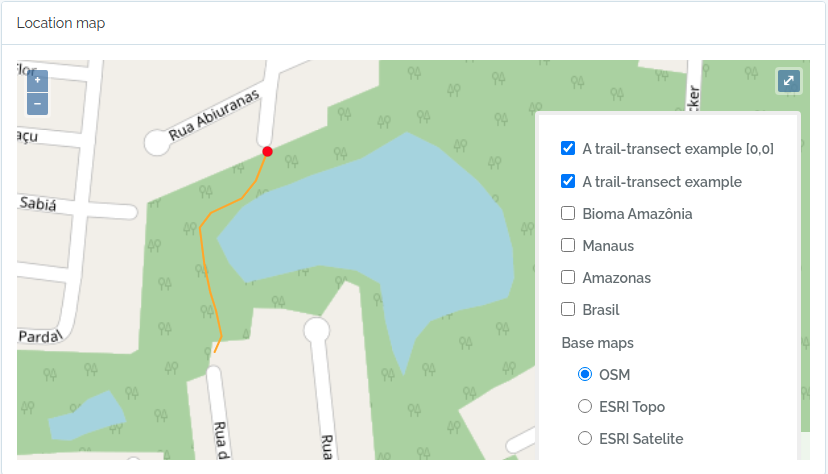
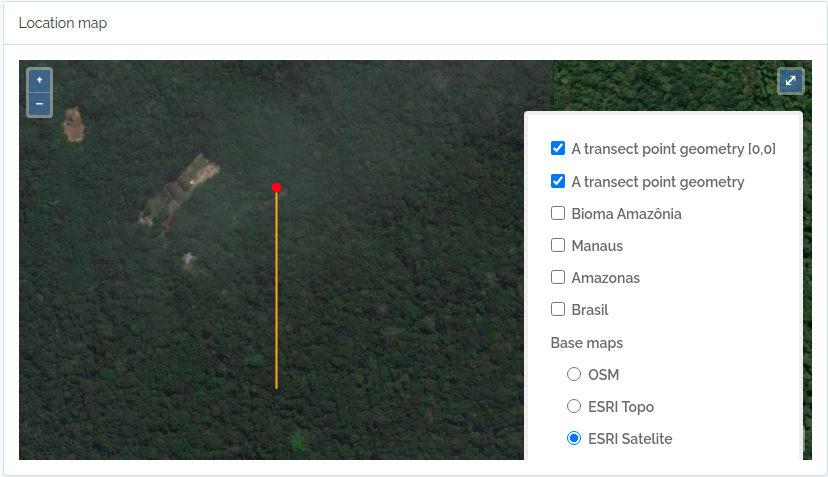
Este código importará dois transectos, um definido por uma geometria (LINESTRING), e o outro apenas por uma única coordenada geográfica (POINT). Veja as figuras abaixo para o resultado importado.
#geometry of transect in Manaus
#read trail from a kml file
#library(rgdal)
#file = "acariquara.kml"
#file.exists(file)
#mykml = readOGR(file)
#library(rgeos)
#geom = rgeos::writeWKT(mykml,byid=TRUE)
#above will output:
geom = "LINESTRING (-59.9616459699999993 -3.0803612500000002, -59.9617394400000023 -3.0805952900000002, -59.9618530300000003 -3.0807376099999999, -59.9621049400000032 -3.0808563200000001, -59.9621949100000009 -3.0809758500000002, -59.9621587999999974 -3.0812666800000001, -59.9621092399999966 -3.0815010400000000, -59.9620656999999966 -3.0816403499999998, -59.9620170600000009 -3.0818584699999998, -59.9620740699999999 -3.0819864099999998)";
#prep data frame
#o valor y refere-se a um buffer em metros aplicado à trilha
#y é usado para validar a inserção de indivíduos relacionados
to.odb = data.frame(name='A trail-transect example',y=20, notes='OpenDataBio transect example',geom=geom, adm_level = 101,stringsAsFactors=F)
#import
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=to.odb,odb_cfg=cfg)
#IMPORTA UM SEGUNDO TRANSECTO SEM GEOMETRIA
# então você precisa informar o valor x, que é o comprimento do transecto
#ODB irá mapear este transecto orientado pelo parâmetro azimute (sul no exemplo abaixo)
#point geometry = ponto inicial
geom = "POINT(-59.973841 -2.929822)"
to.odb = data.frame(name='A transect point geometry',x=300, y=20, azimuth=180,notes='OpenDataBio point transect example',geom=geom, adm_level = 101,stringsAsFactors=F)
odb_import_locations(data=to.odb,odb_cfg=cfg)
locais = odb_get_locations(params=list(adm_level=101),odb_cfg = cfg)
locais[,c('id','locationName','parentName','levelName')]
O código acima resultará nas duas localidades a seguir: