Modelo conceitual
Visão geral sobre a estrutura da base de dados e das relações entre modelos!
Se você quiser ajudar no desenvolvimento, leia com atenção o modelo conceitual do OpenDataBio antes de começar a colaborar.
Para facilitar a compreensão dos conceitos, já que incluem muitas tabelas e relacionamentos complexos, o modelo de dados OpenDataBio está aqui dividido nas quatro categorias listadas abaixo.
1 - Objetos Centrais
Objetos que podem receber Medições de Váriaveis dos usuários
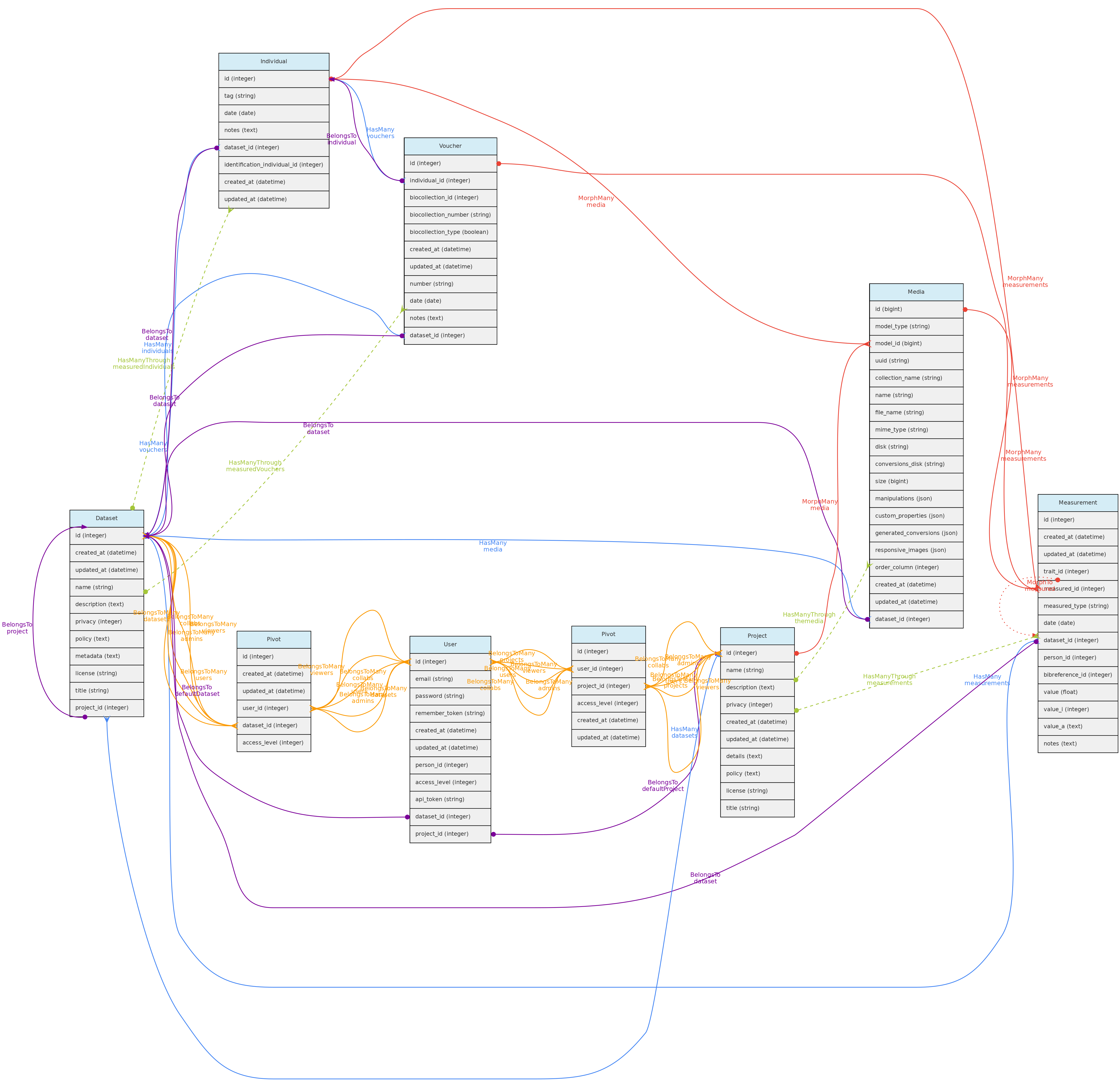
Os objetos centrais são: Localidades, Vouchers, Indivíduos e Taxons. Essas entidades são consideradas “centrais” porque podem receber Medições, ou seja, você pode registrar valores para qualquer Variável.
-
O objeto Indivíduo refere-se a um organismo individual que foi observado uma vez (uma ocorrência) ou foi marcado para monitoramento, como uma árvore em uma parcela permanente, uma ave anilhada, um morcego rastreado por rádio. Os indivíduos podem ter um ou mais Vouchers em uma BioColeção e um ou vários Localidades e terão uma Identificação taxonômica. Qualquer atributo medido ou tomado para um individuo pode ser associado a este objeto por meio do modelo Medição.
-
O objeto Vouchers é para registros de espécimes coletados de Indivíduo e depositados em uma BioColeção. A identificação taxonômica e a localização de um voucher é aquela do próprio indivíduo a que pertence. Medições podem ser vinculadas a um Voucher quando você deseja registrar explicitamente os dados para aquela amostra específica (por exemplo, medições morfológicas; um marcador molecular de uma extração de uma amostra em um coleta de tecido). Caso contrário, você pode simplesmente registrar a medição para o indivíduo ao qual o voucher pertence. O modelo de voucher também está disponível como tipo especial de Variável, o LinkType, tornando possível registrar contagens para o táxon do voucher em um determinado local.
-
O objeto Localidades contém geometrias espaciais, como pontos e polígonos, e inclui parcelas e transectos como casos especiais. Um Indivíduo pode ter uma localidade (por exemplo, uma planta) ou multiplas localidades (por exemplo, um animal monitorado). As localidades do tipo PARCELA e TRANSECTO podem ser registradas como geometria espacial ou apenas com geometria de ponto, e podem ter dimensões cartesianas (metros) registradas. Os indivíduos também podem ter posições cartesianas (X e Y ou angle e distance) em relação à sua Localidade, permitindo contabilizar o mapeamento tradicional de indivíduos em unidades de amostragem. Medições ecológicas relevantes, como dados de solo ou clima, são exemplos de Medições que podem ser vinculadas a localidades.
-
O objeto Taxon, além de seu uso para a Identificação taxonômica de Indivíduos, pode receber Medições, permitindo a organização de dados secundários publicados ou qualquer tipo de informação ligada a um nome taxonômico. Uma Referência Bibliográfica pode ser incluída para indicar a fonte de dados. Além disso, o modelo Taxon está disponível como tipo especial de Variável, o LinkType, tornando possível registrar contagens de Taxons em um determinado local.
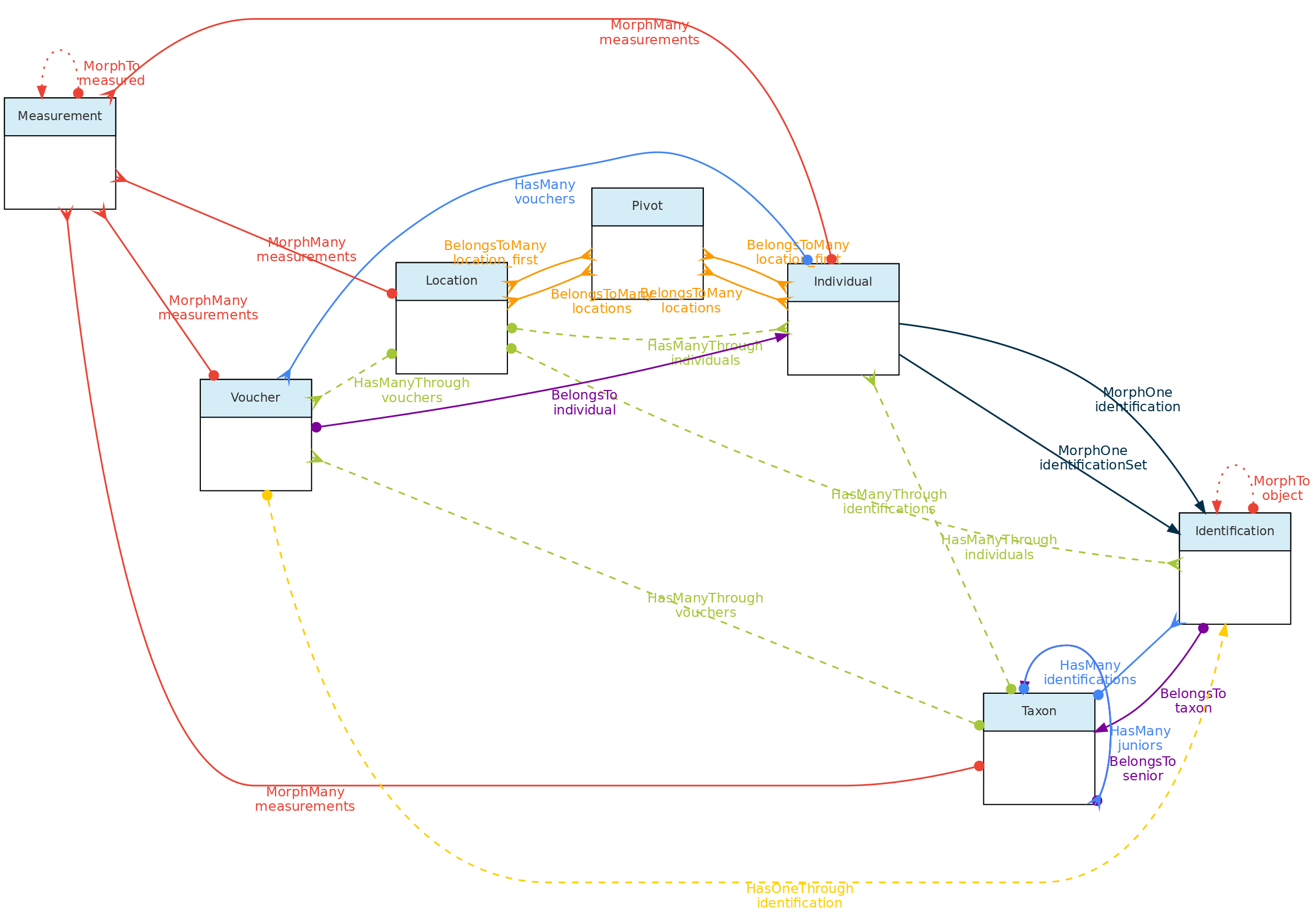
Esta figura mostra as relações entre os objetos centrais e com o modelo Medições. O Identificação também está incluído para maior clareza. Links sólidos são relacionamentos diretos, enquanto links tracejados são relacionamentos indiretos (por exemplo, os táxons têm muitos vouchers por meio de indivíduos e muitos indivíduos por meio de identificações). As linhas sólidas vermelhas vinculam os Objetos centrais com o modelo de Medição por meio de relações polimórficas. As linhas pontilhadas no modelo de Medição apenas permitem o acesso ao objeto central medido e aos modelos de variáveis do tipo de link.
Localidades
O modelo Localidades armazena dados que representam locais do mundo real. Eles podem ser países, cidades, unidades de conservação ou qualquer polígono espacial, ponto ou trilha na superfície da Terra. Esses objetos são hierárquicos e possuem um relacionamento pai-filho implementado pelo Nested Set Model para dados hierárquicos da biblioteca Laravel Baum que facilita tanto a validação quanto as consultas.
Tipos de Localidades especiais são parcelas e transectos, que juntamente com pontos permitem diferentes métodos de amostragem usados em estudos de biodiversidade. Esses tipos de Localidades também podem ser vinculados a uma localidade administrativa pai e, além disso, a três tipos adicionais de localidades pai como Unidades de Conservação, Territórios Indígenas e qualquer camada Ambiental representando classes de vegetação, classes de solo , etc … com geometrias espaciais definidas.
Indivíduo
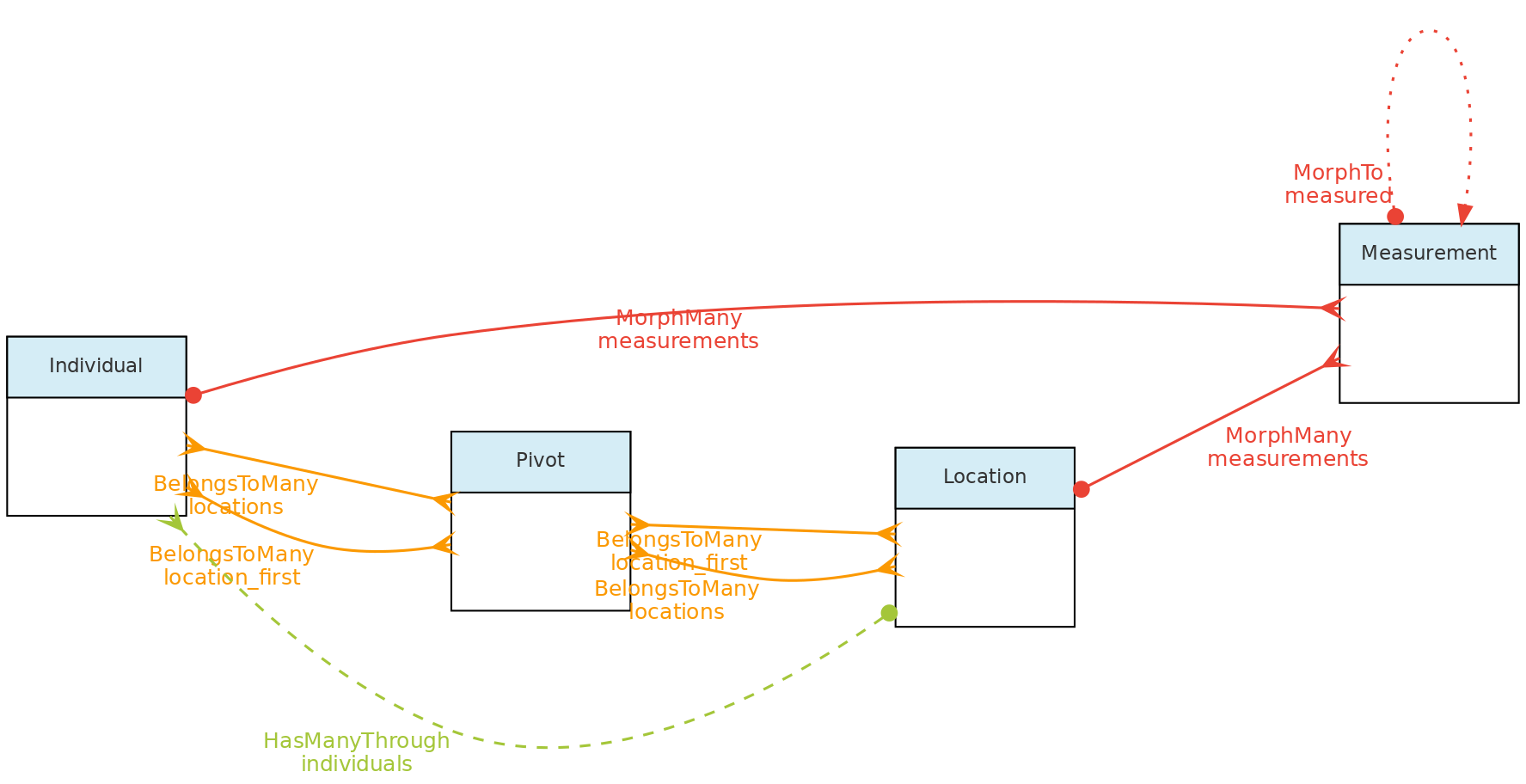 Esta figura mostra os relacionamentos do modelo
Esta figura mostra os relacionamentos do modelo Location através dos métodos implementados nas classes mostradas. A tabela dinâmica que vincula Localidade a Indivíduos permite que um indivíduo tenha vários locais e cada local para o indivíduo tenha atributos específicos como date_time, altitude, relative_position e notes.
 As mesmas tabelas relacionadas com o modelo
As mesmas tabelas relacionadas com o modelo Location com as relações diretas e não polimórficas indicadas.
Tabela locations
- As colunas
parent_id junto com rgt, lft e deph são usadas para definir o modelo de conjunto aninhado para consultar ancestrais e descendentes de forma rápida. Apenas parent_id é especificado pelo usuário, as outras colunas são calculadas pela biblioteca Baum a partir dos valores id+parent_id que definem a hierarquia. O mesmo modelo hierárquico é usado para o Taxons, mas para locais há uma restrição espacial, ou seja, um filho deve estar dentro de uma geometria pai.
- A coluna
adm_level indica o nível administrativo, ou tipo, de localidade. Por padrão, os seguintes adm_level são configurados no OpenDataBio:
2 para o país, 3 para a primeira divisão dentro do país (província, estado), 4 para a segunda divisão (por exemplo, município), … até adm_level = 10 como áreas administrativas (o código do país é 2 para permitir a padronização com OpenStreeMaps, que é recomendado seguir se sua instalação incluir dados de diferentes países). Os níveis administrativos podem ser configurados em um OpenDataBio antes de importar quaisquer dados para o banco de dados, consulte o guia de instalação para obter detalhes sobre isso.99 é o código para Unidades de Conservação - uma unidade de conservação é uma location que pode estar vinculada a vários outros locais (qualquer local pode pertencer a uma única UC). Assim, uma localidade pode ter como pai um município e como uc a unidade de conservação a que pertence.98 é o código para Territórios Indígenas - mesmas propriedades das Unidades de Conservação, mas tratadas separadamente apenas porque algumas UCs e TIs podem se sobrepor amplamente como é o caso da região amazônica97 é o código para Camadas ambientais - mesmas propriedades das Unidades de Conservação e Territórios Indígenas, ou seja, podem ser vinculadas como localidade pai adicional a qualquer Ponto, Parcela ou Transecto e, portanto, seus indivíduos relacionados. Armazene polígonos e geometrias de multipolígonos representando classes ambientais, como unidades de vegetação, biomas, classes de solo, etc …100 é o código para parcelas e sub-parcelas - as localidades de tipo parcelas podem ser registradas com geometria de ponto ou polígono, e também devem ter dimensões cartesianas associadas em metros. Se for uma localização de ponto, a geometria é definida a partir do ponto informado. As dimensões cartesianas de uma localidade de tipo parcela também podem ser combinadas com posições cartesianas de subparcelas (ou seja, uma localidade de parcela cujo pai também é uma localidade de parcela) e/ou de indivíduos dentro de tais parcelas, permitindo que indivíduos e subparcelas sejam mapeados dentro de uma subparcela sem especificações de geometria. Em outras palavras, se a geometria espacial da Parcela for desconhecida, ela pode ter como geometria um único ponto GPS ao invés de um polígono, mais suas dimensões x e y. Uma subparcela é uma parcela cuja localidade pai também é uma parcela, e deve consistir em um ponto marcando o início da subparcela mais suas dimensões cartesianas X e Y. Se a geometria do início da subplot for subparcela, ela pode ser armazenada como uma posição relativa à parcela pai usando startx e starty.101 para transectos - como parcelas, os transectos podem ser registrados tendo uma geometria LineString ou simplesmente uma única coordenada de Latitude e Longitude e uma dimensão. A dimensão cartesiana x para transectos representa o comprimento em metros e é usada para criar linha (orientada para o Norte) quando apenas um ponto é informado. A dimensão y é usada para validar os indivíduos como pertencentes à uma localidade do tipo transecto e representa a distância máxima da linha que um indivíduo deve cair para ser detectado naquele local.999 para localidades ‘POINT’ como waypoints GPS - isto é para registro de qualquer ponto no espaço
- A coluna
datum pode registrar a propriedade do datum da geometria, se conhecida. Se deixado em branco, o local é considerado armazenado usando o datum WGS84. Porém, não há conversor embutido de outros tipos de dados. Portanto os mapas exibidos podem ficar incorretos se diferentes projeções forem usadas. Fortemente recomendado projetar dados como WSG84 para padronização.
- A coluna
geom armazena a geometria da localização no banco de dados, permitindo consultas espaciais em linguagem SQL, como detecção das localidades pai. A geometria de um local pode ser POINT, POLYGON, MULTIPOLYGON ou LINESTRING e deve ser formatada usando Well-Known-Text representação geométrica da localidade. Quando um POLYGON é informado, o primeiro ponto dentro da string geométrica é privilegiado, ou seja, pode ser utilizado como referência para marcações relativas. Por exemplo, tal ponto será a referência para as colunas startx e starty de uma subparcela. Portanto, para as geometrias plot e transect, importa qual ponto é listado primeiro na geometria WKT
Acesso a dados usuários completos podem registrar novas Localidades, editar detalhes de Localidades e remover registros de Localidades que não têm dados associados. As Localidades têm acesso público.
Indivíduos
O objeto Indivíduo representa um registro para um organismo individual. Pode ser uma única ocorrência no espaço-tempo de um animal, planta ou fungo, ou um indivíduo monitorado ao longo do tempo, como uma planta em parcela florestal permanente, ou um animal de captura-recaptura ou rádio-rastreamento.
Um Indivíduo pode ter um ou mais Vouchers representando amostras físicas do indivíduo armazenadas em uma ou mais BioColeção e pode ter um ou mais Localidades, representando o local ou locais onde o indivíduo foi registrado.
Indivíduos também podem ter uma Identificação taxonômica, que pode ser própria ou pode depender da identificação de outro indivíduo (identificação-dependente). A identificação é herdada por todos os Vouchers registrados para o Indivíduo. Portanto, os vouchers não têm sua identificação própria.
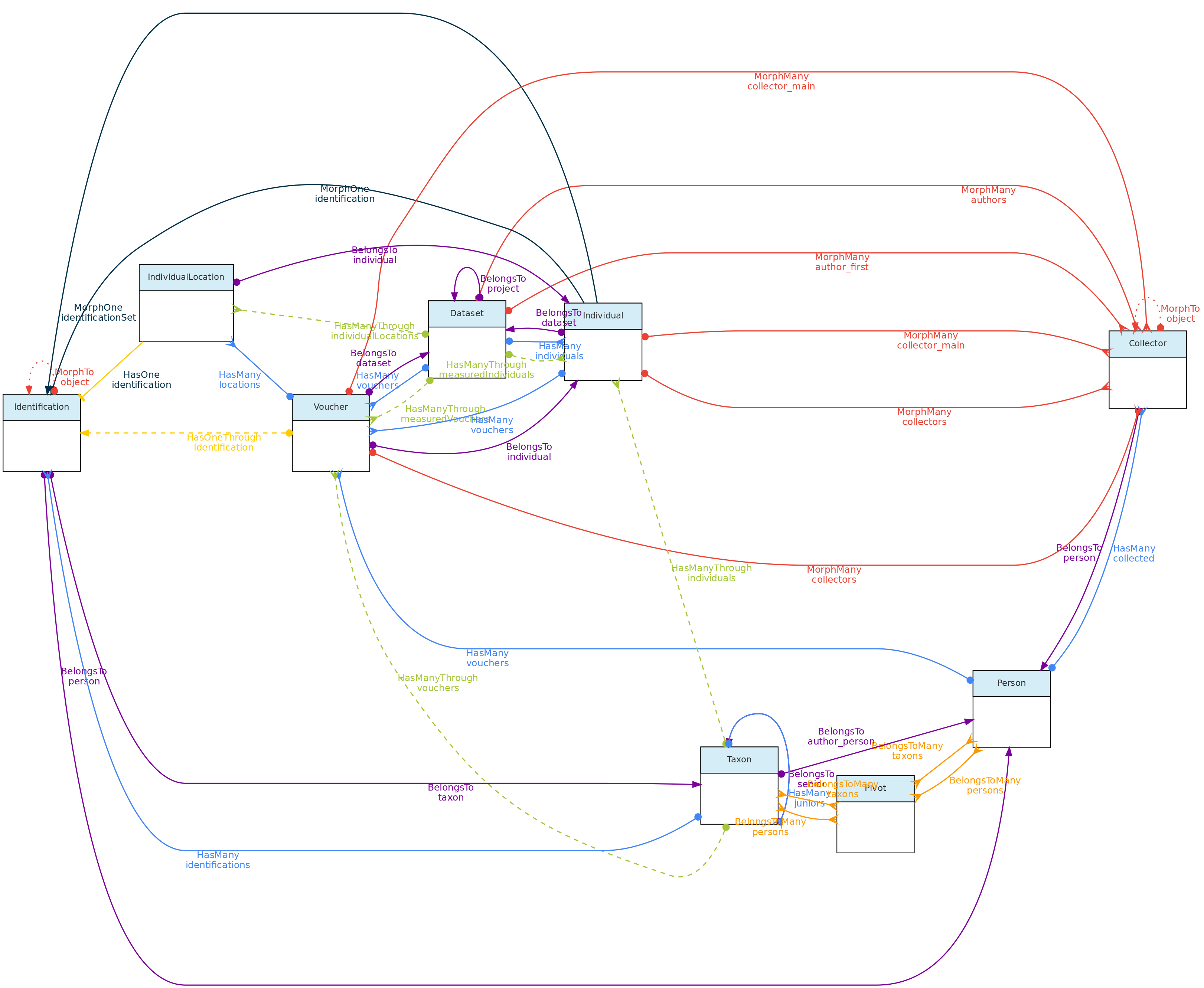 Esta figura mostra o Modelo Indivíduo e os modelos aos quais ele se relaciona, exceto os modelos de Medição e Localidades, já que seus relacionamentos com Indivíduos são mostrados em outra parte desta página. As linhas indicam os
Esta figura mostra o Modelo Indivíduo e os modelos aos quais ele se relaciona, exceto os modelos de Medição e Localidades, já que seus relacionamentos com Indivíduos são mostrados em outra parte desta página. As linhas indicam os métodos ou funções implementadas nas classes para acessar os relacionamentos. As linhas tracejadas indicam relações indiretas e as cores os diferentes tipos de métodos do Laravel Eloquent.
 As mesmas tabelas relacionadas com o modelo Indivíduo, suas colunas e as relações diretas e não polimóficas entre elas.
As mesmas tabelas relacionadas com o modelo Indivíduo, suas colunas e as relações diretas e não polimóficas entre elas.
Tabela individuals
- Um registro de um Indivíduo deve especificar pelo menos uma Localidade onde foi registrado, a
date do registro, o identificador local tag e o collectors do registro e o dataset ao qual o indivíduo pertence.
- A localidade pode ser qualquer localidade cadastrado, independente do nível, permitindo armazenar registros históricos cujo georreferenciamento é apenas um local administrativo. Localidades são armazenadas na tabela
individual_location, tendo colunas date_time, altitude,notes e relative_position.
- A coluna
relative_position armazena as coordenadas cartesianas do Indivíduo em relação à sua localidade. Isso é apenas para indivíduos localizados em locais do tipo plot, transect ou point. Por exemplo, uma parcela com dimensões de 100x100 metros (1ha) pode ter um indivíduo com posição relativa = PONTO (50 50), que colocará o indivíduo no centro do local (isso é mostrado graficamente na interface da web conforme definido pelas coordenadas x e y do indivíduo). Se a localidade for uma subparcela, a posição dentro da parcela pai também pode ser calculada (isso foi projetado com as parcelas do ForestGeo em mente e é uma coluna na API individual GET. Se a localidade for um PONTO, a posição_relativa pode ser informada como angle (= azimute) e distance, atributos frequentemente medidos em métodos de amostragem. Se a localidade for um TRANSECT, a posição_relativa posiciona o indivíduo em relação à linha, sendo x a distância ao longo do transecto a partir do primeiro ponto, e o y a distância perpendicular onde o indivíduo está localizado, também levando em consideração alguns métodos de amostragem;
- O campo
date nos modelos Indivíduo, Voucher, Medição e Identificação pode ser uma Data Incompleta, ou seja, apenas o ano ou ano + mês podem ser registrados.
- A tabela Collector representa os coletores para um indivíduo ou voucher e está vinculada ao Modelo de pessoa. A tabela de coletores possui uma relação polimórfica com os objetos Voucher e Individual, definidos pelas colunas
object_id e object_type, permitindo múltiplos coletores para cada registro individual ou voucher. O main_collector indicado é apenas o primeiro coletor listado para essas entidades.
- O campo
tag é um código identificador para o indivíduo. Pode ser o número escrito na etiqueta de alumínio de uma árvore em uma parcela florestal, o número de uma anilha numa ave, ou o número de coletor de um espécime. A combinação de main_collector + tag + first_location é restrita a ser única no OpenDataBio.
A identificação taxonômica de um indivíduo pode ser definida de duas maneiras:
- para identificação-própria um registro de Identificação taxonômica é criado na tabela de identifications e a coluna
identification_individual_id é preenchida com o próprio id do indivíduo
- para identificação-dependente, o id do Indivíduo que possui a Identificação é armazenado na coluna
identification_individual_id.
- Conseqüentemente, o modelo Indivíduo contém dois métodos para se relacionar com o modelo de Identificação: um que define identificação-própria e outro que recupera as identificações taxonômicas usando a coluna
identification_individual_id.
- Os indivíduos podem ter um ou mais Vouchers depositados em uma BioColeção.
Acesso a dados Indivíduos pertencem a conjuntos de dados, então a política de acesso do conjuntos de dados se aplica aos indivíduos nele inseridos. Apenas os colaboradores e administradores do Conjunto de Dados podem inserir ou editar indivíduos, mesmo se o conjunto de dados for de acesso público.
Taxons
A ideia geral por trás do modelo Taxon é ferramentas para incorporar facilmente nomes taxonômicos válidos de repositórios Taxonômicos Online (atualmente Tropicos.org, GBIF e ZOOBANK estão implementados), mas permitindo a inclusão de nomes que também não são considerados válidos porque eles ainda são não publicados (por exemplo, um morfotipo), ou o usuário discorda da sinonímia publicada, ou o usuário deseja que todos os sinônimos sejam registrados como táxons inválidos no sistema. Além disso, permite definir um nível taxonômico de tipo clado, permitindo armazenar, além das categorias de classificação taxonômica, qualquer nó da árvore da vida. Qualquer táxon registrado pode ser usado em identificações de Indivíduos e Medições podem ser vinculadas a nomes taxonômicos.
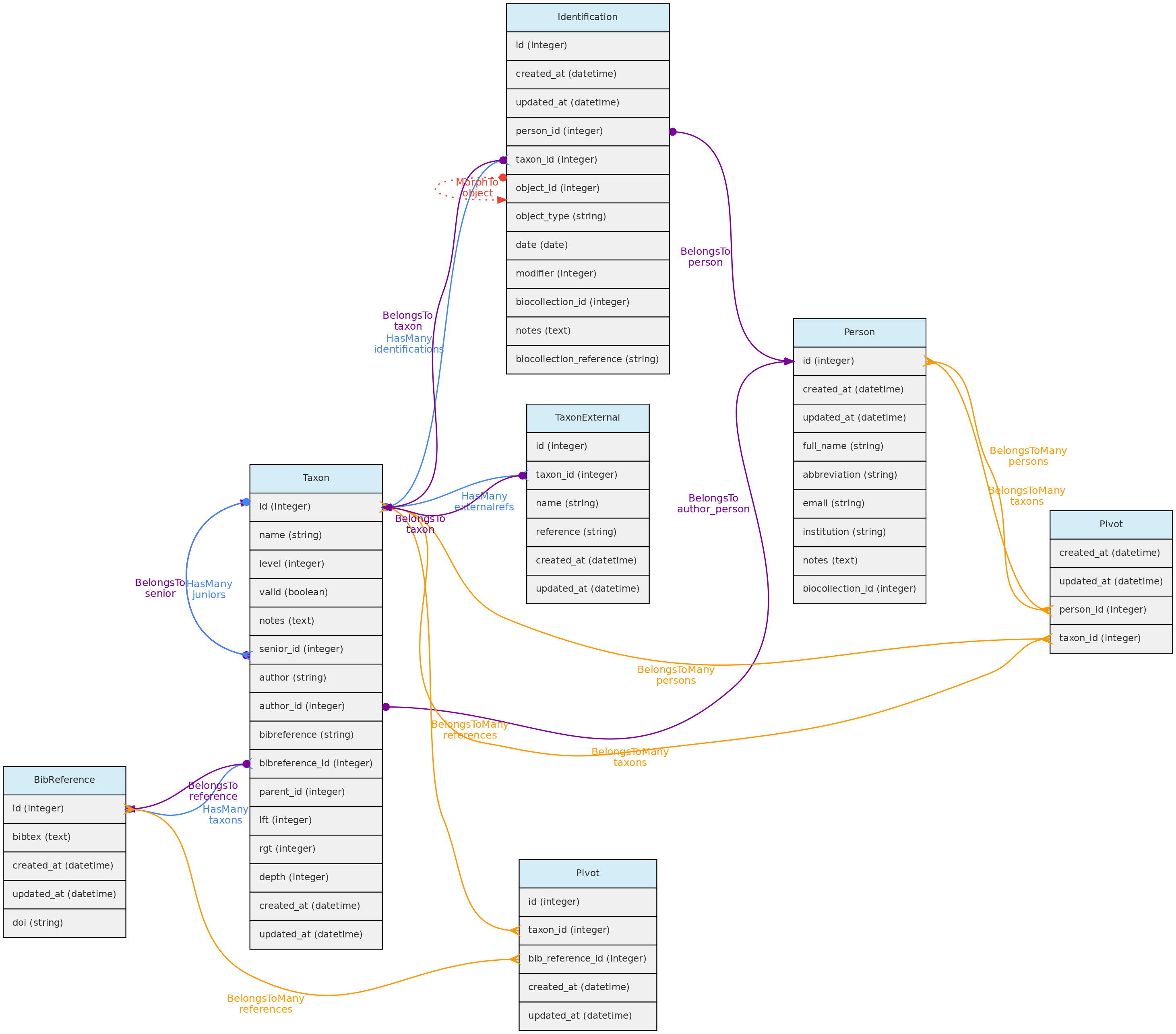 Modelo Taxon e suas relações. As linhas que ligam as tabelas indicam os
Modelo Taxon e suas relações. As linhas que ligam as tabelas indicam os métodos implementados nas classes mostradas, com cores indicando diferentes relacionamentos
Tabela Taxon
- Como, Localidades, o modelo Taxon tem um relacionamento pai-filho, implementado usando o modelo de conjunto aninhado para dados hierárquicos da biblioteca Laravel Baum que permite consultar ancestrais e descendentes. Consequentemente, as colunas
rgt, lft e deph da tabela de taxons são preenchidas automaticamente por esta biblioteca na inserção ou atualização dos dados.
- Para ambos, Taxon
author e Taxon bibreference, existem duas opções:
- Para nomes publicados, a autoria da string recuperada pelas APIs será colocada na coluna
author = string. Para nomes não publicados, o autor é uma pessoa e será armazenado na coluna author_id.
- Somente nomes publicados podem ter relação com BibReferences. O campo de string
bibreference da tabela Taxon armazena as strings recuperadas por meio de APIs externas, enquanto o bibreference_id se vincula a um objeto Referência Bibliográfica. Eles são usados para armazenar a publicação onde o nome do táxon é descrito e pode ser inserido em ambos os formatos.
- Além disso, um registro de táxon também pode ter muitas outras referências de Bib por meio de uma tabela dinâmica (
taxons_bibreference), permitindo vincular qualquer número de referências bibliográficas a um nome de táxon.
- A coluna
level representa a classificação taxonômica (como ordem, gênero, etc.). Ele é numericamente codificado e padronizado de acordo com as regras gerais do IAPT, mas deve acomodar também categorias de nível de táxon relacionadas a animais. Consulte os códigos disponíveis na API Taxon para obter a lista de códigos.
- A coluna
parent_id indica o pai do táxon, que pode estar vários níveis acima dele. O nível dos pais deve ser estritamente mais alto do que o nível do táxon, mas você não precisa seguir a hierarquia completa. É possível registrar um táxon sem os pais, por exemplo, um morfotipo não publicado para o qual o gênero e a família são desconhecidos pode ter uma ordem como pai.
- Os nomes das classificações taxonômicas são traduzidos de acordo com o
locale definido pelo sistema, que também traduz a interface da web (atualmente implementados apenas em português e inglês).
- O campo
nome da tabela de táxons contém apenas a parte específica do nome (no caso de espécies, o epíteto específico), mas a inserção e exibição dos táxons por meio da API ou interface web deve ser feito com a combinação fullname.
- É possível incluir sinônimos na tabela Taxon. Para isso, deve-se preencher o relacionamento
senior, que é o id do nome aceito (valid) para um táxon invalid. Se senior_id for preenchido, o táxon é um sinônimo junior e deve ser marcado como invalid.
- Ao inserir um novo táxon publicado, apenas o
nome é necessário. O nome será validado e o autor, referência e sinônimos serão recuperados usando os seguintes serviços de API:
- BackBone Taxonômico do GBIF - esta será a primeira verificação, da qual links para Tropicos e IPNI também podem ser recuperados se registrar um nome de planta.
- Tropicos - se não for encontrado no GBIF, o ODB pesquisará o nome no banco de dados de nomenclatura do Jardim Botânico do Missouri.
- IPNI - o Índice Internacional de Nomes Individuais é outro banco de dados usado para validar nomes individuais (temporariamente desativado)
- MycoBank - usado para validar um nome se não for encontrado pelos Tropicos nem IPNI apis, e usado para validar nomes para Fungi. Temporariamente desativado
- ZOOBANK - quando GBIF, Tropicos, IPNI e MycoBank não conseguem encontrar um nome, então o nome é testado contra a api ZOOBANK, que valida os nomes dos animais. Não fornece publicação de táxons, no entanto.
- Se um nome de táxon for encontrado nos bancos de dados Nomenclatural, o respectivo ID do repositório é armazenado nas tabelas
taxon_external, criando um link entre o registro do táxon OpenDataBio e o banco de dados nomenclatural externo.
- Pessoas pode ser definidas como especialistas em táxons por meio de uma tabela dinâmica. Assim, um objeto Taxon pode ter vários especialistas taxonômicos registrados no OpenDataBio.
Acesso a dados: usuários plenos podem registrar um novo táxon e editar os registros existentes se eles não tiverem sido usados para identificação. Atualmente é impossível remover um táxon do banco de dados. A lista de táxons tem acesso público.
Vouchers
O modelo Voucher é usado para armazenar registros de espécimes ou amostras de indivíduos depositados em BioColeções. Portanto, as únicas informações obrigatórias exigidas para registrar um Voucher são individual, biocollection e se o espécime é um tipo de nomenclatural (o padrão é non-type se não informado).
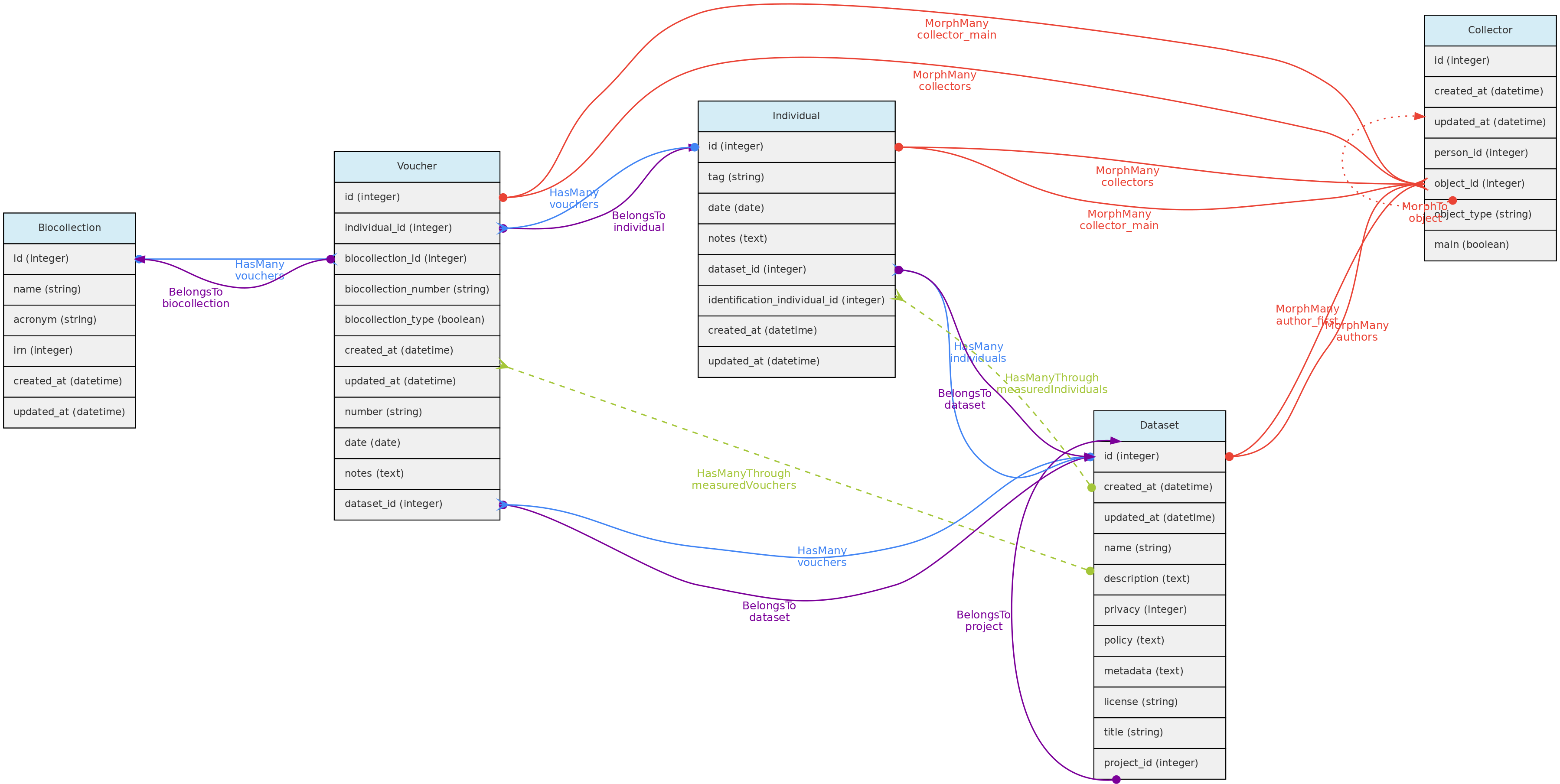 Modelo de voucher e suas relações. As linhas que ligam as tabelas indicam os
Modelo de voucher e suas relações. As linhas que ligam as tabelas indicam os métodos implementados nos modelos mostrados, com cores indicando diferentes relacionamentos. Note que a Identificação nem a Localidade são mostradas porque os Vouchers não têm seus próprios registros para esses dois modelos, eles são apenas herdados do Indivíduo ao qual o Voucher pertence
Vouchers table explained
- O Voucher pertence a um Indivíduo e a uma Biocoleção, portanto o
individual_id e o biocollection_id são obrigatórios nesta tabela;
biocollection_number é o código alfanumérico do Voucher na Biocoleção, pode ser ’nulo’ para usuários que desejam apenas indicar que um Indivíduo registrado tem Vouchers em uma Bicoleção específica, ou para Vouchers registrados para biocoleções que não tem um código identificador;biocollection_type - é um código numérico que especifica se o Voucher na BioCollection é um tipo nomenclatural. O padrão é 0 (não é um tipo); 1 apenas para ‘Tipo’, uma forma genérica e outros números para outros tipos nomenclaturiscollectors, um ou múltiplos, são opcionais para Vouchers, exigidos apenas se forem diferentes dos Coletores do Indivíduo. Caso contrário, os coletores do Indivíduo são herdados pelo Voucher. Como para indivíduos, eles são implementados por meio de uma relações polimórfica com a tabela de collectors e o primeiro coletor é o coletor_principal para o voucher, ou seja, aquele que se relaciona com number.number, este é o número do coletor, mas como coletores, só deve ser preenchido se for diferente do valor da tag do indivíduo. Portanto, collectors, number e date são úteis para registrar Vouchers para Indivíduos que têm Vouchers coletados em momentos diferentes por pessoas diferentes.- O campo
date nos modelos indivíduos e Voucher pode ser uma data incompleta. Exigido apenas se for diferente do indivíduo a quem o voucher pertence.
dataset_id o Voucher pertence a um Dataset, que controla a política de acesso;notes qualquer anotação de texto para o Voucher.- O modelo de Voucher interage com o modelo BibReference, permitindo vincular citações múltiplas a Vouchers. Isso é feito através da table
voucher_bibreference.
Acesso a dados Os vouchers pertencem a Conjuntos de Dados, portanto, a política de acesso a conjuntos de dados se aplica aos vouchers nele contidos. Os vouchers podem ter um conjunto de dados diferente de seus indivíduos. Se a política do conjunto de dados do Voucher for de acesso aberto e a Indivíduo não, o acesso aos dados do voucher será incompleto, portanto, o conjunto de dados do Voucher deve ter a mesma política de acesso ou uma política de acesso menos restrito do que o conjunto de dados do Indivíduo. Apenas os colaboradores e administradores do conjunto de dados podem inserir ou editar vouchers em um conjunto de dados, mesmo se o conjunto de dados for de acesso público.
2 - Objetos de Atributos
Objetos para variáveis definidas pelo usuário e suas medições
Medições
A tabela measurements armazena os valores para variáveis medidas para os objetos centrais. Seu relacionamento com os objetos centrais é definido por uma relações polimórficas usando as colunas measured_id e measured_type. Essas relações MorphTo são ilustradas e explicadas na página objetos centrais.
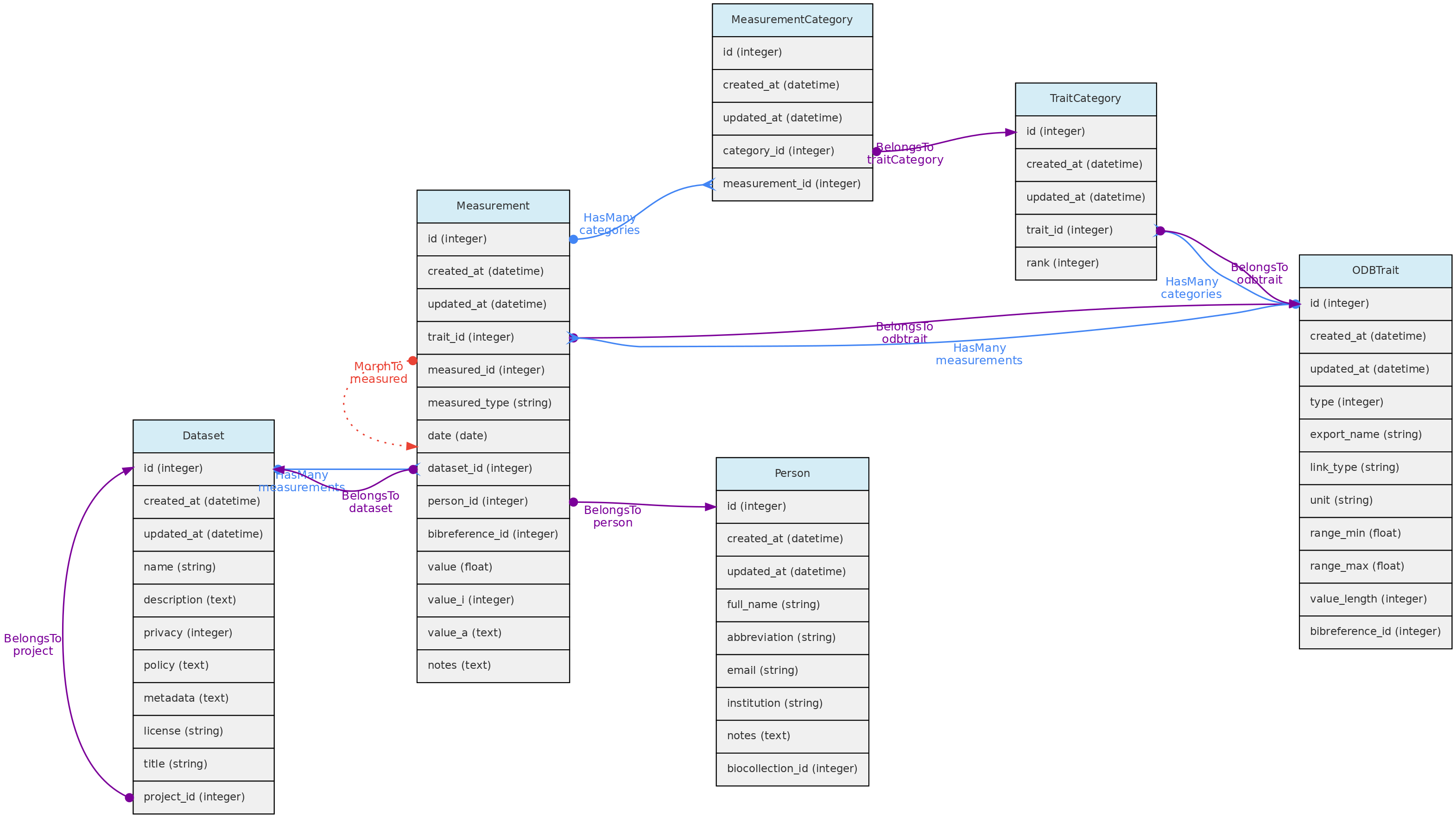
- medições devem pertencer a um Conjuntos de dados - coluna
dataset_id, que controla a política de acesso
- Uma Pessoa deve ser indicada como o medidor (
person_id);
- A coluna
bibreference_id pode ser usada para vincular medições extraídas de publicações à sua fonte fonte Bibliográfica
- O valor para a variável medida (
trait_id) será armazenado em colunas diferentes, dependendo do tipo de variável:
value - esta coluna flutuante armazenará valores para características reais quantitativas;value_i - esta coluna inteira armazenará valores para características Quantitative Integer; e é um campo opcional para características do tipo Link, permitindo, por exemplo, armazenar contagens para uma espécie (uma característica Link Taxon) em um local.value_a - esta coluna de texto armazenará valores para os tipos de traço Texto, Cor e Espectral.
- Valores para variáveis categóricas e ordinais são armazenados na tabela
measurement_category
data - a data de medição é obrigatória em todos os casos
Acesso a dados As medições pertencem a Conjuntos de dados, portanto, a política de acesso a conjuntos de dados se aplica às medições nele. Apenas os colaboradores e administradores do conjunto de dados podem inserir ou editar medições em um conjunto de dados, mesmo se o conjunto de dados for de acesso público.
Variáveis
A tabela traits representa as variáveis definidas pelo usuário para coletar Mediçõespara um dos objetos centrais.
Essas características personalizadas fornecem enorme flexibilidade aos usuários para registrar suas variáveis de interesse. Obviamente, tal flexibilidade tem um custo na padronização dos dados, pois uma mesma variável pode ser registrada de forma diferentes em qualquer instalação do OpenDataBio. Para minimizar a redundância na ontologia de características, os usuários que criam características são avisados sobre esse problema e uma lista de características semelhantes é apresentada no caso de ser encontrada por comparação de nomes de características.
As variáveis têm restrições de edição para evitar perda de dados ou alteração não intencional do significado dos dados. Portanto, embora a lista de variáveis esteja disponível para todos os usuários, as definições de variáveis não podem ser alteradas se outra pessoa também usou a variável para armazenar medições.
Variáveis são entidades traduzíveis, então seus valores de name e description podem ser armazenados em vários idiomas (veja traduções de usuário. Isso é colocado na tabela user_translations através de uma relação polimórfica.
A definição da variável deve ser tão específica quanto necessário. A medição da altura das árvores usando medição direta ou um clinômetro, por exemplo, pode não ser facilmente convertida de uma para outra e deve ser armazenada em variáveis diferentes. Portanto, é altamente recomendável que o campo de definição de variável inclua informações como instrumento de medição e outros metadados que permitam que outros usuários entendam se podem usar sua variável ou criar uma nova.
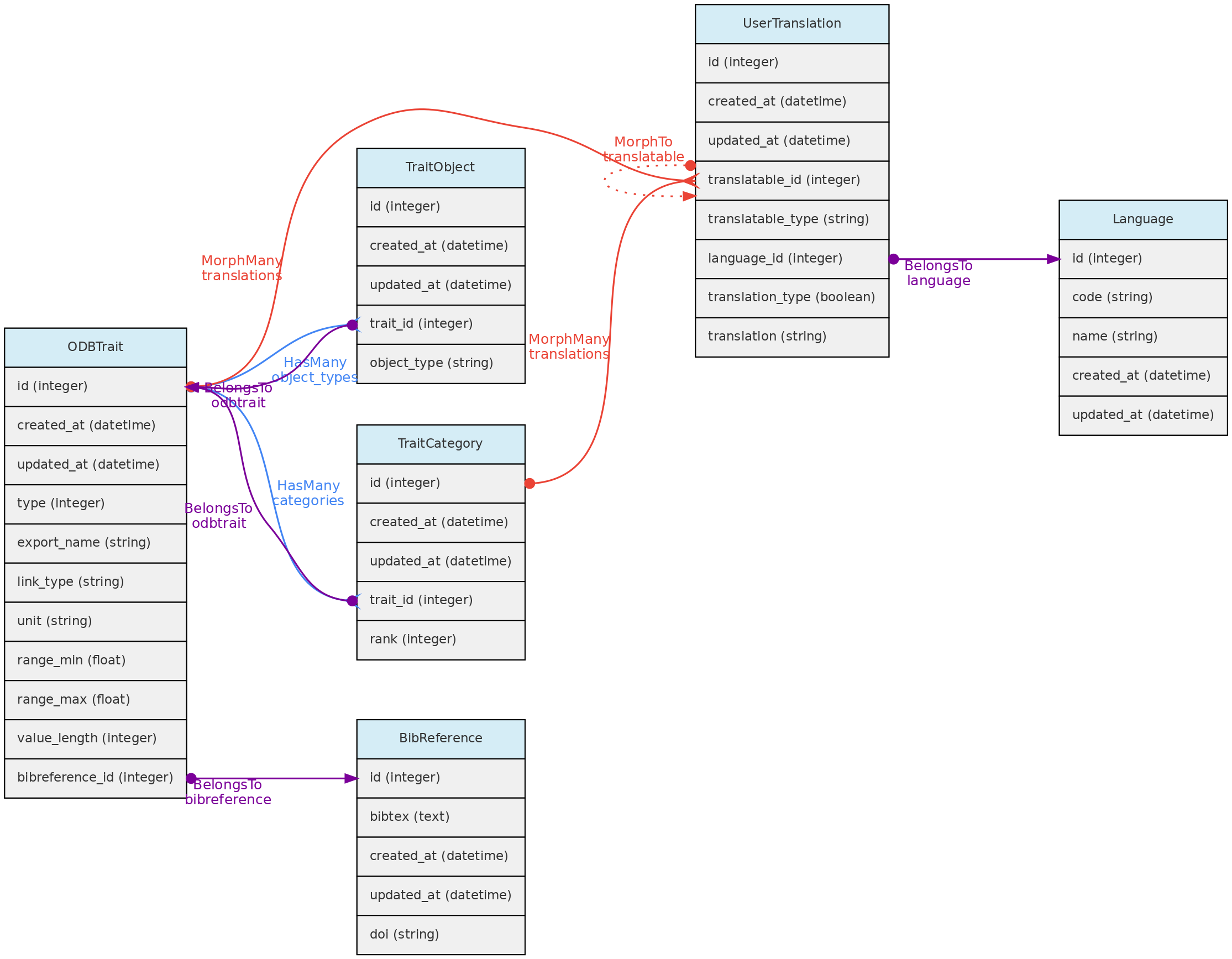
- A definição da variável deve incluir um
export_name, que será usado durante as exportações de dados nos formulários de entrada da interface. Os nomes de exportação devem ser únicos e não devem ter tradução. São recomendados nomes de exportação curtos e [camelCase](https://en.wikipedia.org/wiki/Camel_case) ou [PascalCase](https://en.wikipedia.org/wiki/pascal_case).
- Os seguintes tipos de características estão disponíveis:
- Quantitativo real - para números reais;
- Número inteiro quantitativo - para contagens;
- Categórico - para varáveis categóricas de seleção única;
- Múltiplo categórico - para muitas categorias selecionáveis;
- Ordinal categórico - para uma categoria ordenada selecionável (dados semiquantitativos);
- Texto - para qualquer valor de texto;
- Cor - para qualquer valor de cor, especificado pelo código de cor hexadecimal (paleta de cores)
- Link - este é um tipo de variável especial no OpenDataBio. Apenas links para Taxons e Vouchers estão implementados. Exemplo de uso: se você deseja armazenar contagens de espécies conduzidas em uma Localidade, você pode criar uma variável do tipo de link_taxon ou link_voucher. A medição para tal variável terá um campo opcional
value para armazenar as contagens. Este tipo de característica também pode ser usado para especificar o hospedeiro de um parasita ou o número de insetos predadores.
- Espectral - projetado para acomodar dados espectrais, compostos de vários valores de absorbância ou refletância para diferentes números de onda.
- GenBank - armazena os números de acesso do GenBank que permitem recuperar dados moleculares vinculados a indivíduos ou vouchers armazenados no banco de dados através do GenBank Serviço API.
- A tabela Traits contém campos que permitem a validação do valor da medição, dependendo do tipo de variável:
range_max e range_min - se definido para variáveis quantitativas, as medições terão que se ajustar ao intervalo especificado;value_length - obrigatório apenas para variáveis espectrais, valida o comprimento (número de valores) de uma medição espectral;link_type - se a variável for do tipo Link, a medição em value_i deve ser um id do objeto do tipo de link;- Para variáveis do tipo cor, os valores são validados no processo de criação da medição e devem estar em conformidade com um código hexadecimal de cores. Um seletor de cores é apresentado na interface web para inserção e edição de medições de cores;
- Variáveis categóricas e ordinais serão validadas para as categorias registradas ao importar medições por meio da API;
- A coluna
unit define a unidade de medida para a variável.
- A coluna
bibreference_id é a chave de uma única Referência Bibliográfica que pode ser vinculad à definição da variável.
- A tabela
trait_objects armazena o tipo de objetos centrais para o qual a variável pode ter uma medição;
Acesso aos dados O nome, definição, unidade e categorias de uma variável não podem ser atualizados ou removidos se houver alguma medição registrada no banco de dados. As únicas exceções são: (a) é permitido adicionar novas categorias para variáveis categóricas (não ordinais); (b) o usuário atualizando a variavel é a única pessoa que possui medições para a característica; (c) o usuário que atualiza a variável é um administrador de todos os conjuntos de dados com medições usando a variável.
Um Formulário é um grupo organizado de variáveis definido por um usuário para criar um formulário personalizado para inserir medições através da interface web. Um Formulário consiste em um grupo de variáveis ordenadas, que podem ser marcadas como “obrigatórias”. As entidades relacionadas são o Relatório e o Filtro.
Isso ainda é experimental e em desenvolvimento
3 - Objetos de Acesso
Objetos que controlam o acesso e distribuição de dados!
Os objetos centrais são: Localidades, Vouchers, Individual e Taxons. Essas entidades são consideradas “centrais” porque podem receber Medições, ou seja, você pode registrar valores para qualquer Variável.
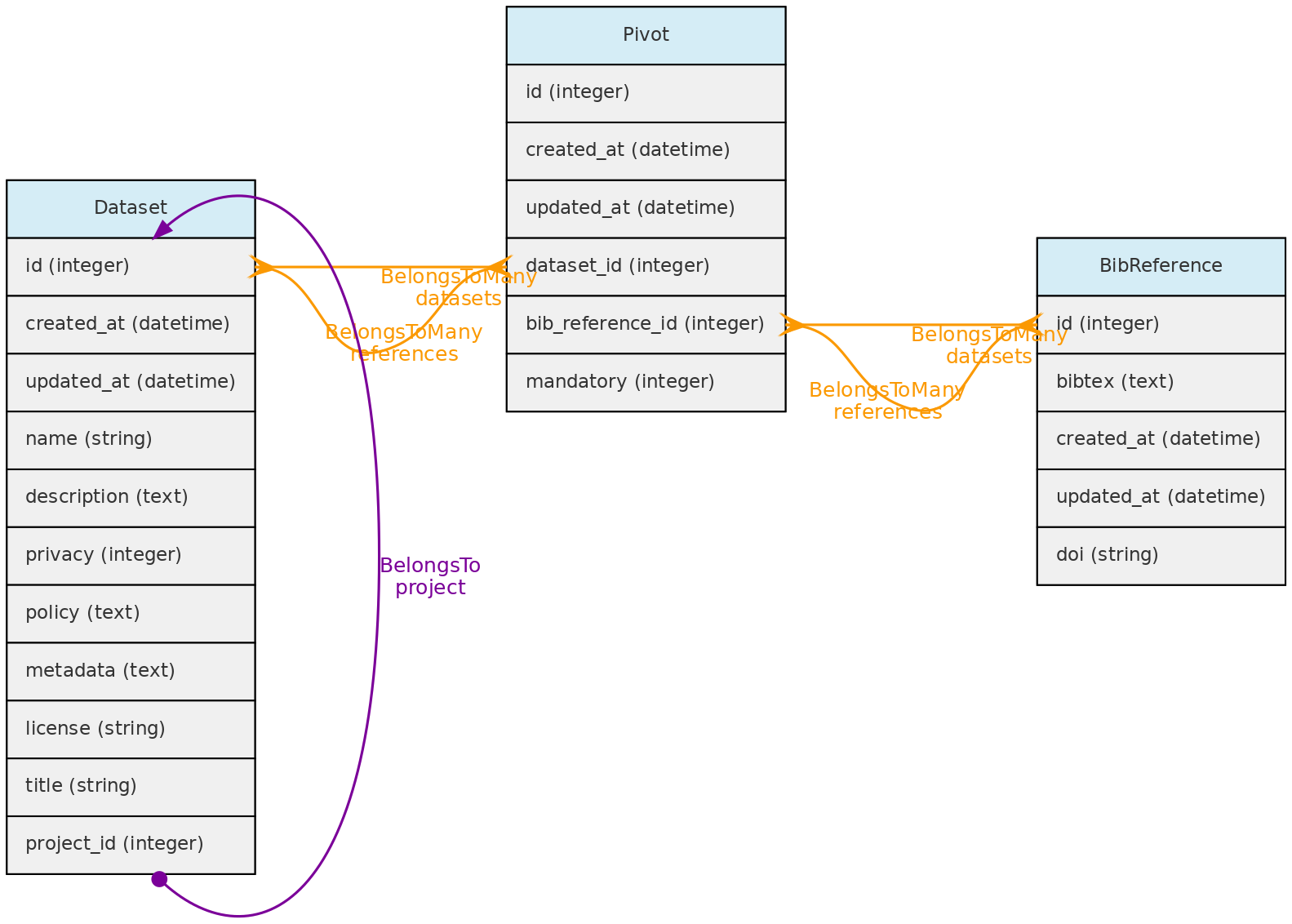
Conjuntos de dados controlam acesso aos dados e representam um publicação de dados dinâmica, cuja versão é definida pela data da última edição do registros incluídos. Conjuntos de dados contém Medições, Indivíduos, Vouchers e Arquivos de Mídia.
Projetos são apenas grupos de Conjuntos de dados e de Usuários, representando grupos de usuários com acessibilidade comum a conjuntos de dados cuja privacidade é definida para ser controlada por um projeto.
BioColeções - este modelo serve para criar uma lista reutilizável de acrônimos de Coleções Biológicas no registro de Vouchers. Mas tem opcionalmente a possibilidade de gerir uma coleção de registros de Vouchers e seus Indivíduos, paralelamente ao controle provido por Conjuntos de dados. O controle é apenas na edição e na entrada de dados. Neste caso a BioColeção é administrada pelo sistema.
Projetos e BioColeções devem ter pelo menos um Usuário definido como administrador, que tem controle total sobre o conjunto de dados ou projeto, incluindo a atribuição das seguintes funções a outros usuários: administrador, colaborador ouvisualizador:
- Colaboradores podem inserir e editar objetos, mas não podem excluir registros, nem alterar o conjunto de dados ou a configuração do projeto.
- Visualizadores têm acesso somente de leitura aos dados que não são de acesso aberto.
- Apenas usuários plenos e super-admins podem ser designados como administradores ou colaboradores. Assim, se um usuário que era administrador ou colaborador de um conjunto de dados for rebaixado a “Usuário registrado”, ele se tornará um visualizador.
- Super-admins apenas podem habilitar uma BioColeção para ser administrada pelo sistema.
BioColeções
O modelo Biocollection tem duas funções: (1) prover uma lista de acrônimos para registrar Vouchers de qualquer Coleção Biológica ; (2) gerir os dados de uma Coleção Biológica, facilitando o registro de novos dados (qualquer usuário entra seus dados usando as validações realizadas pelo software e solicita pela interface aos curadores da Coleção o registro dos dados que é feito pelos usuários autorizados na BioColeção, que a partir da transferência passa a controlar a edição dos dados dos Indivíduos. A opção (2) precisa ser implementada por um usuário SuperAdministrador do sistema, que ao habilitar a BioColeção para ser administrada pelo sistema, implementa o modelo de solicitações ODBRequest para que usuários possam pedir dados, amostras, ou registros e mudanças nos dados.
O objeto Biocollection pode ser uma Coleção Biológica formal, como as registradas no Index Herbariorum, ou qualquer outra Coleção Biológica.
O objeto Biocollection também interage com o modelo Person. Quando uma Pessoa está vinculada a uma Biocoleção, ela será listada como especialista taxonômico e pode ter também um vínculo com Taxons.
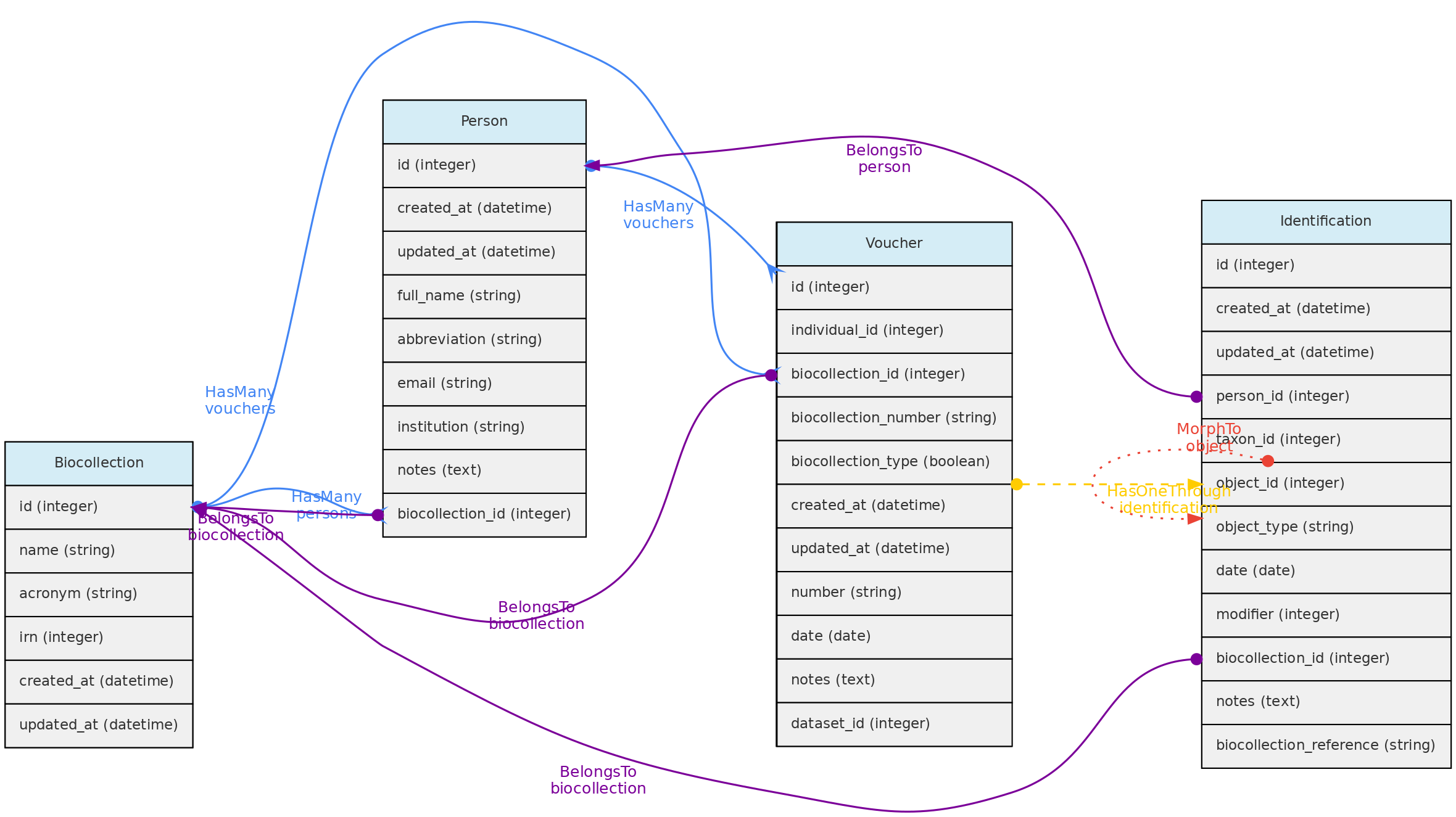
Acesso a dados - Usuários plenos podem registrar BioColeções, mas apenas usuários super-administradores podem tornar uma BioColeção adminstrável pelo sistema. Remover BioColeções pode ser feito se não há Vouchers ligados e se não é administrada pelo sistema. Se administrável, o modelo interage com Usuários, que podem ser administradores (curadores, pode tudo) ou colaboradores (podem entrar e editar dados, mas não podem apagar registros). Dados de outros Conjuntos de Dados podem fazer parte da BioColeção, permitindo que usuários tenham seus dados completo, mas o controle da edição dos registros é da BioColeção.
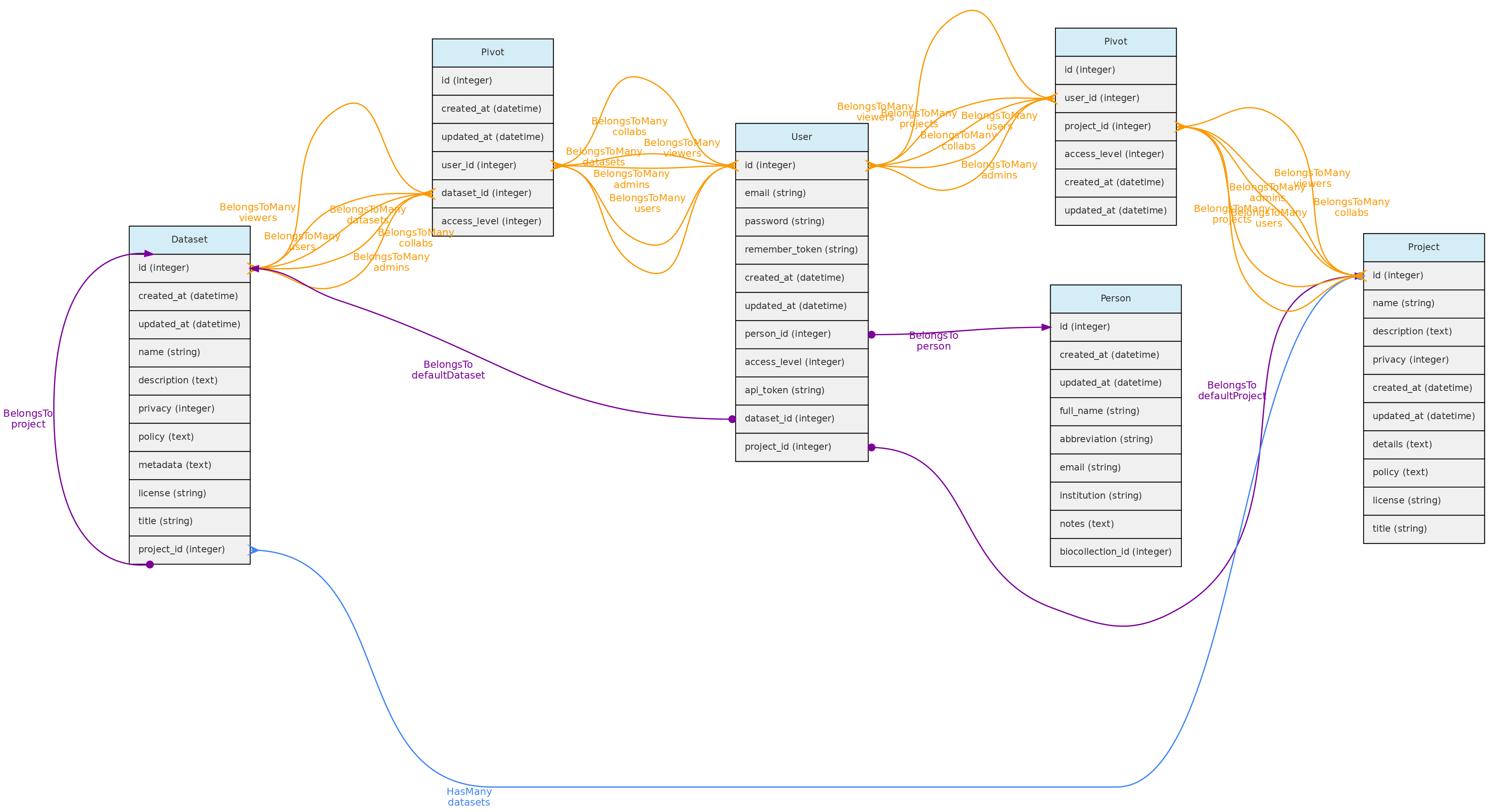
Conjuntos de Dados
Conjuntos de Dados são grupos de Medições, Indivíduos, Vouchers Arquivos de Mídia, ae podem ter um ou mais Usuários administrators, collaborators or viewers. Administradores podem definir o nivel de acesso para acesso público, restrito à usuários cadastrados or restrito à usuários autorizados or restrito à usuários do projeto. Este controle de acesso aos dados dentro de um conjunto de dados, conforme exemplificado no diagrama abaixo:
Conjuntos de dados podem ter muitas Referências bibliográficas, que junto com os campos policy, metadata permitem anotar o conjunto de dados com informações relevantes para o compartilhamento de dados:
* Vincule qualquer publicação que tenha usado o conjunto de dados e, opcionalmente, indique se são de citação obrigatória ao usar os dados;
* Defina uma política de dados específica ao usar os dados além de uma licença pública [CreativeCommons.org]((https://creativecommons.org/licenses/)
* Detalhe quaisquer metadados relevantes, além daqueles que são automaticamente recuperados do banco de dados, como as definições dos Variáveis medidas.
Projetos
Projetos são apenas grupos de Conjuntos de dados e interagem com Usuários, tendo administradores,colaboradores ou visualizadores. Esses usuários podem controlar todos os conjuntos de dados dentro do Projeto que tenham como política de acesso restrita aos usuários do projeto.
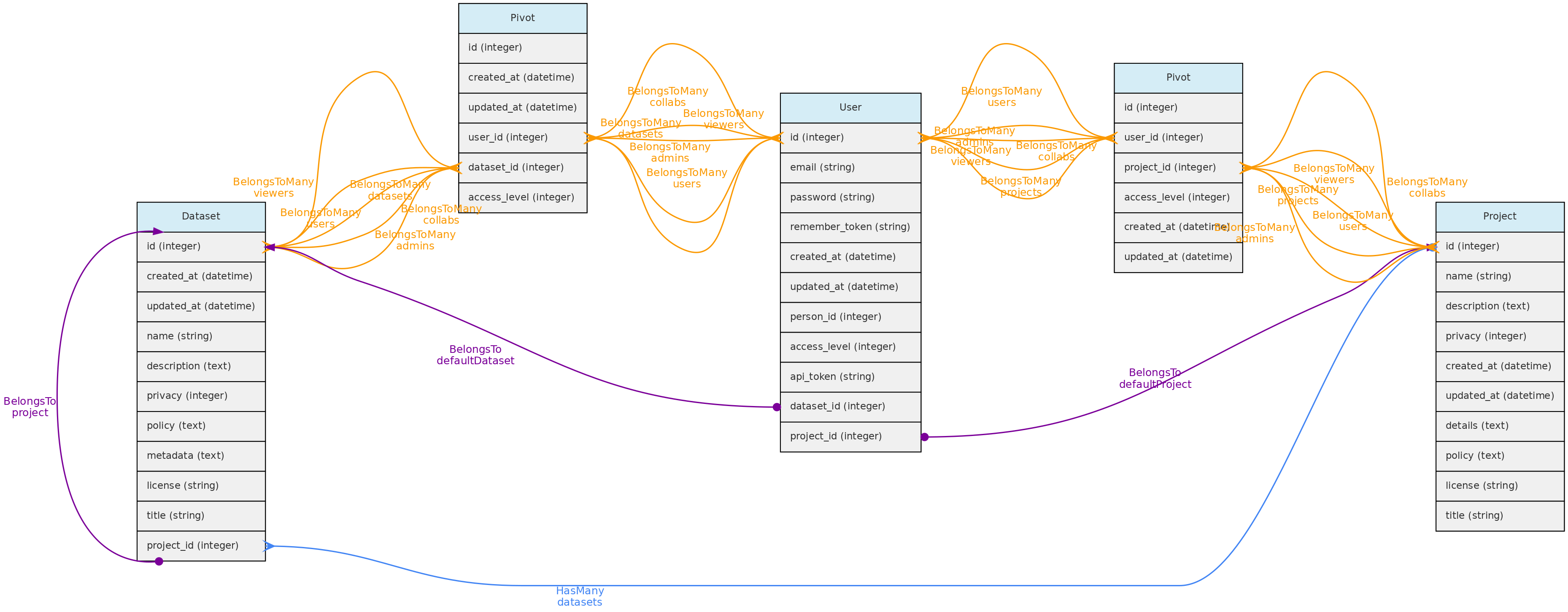
Usuários
O tabela users armazena informações sobre os usuários e administradores do banco de dados. Cada Usuário pode ser associado a uma Pessoa. Quando esse usuário insere novos dados, essa pessoa é usada como a pessoa padrão nos formulários. A pessoa só pode estar associada a um único usuário.
Existem três níveis de acesso possíveis para um usuário:
* Usuário registrado (o nível mais baixo) - tem muito poucas permissões
* Usuário pleno ou completo - podem ser atribuídos como administradores ou colaboradores de Projetos e Conjuntos de Dados;
* SuperAdmin (o nível mais alto). - os superadministradores têm acesso a todos os objetos, independentemente da configuração do conjunto de dados. Categoria para os administradores da instalação.
Cada usuário é atribuído ao nível usuário registrado quando ele ou ela se registra em um sistema OpenDataBio. Depois disso, alguém que seja SuperAdmin pode promovê-lo a Usuário Pleno ou SuperAdmin. Os SuperAdmins também podem editar outros usuários e removê-los do banco de dados.
Para cada usuário pleno é criado um projeto e um conjunto de dados de uso pessoal. Esses workspaces permitem que os usuários importem dados antes de incorporá-los a um projeto maior ou mais definitivo.
Acesso a dados: os usuários são criados no momento do registro. Apenas os administradores podem atualizar e excluir registros do usuário.
Jobs do usuário
A tabela user_jobs é usada para armazenar temporariamente tarefas que são executadas em segundo plano, como a importação e exportação de dados. Qualquer usuário tem permissão para criar um Job; cancelar seus próprios Jobs; listar os Jobs que não foram excluídos.
A tabela jobs contém os dados usados pelo framework Laravel para interagir com a Queue. Os dados desta tabela são excluídos quando o trabalho é executado com êxito. A tabela user_jobs é usada para manter essas informações, além de permitir logs do processo, repetir tarefas com erros e cancelar tarefas que ainda não foram concluídas.
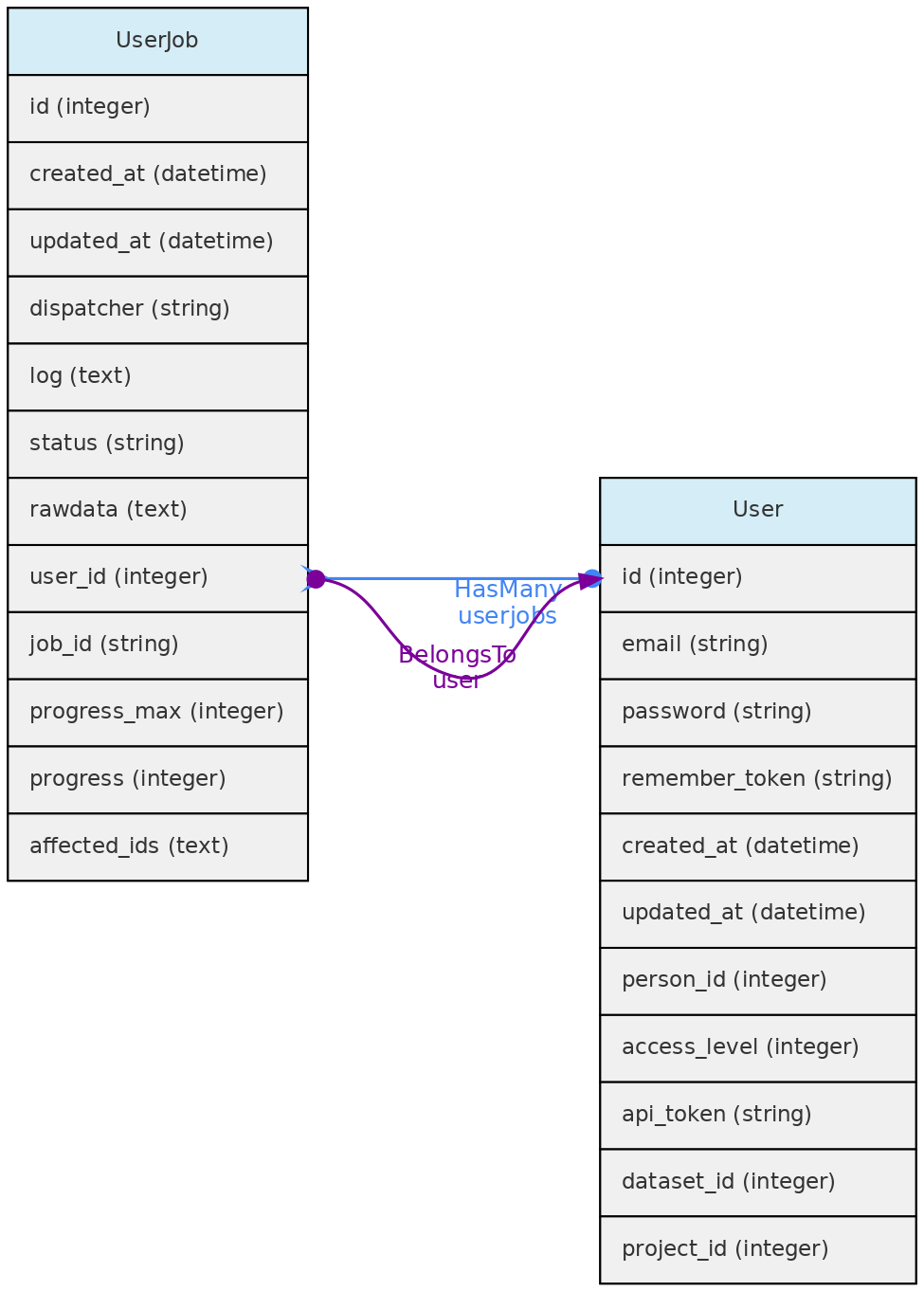
Acesso a dados: Cada usuário registrado pode ver, editar e remover seus próprios Jobs.
4 - Objetos Auxiliares
Bibliotecas de uso comum, como Pessoas, Referências Bibliográficas, BioColeção e Traduções de Usuário!
Referências Bibliográficas
A tabela bib_references contém basicamente referências formatadas em BibTex armazenadas na coluna bibtex. Você pode facilmente importar referências para o OpenDataBio através da interface, apenas especificando o doi ou simplesmente enviando um registro bibtex ou um arquivo .bib. Essas referências bibliográficas podem ser usadas para:
- Conjuntos de dados - com a opção de definir referências para as quais a citação é obrigatória quando o Conjunto de Dados for usado em publicações; mas todas as referências que usaram o conjunto de dados podem ser vinculadas ao conjunto de dados; os links são feitos com uma tabela dinâmica chamada
dataset_bibreference;
- Taxons:
- para especificar a referência na qual o nome do táxon foi descrito, atualmente obrigatório em algumas revistas taxonômicas como PhytoTaxa. Esta referência de descrição é armazenada no
bibreference_id da tabela Taxons.
- para registrar qualquer referência a um nome de táxon, que são então vinculados por meio de uma tabela dinâmica chamada
taxons_bibreference.
- Vincule uma Medição a uma fonte publicada;
- Indique a origem de uma definição de Variável.
- Indique citações obrigatórias para um conjunto de dados ou vincule referências usando os dados para um conjunto de dados
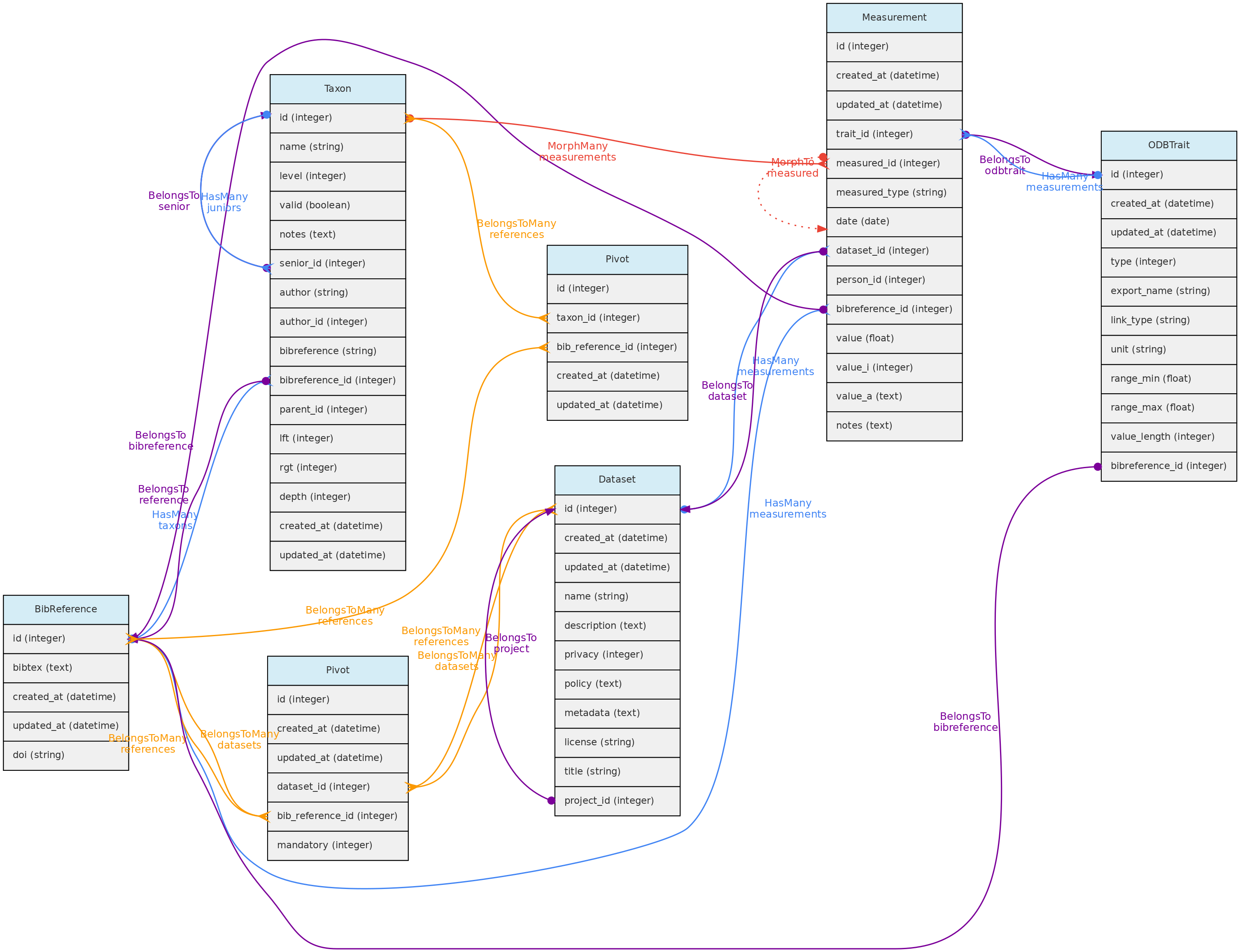 O modelo BibReference e seus relacionamentos. As linhas que ligam as tabelas indicam os
O modelo BibReference e seus relacionamentos. As linhas que ligam as tabelas indicam os métodos implementados, com cores indicando diferentes relacionamentos
Tabela Bibreferences
- A
bibtexKey, autores e outros campos relevantes são extraídos do registro em formato bibtex armazenado na coluna bibtex.
- A ** bibtexkey deve ser única** no banco de dados e uma função auxiliar é fornecida para padronizá-la com o formato
<von? sobrenome> <ano> <primeira palavra do título>. A “parte von” do nome é “von”, “di”, “de la”, que fazem parte do sobrenome de alguns autores. A primeira palavra do título ignora palavras de interrupção comuns, como “a”, “o” ou “em”.
- DOI para uma referencia pode ser especificado no campo BibTex relevante ou em uma entrada de texto separada, e são armazenados no campo
doi quando presente. Uma API externa encontra o registro bibliográfico quando um usuário informa o doi na interface.
**Acesso a dados** [usuários completos](/docs/concepts/data-access/#user) podem registrar novas referências, editar detalhes de referências e remover registros de referência que não têm dados associados. BibReferences tem acesso público!
Identificação Taxonômica
O modelo Identificação representa a identificação taxonômica de Indivíduos.
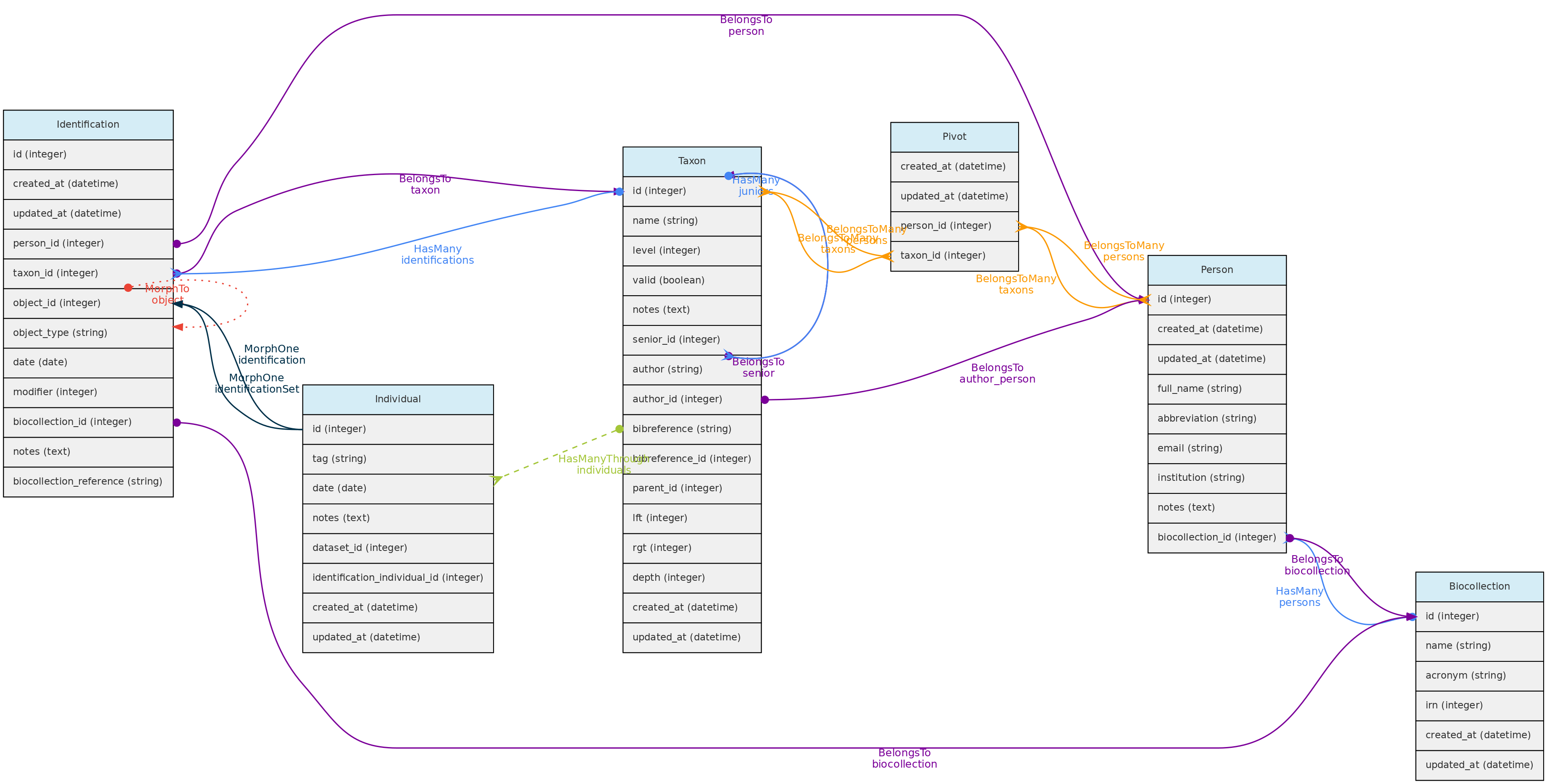 Modelo de identificação e suas relações. Linhas ligando tabelas indicam os
Modelo de identificação e suas relações. Linhas ligando tabelas indicam os métodos implementados, com cores indicando diferentes relacionamentos do Laravel Eloquent
Tabela identifications
- O modelo de Identificação inclui vários campos opcionais, mas além de
taxon_id, person_id, a Pessoaresponsável pela identificação e a date de identificação são obrigatórios.
- O valor de
date pode ser uma Data Incompleta, por exemplo apenas o ano ou ano + mês podem ser registrados.
- Os seguintes campos são opcionais:
modifier - é um código numérico que anexa um modificador taxonômico ao nome. Valores possíveis ’s.s.’ = 1, ’s.l.’ = 2, ‘cf.’ = 3, ‘aff.’ = 4, ‘vel aff.’ = 5, o padrão é 0 (nenhum).notes - um texto de escolha, útil para adicionar comentários à identificação.biocollection_id e biocollection_reference - esses campos devem ser usados para indicar que a identificação é baseada na comparação com um voucher depositado em uma coleção biológica e cria um link entre o indivíduo identificado e o espécime da BioColeção no qual a identificação foi baseada. biocollection_id armazena o id de BioColeção e biocollection_reference o identificador único do espécime comparado, ou seja, seria o equivalente ao biocollection_number do modelo Voucher, mas esta referência não precisa ser de um voucher registrado no banco de dados.
- O relacionamento com o modelo individual é definido por relações polimórficas usando os campos
object_type e object_id. [Na estrutura atual, isso pode ser substituído por um ‘individual_id’ na tabela de identificação. A relação polimórfica foi herdada de uma versão anterior de desenvolvimento e foi mantida porque o modelo de Identificação poderá ser necessário para vincular outros objetos].
- Mudanças na Identificação de Indivíduos são auditadas para rastrear o histórico de mudanças
Acesso aos dados: as identificações são atributos dos Indivíduos e não possuem acesso independente!
Pessoas
O modelo Persons armazena nomes de pessoas que podem ou não ser um Usuário diretamente envolvido com a base de dados. Pessoas podem ser:
* coletores de Vouchers, Indivíduos e Arquivos de Mídia
* identificadores taxonômicos de Indivíduos;
* medidores de Medições;
* autores para nomes não publicados de Taxons;
* especialistas taxonômicos - ligados ao modelo Taxon pela tabela person_taxon;
* autores de Conjuntos de Dados
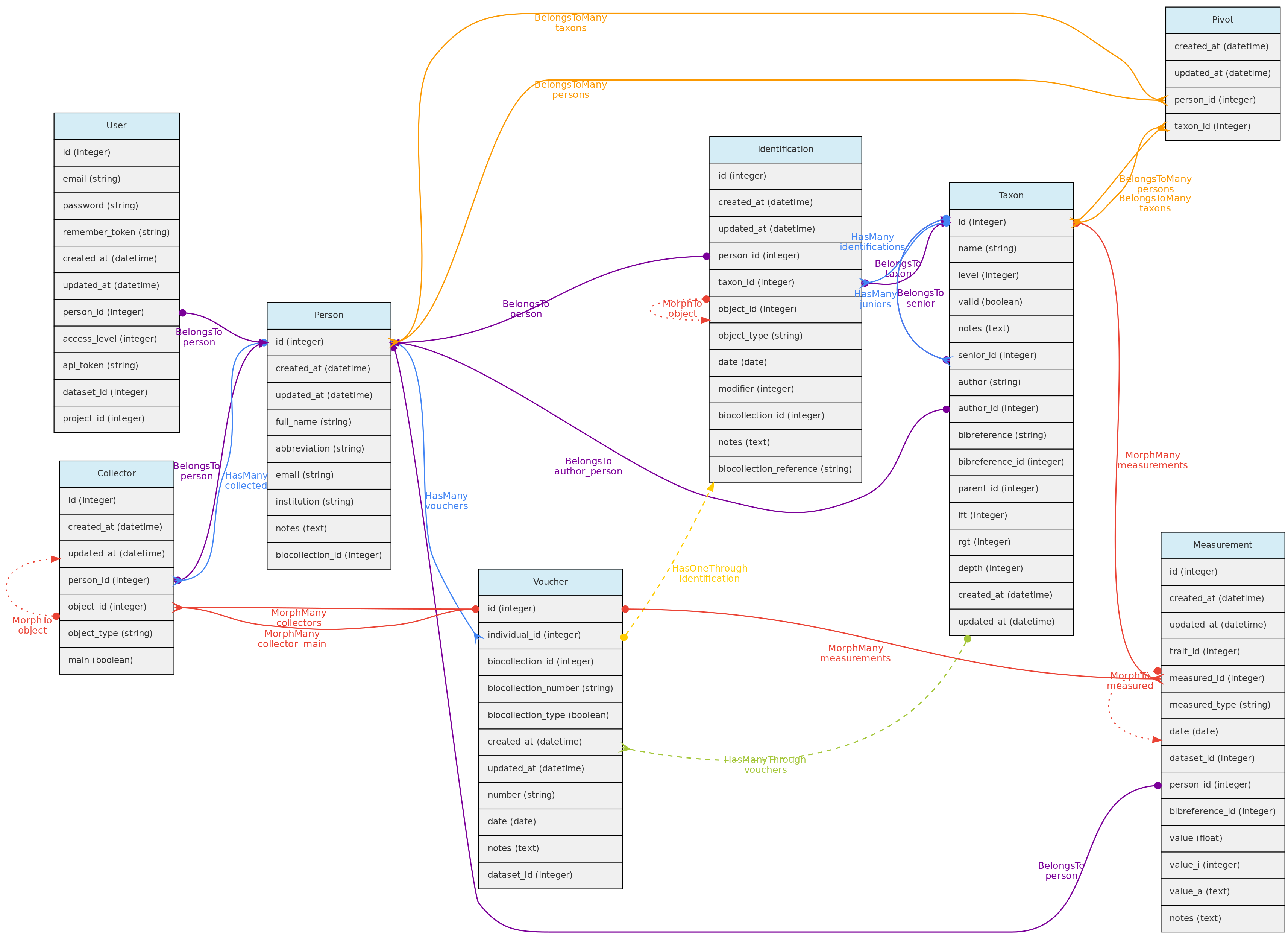 Modelo Persons e seus relacionamentos. As linhas que ligam as tabelas indicam os
Modelo Persons e seus relacionamentos. As linhas que ligam as tabelas indicam os métodos implementados, com cores indicando diferentes tipos de métodos do Laravel Eloquent, linhas sólidas as relações diretas e tracejadas as indiretas
Tabela persons
- as colunas obrigatórias são a pessoa
full_name e abbreviation;
- ao cadastrar uma nova pessoa, o sistema sugere o nome
abbreviation, mas o usuário é livre para alterá-lo para melhor adaptá-lo à abreviatura usual de cada pessoa. A ** abreviatura deve ser única ** no banco de dados, duplicatas não são permitidas na tabela Pessoas. Portanto, duas pessoas com exatamente o mesmo nome devem ser diferenciadas de alguma forma na coluna abbreviation.
- A coluna
biocollection_id da tabela Pessoas é usada para listar a qual BioColeção uma pessoa está associada, que pode ser usada quando a Pessoa também é um especialista taxonômico.
- Adicionalmente, também podem ser informados o
e-mail e a institution a que pertence a pessoa.
- Cada usuário pode ser vinculado a uma pessoa pelo
person_id na tabela Usuário. Essa pessoa é então usada como a pessoa ‘padrão’ quando o usuário está logado no sistema.
**Acesso a dados** [usuários completos](/docs/concepts/data-access/#user) podem registrar novas pessoas e editar as pessoas inseridas e remover pessoas que não possuem dados associados. Os administradores podem editar qualquer pessoa. A lista de pessoas tem acesso público.
Arquivos de Mídia
Arquivos de mídia são semelhantes a Medições no sentido de que podem estar associados a qualquer objeto central. Os arquivos de mídia podem ser imagens (jpeg, png, gif, tif), arquivos de vídeo ou áudio e podem ser disponibilizados gratuitamente ou colocados em um Conjuntos de dados com uma política de acesso definida. Uma licença CreativeCommons.org pode ser atribuída a eles. Os arquivos de mídia podem ser anotados, ou seja, você pode definir palavras-chave para eles, permitindo consultá-los por Tags. Por exemplo, uma imagem individual pode ser marcada com ‘flores’ ou ‘frutos’ para indicar o que está na imagem, ou uma tag que informa sobre a qualidade da imagem.
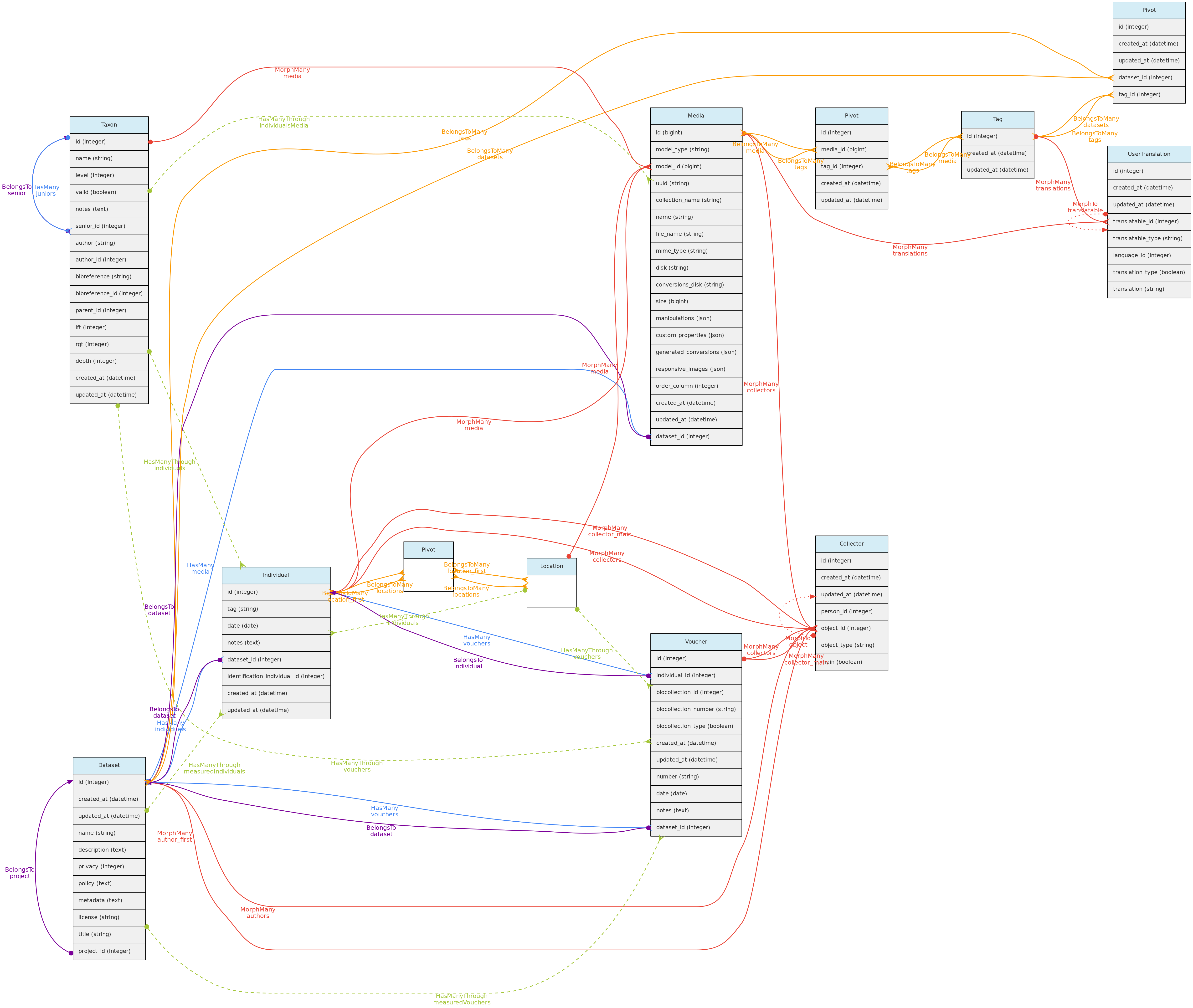
- Os arquivos de mídia (imagem, vídeo, áudio) são vinculados aos objetos centrais por meio de uma relações polimórfica definida pelas colunas
model_id e model_type.
- Múltiplas Pessoas podem ser associadas à Mídia para créditos, eles estão vinculados à tabela collectors, também através de relações polimóficas.
- Uma Mídia pode ter uma
description em cada idioma configurado na tabela languages, que será armazenada na tabela user_translations. As entradas para cada idioma são mostradas nos formulários da interface.
- Os arquivos de mídia não são armazenados no banco de dados, mas na pasta de armazenamento do servidor.
- É possível fazer upload em lote de arquivos de mídia por meio da interface web, exigindo também um arquivo informando os objetos aos quais vincular a mídia e os metadados.
Acesso a dados usuários completos podem registrar arquivos de mídia e excluir aqueles que eles inseriram. Se a mídia estiver em um conjunto de dados, os administradores do conjunto de dados podem excluir a mídia além do usuário. Os arquivos de mídia têm acesso público, exceto quando vinculados a um conjunto de dados com restrições de acesso.
Tag Model
O modelo Tag permite que os usuários definam palavras-chave traduzíveis que podem ser usadas para sinalizar Conjuntos de dados, Projetos ou Arquivos de Mídia. O modelo Tag está vinculado a esses objetos por meio de uma tabela para cada um, denominada dataset_tag, project_tag e media_tag, respectivamente.
Um Tag pode ter name e description em cada idioma configurado na tabela de Idiomas, que serão armazenados na tabela user_translations. As entradas para cada idioma são mostradas nos formulários da interface.
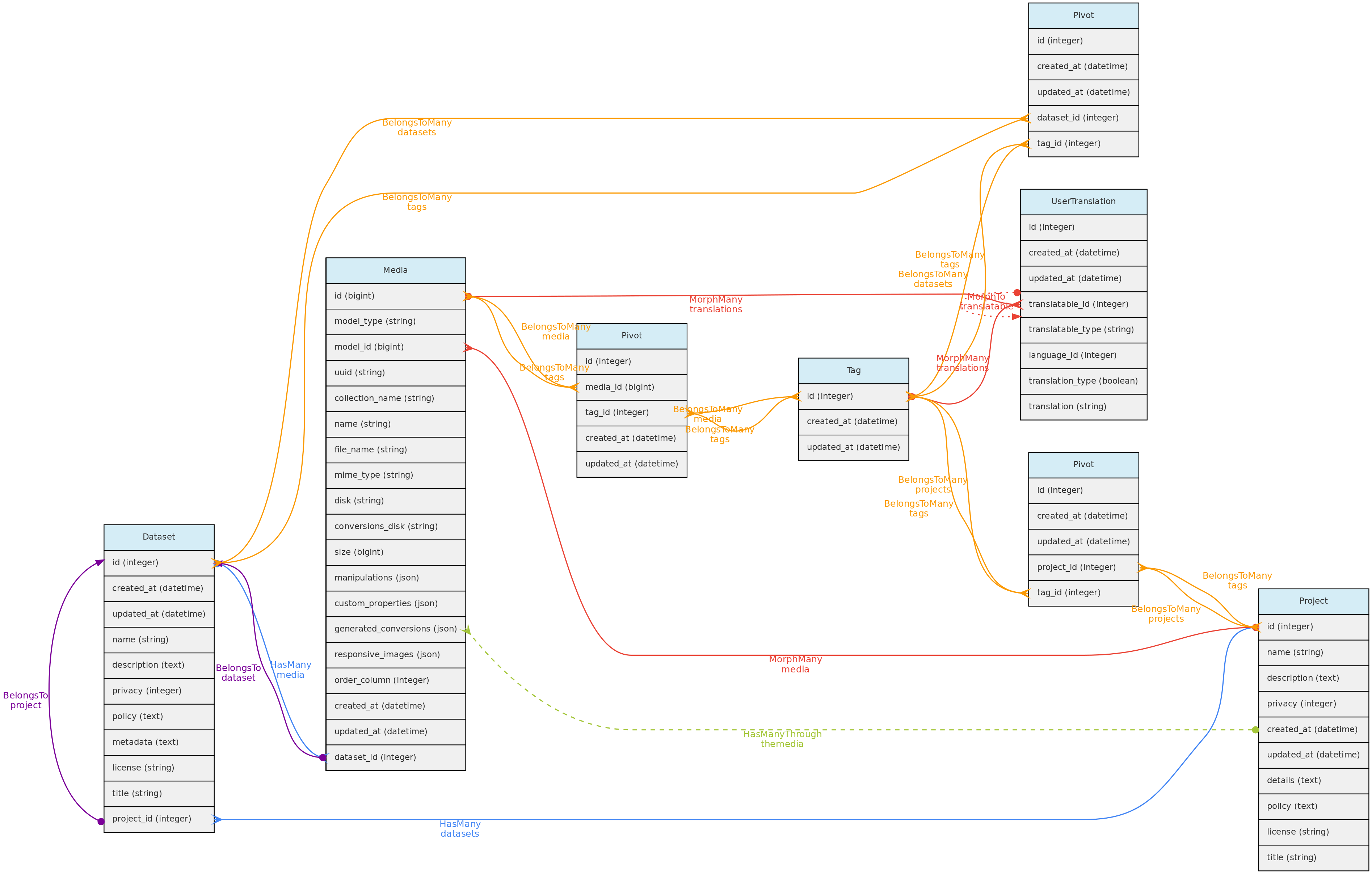
Acesso a dados usuários completos podem registrar tags, editar as inseridas e excluir as que não foram usadas. As tags têm acesso público, pois são apenas palavras-chave para facilitar a navegação.
Traduções do usuário
O modelo UserTranslation armazena as traduções de dados do usuário para: descrições e nomes de Variáveis e de categorias para variáveis categóricas; descrições de Arquivos de Mídia e para Tags. As relações com esses modelos são estabelecidas por relações polimórfica usando os campos translatable_type e translatable_id. Este modelo permite traduções para qualquer idioma listado na tabela languages, atualmente acessível apenas para inserção e edição diretamente no banco de dados SQL. Os formulários de entrada na interface web serão listados para os idiomas registrados.
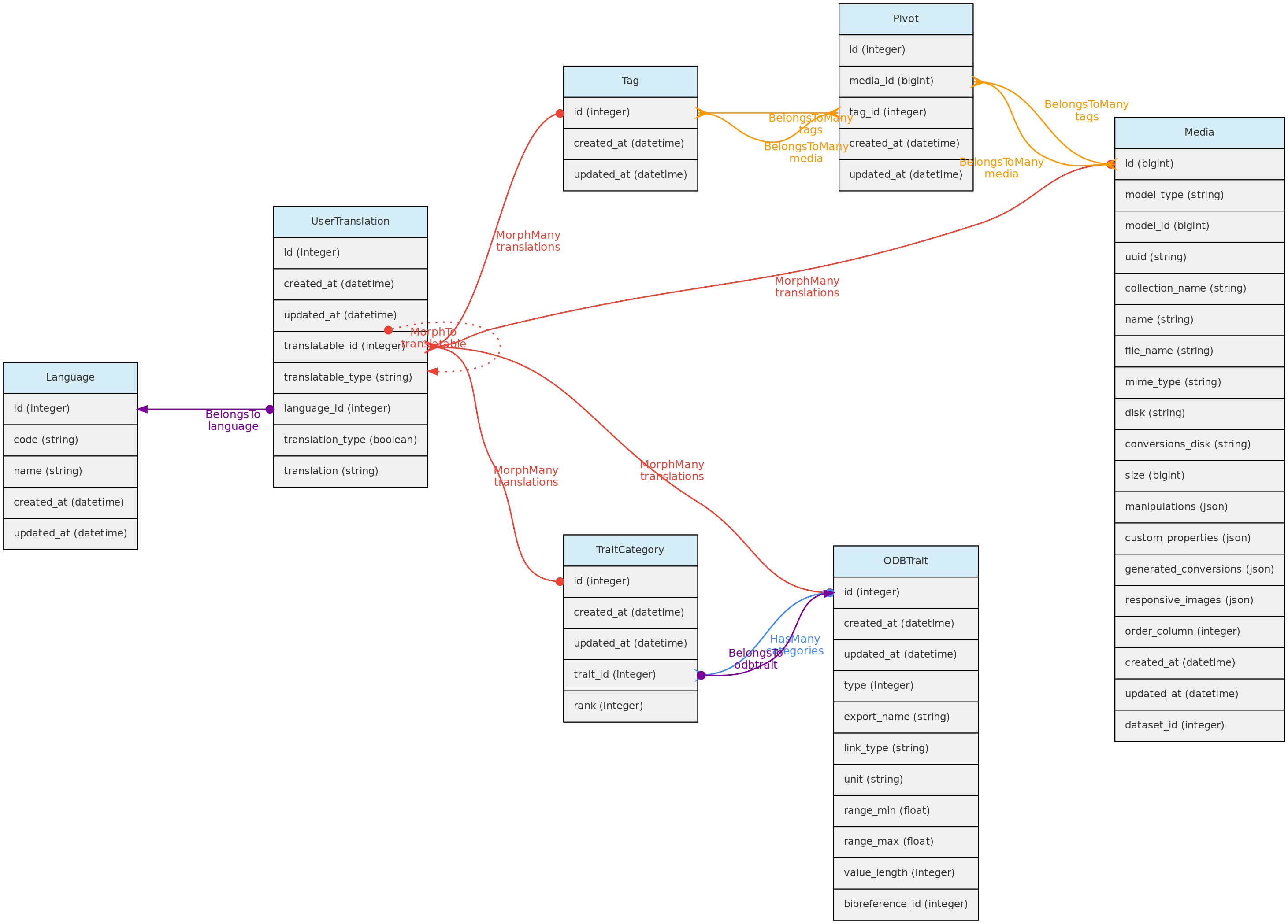
Datas incompletas
Datas para Vouchers, Indivíduos, Medições e Identificações podem ser incompletas, mas pelo menos ano é obrigatório em todos os casos. As colunas date nas tabelas são do tipo ‘date’ e as datas incompletas são armazenadas com 00 na parte ausente: ‘2005-00-00’ quando apenas o ano é conhecido; ‘1988-08-00’ quando apenas o mês é conhecido.
Auditando mudanças
As modificações nos registros do banco de dados são registradas na tabela activity_log. Esta tabela é gerada pelo pacote ActivityLog. As atividades são mostradas em um link ‘Histórico’ fornecido no show.view dos modelos.
- O pacote armazena as alterações como json no campo
properties, que contém dois elementos: attribute e old, que são basicamente os valores novos vs antigos que foram alterados. Essa estrutura deve ser respeitada.
- A classe ActivityFunctions contém funções personalizadas para ler as propriedades do registro Json armazenado na tabela
activity_log e encontra os valores para mostrar na tabela de dados History;
- A maioria das mudanças são registradas pelo pacote como um ’trait’ chamada dentro da classe. Estes permitem registrar automaticamente a maioria das atualizações e são configurados para registrar apenas os campos que foram alterados, não registros inteiros (opção
dirty). Além disso, a criação de registros não é anotada como atividade, apenas as alterações.
- Algumas alterações, como de coletores e de identificações indivíduos são registradas separadamente, pois envolvem tabelas relacionadas e o registro é especificado nos arquivos do Controlador;
- O registro contém um campo
log_name que agrupa os tipos de registro e é usado para distinguir os tipos de atividade e é útil para pesquisar a tabela de dados do histórico;
- Dois registros especiais também são feitos para Conjuntos de Dados:
- Qualquer download de um Conjunto de Dados pela interface é registrado, então os administradores podem rastrear quem e quando o conjunto de dados foi baixado;
- Qualquer solicitação de conjunto de dados também é registrada pelo mesmo motivo
O clean-command do pacote NÃO DEVE ser usado durante uma instalação em produção, caso contrário, apagará todas as alterações registradas. Se executado, apagará os logs anteriores ao tempo especificado no arquivo /config/activitylog.php.
A tabela ActivityLog tem a seguinte estrutura:
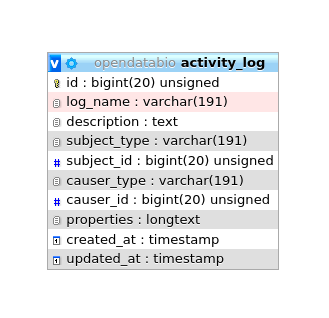
causer_type e causer_id serão o usuário que fez a alteraçãosubject_type e subject_id serão o modelo e registro alteradoslog_name - para agrupar logs e permite consultasdescription - um tanto redundante com log_name em um contexto OpenDataBio.properties - armazena as mudanças, por exemplo, e a mudança de identificação terá um log como:
{
"attributes":
{
"person_id":"2",
"taxon_id":"1424",
"modifier":"2",
"biocollection_id":"1",
"biocollection_reference":"1234",
"notes":"A new fake note has been inserted",
"date":"2020-02-08"},
"old":{
"person_id":674,
"taxon_id":1413,
"date":"1995-00-00",
"modifier":0,
"biocollection_id":null,
"notes":null,
"biocollection_reference":null
}
}

 Esta figura mostra os relacionamentos do modelo
Esta figura mostra os relacionamentos do modelo  As mesmas tabelas relacionadas com o modelo
As mesmas tabelas relacionadas com o modelo  Esta figura mostra o Modelo Indivíduo e os modelos aos quais ele se relaciona, exceto os modelos de Medição e Localidades, já que seus relacionamentos com Indivíduos são mostrados em outra parte desta página. As linhas indicam os
Esta figura mostra o Modelo Indivíduo e os modelos aos quais ele se relaciona, exceto os modelos de Medição e Localidades, já que seus relacionamentos com Indivíduos são mostrados em outra parte desta página. As linhas indicam os  As mesmas tabelas relacionadas com o modelo Indivíduo, suas colunas e as relações diretas e não polimóficas entre elas.
As mesmas tabelas relacionadas com o modelo Indivíduo, suas colunas e as relações diretas e não polimóficas entre elas. Modelo Taxon e suas relações. As linhas que ligam as tabelas indicam os
Modelo Taxon e suas relações. As linhas que ligam as tabelas indicam os  Modelo de voucher e suas relações. As linhas que ligam as tabelas indicam os
Modelo de voucher e suas relações. As linhas que ligam as tabelas indicam os 







 O modelo BibReference e seus relacionamentos. As linhas que ligam as tabelas indicam os
O modelo BibReference e seus relacionamentos. As linhas que ligam as tabelas indicam os  Modelo de identificação e suas relações. Linhas ligando tabelas indicam os
Modelo de identificação e suas relações. Linhas ligando tabelas indicam os  Modelo Persons e seus relacionamentos. As linhas que ligam as tabelas indicam os
Modelo Persons e seus relacionamentos. As linhas que ligam as tabelas indicam os 


