Este espaço do site OpenDataBio é para estudos de caso de uso do OpenDataBio pela interface, mas principalmente usando o OpenDataBio R package. Como contribuir com um tutorial.
This the multi-page printable view of this section. Click here to print.
Tutoriais
- 1: Obter dados via R
- 2: Importar dados via R
- 2.1: Importar Localidades
- 2.2: Importar Taxons
- 2.3: Importar Pessoas
- 2.4: Importar Variáveis
- 2.5: Importar Indivíduos & Vouchers
- 2.6: Importar Medições
1 - Obter dados via R
O pacote Opendatabio-R foi criado para permitir aos usuários interagir com um servidor OpenDataBio, para obter (GET) dados ou importar (POST) dados para a base de dados. Este tutorial é um exemplo básico de como obter dados.
Configure a conexão
- Configure a conexão com o servidor OpenDataBio usando a função
odb_config()do pacote. Os parâmetros mais importantes para esta função sãobase_url, que deve apontar para a URL da API do seu servidor OpenDataBio etoken, que é o token de acesso usado para autenticar seu usuário. - O
tokensó é necessário para obter dados de conjuntos de dados que possuem uma das políticas de acesso restrito. Os dados dos conjuntos de dados de acesso público podem ser extraídos sem a especificação do token. - Seu token está disponível em seu perfil na interface web
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
A configuração mais avançada envolve a definição de uma versão de API específica, um agente de usuário personalizado ou outros cabeçalhos HTTP, mas isso não é coberto aqui.
Teste sua conexão
A função odb_test() pode ser usada para verificar se a conexão foi bem sucedida e se
seu usuário foi identificado corretamente:
odb_test(cfg)
#will output
Host: https://opendb.inpa.gov.br/api/v0
Versions: server 0.9.1-alpha1 api v0
$message
[1] "Success!"
$user
[1] "admin@example.org"
Como alternativa, você pode especificar esses parâmetros como variáveis de sistema. Antes de iniciar o R, configure isso em seu shell (ou adicione ao final de seu arquivo .bashrc):
export ODB_TOKEN="YourToken"
export ODB_BASE_URL="https://opendb.inpa.gov.br/api"
export ODB_API_VERSION="v0"
Obter dados
Verifique a [Referência rápida da API GET]/docs/api/quick-reference) para obter uma lista completa de endpoints e parâmetros de solicitação.
Para dados de acesso público o token é opcional. Abaixo dois exemplos para Localidades e Taxons que são de acesso aberto. Siga um raciocínio semelhante para usar os demais endpoints. Veja a ajuda do pacote em R para todas as funções odb_get_{endpoint} disponíveis.
Obtendo nomes de táxons
Consulte GET API Taxon Endpoint para uma lista dos parâmetros de solicitação e uma lista de campos de resposta.
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
#get id for a taxon
mag.id = odb_get_taxons(params=list(name='Magnoliidae',fields='id,name'),odb_cfg = cfg)
#use this id to get all descendants of this taxon
odb_taxons = odb_get_taxons(params=list(root=mag.id$id,fields='id,scientificName,taxonRank,parent_id,parentName'),odb_cfg = cfg)
head(odb_taxons)
Algo como, dependo da sua base:
id scientificName taxonRank parent_id parentName
1 25 Magnoliidae Clado 20 Angiosperms
2 43 Canellales Ordem 25 Magnoliidae
3 62 Laurales Ordem 25 Magnoliidae
4 65 Magnoliales Ordem 25 Magnoliidae
5 74 Piperales Ordem 25 Magnoliidae
6 93 Chloranthales Ordem 25 Magnoliidae
Obtendo locais
Consulte GET API Location Endpoint para os parâmetros de solicitação e uma lista de campos de resposta.
Obtenha alguns campos listando todas as Unidades de Conservação (adm_level=99) registradas no servidor:
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
odblocais = odb_get_locations(params = list(fields='id,name,parent_id,parentName',adm_level=99),odb_cfg = cfg)
head(odblocais)
Se o servidor usar os dados de seed fornecidos o resultado será:
id name
1 5628 Estação Ecológica Mico-Leão-Preto
2 5698 Área de Relevante Interesse Ecológico Ilha do Ameixal
3 5700 Área de Relevante Interesse Ecológico da Mata de Santa Genebra
4 5703 Área de Relevante Interesse Ecológico Buriti de Vassununga
5 5707 Reserva Extrativista do Mandira
6 5728 Floresta Nacional de Ipanema
parent_id parentName
1 6 São Paulo
2 6 São Paulo
3 6 São Paulo
4 6 São Paulo
5 6 São Paulo
6 6 São Paulo
Pegar as parcelas importadas no tutorial de localidades.
Para obter um objeto espacial no R, use a função readWKT do pacote rgeos.
library(rgeos)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
locais = odb_get_locations(params=list(adm_level=100),odb_cfg = cfg)
locais[,c('id','locationName','parentName')]
colnames(locais)
for(i in 1:nrow(locais)) {
geom = readWKT(locais$footprintWKT[i])
if (i==1) {
plot(geom,main=locais$locationName[i],cex.main=0.8)
axis(side=1,cex.axis=0.5)
axis(side=2,cex.axis=0.5,las=2)
} else {
plot(geom,main=locais$locationName[i],add=T,col='red')
}
}
Figura gerada:

2 - Importar dados via R
O pacote Opendatabio-R foi criado para permitir aos usuários interagir com um servidor OpenDataBio, tanto para obter (GET) dados ou para importar (POST) dados para o banco de dados. Este tutorial é um exemplo básico de como importar dados.
Configure a conexão
- Configure a conexão com o servidor OpenDataBio usando a função
odb_config()do pacote. Os parâmetros mais importantes para esta função sãobase_url, que deve apontar para a URL da API do seu servidor OpenDataBio etoken, que é o token de acesso usado para autenticar seu usuário. - O
tokensó é necessário para obter dados de conjuntos de dados que possuem uma das políticas de acesso restrito. Os dados dos conjuntos de dados de acesso público podem ser extraídos sem a especificação do token. - Seu token está disponível em seu perfil na interface web
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
#create a config object
cfg = odb_config(base_url=base_url, token = token)
#test connection
odb_test(cfg)
Importar Dados (POST API)
Verifique a [Referência rápida da API]/docs/api/quick-reference) para obter uma lista completa dos POST endpoints e os campos necessários para importação de dados.
Funções de importação OpenDataBio-R
Todas as funções de importação têm a mesma assinatura: o primeiro argumento é um data.frame com os dados a serem importados, e o segundo parâmetro é um objeto de configuração gerado por odb_config.
Ao escrever uma solicitação de importação, verifique os documentos da API POST para entender quais colunas podem ser declaradas no data.frame.
Todas as funções de importação retornam um id do job, que pode ser usado para verificar se o job ainda está em execução, se terminou com sucesso ou se encontrou um erro. Este id de trabalho pode ser usado nas funções odb_get_jobs(), odb_get_affected_ids() e odb_get_log(), para encontrar detalhes sobre a submissão de importação (job). Você também pode ver o log em sua lista de trabalhos do usuário na interface da web.
Atenção
Ordem é importante - a importação correta de dados pode depender de registros já registrados. Por exemplo, importar um indivíduo com uma identidade de táxon requer o nome do táxon registrado no banco de dados, então primeiro valide sua lista de táxons usando a API GET e, em seguida, importe os registros para o Indivíduos.Trabalhando com datas e datas incompletas
Para Indivíduos, Vouchers e identificações, você pode usar datas incompletas.
O formato de data usado no OpenDataBio é AAA-MM-DD (ano - mês - dia), portanto, uma entrada válida seria 2018-05-28.
Particularmente em dados históricos, o dia (ou mês) exato pode não ser conhecido, então você pode substituir esses campos por NA: ‘1979-05-NA’ significa “um dia desconhecido, em maio de 1979” e ‘1979-NA- NA ‘significa “dia e mês desconhecidos, 1979”. Você não pode adicionar uma data para a qual tenha apenas o dia, mas pode, se tiver apenas o mês, se for realmente significativo de alguma forma.
2.1 - Importar Localidades
OpenDataBio é distribuído com um conjunto de dados de localidades para o Brasil, que inclui estados, municípios, unidades de conservação federais, terras indígenas e os biomas.
Trabalhar com dados espaciais é uma área delicada, por isso tentamos tornar o fluxo de trabalho para inserir Localidades o mais fácil possível.
Se você deseja fazer upload dos limites administrativos de um país, você também pode baixar um arquivo geojson em OSM-Boundaries e carregue-o diretamente através da interface da web. Ou use o repositório GADM exemplificado abaixo.
A importação é direta, mas os principais problemas a serem considerados:
- OpenDataBio armazena as geometrias de localidades usando representação de texto conhecido (WKT).
- As localidades são hierárquicas, portanto, uma localidade DEVE estar completamente dentro de sua localidade pai. O método de importação tentará detectar as localidades pai com base em sua geometria. Portanto, você não precisa informar um pai. No entanto, às vezes a localidade pai e a localidade filho compartilham uma borda ou têm pequenas erros que evitam a detecção. Portanto, se a importação não colocar o local onde você esperava, pode-se atualizar ou importar informando o pai correto. Quando você informar a localidade pai, uma segunda verificação será realizada adicionando um buffer à localidade pai e deverá resolver o problema.
- Os polígonos de países podem ser importados sem detecção ou definição dos pais, e registros marítimos podem ser vinculados a um pai, mesmo que não estejam contidos no polígono pai. Isso requer informar um campo específico (
ismarine) e deve ser usado nestes casos. - Padronizar a geometria para uma projeção comum de uso no sistema. Fortemente recomendado o uso de EPSG:4326 WGS84. Padronize antes de importar.
- Considere enviar seus polígonos político-administrativos antes de adicionar PONTOS, PLOTS ou TRANSECTOS específicos;
- Unidades de Conservação, Territórios Indígenas e Camadas Ambientais podem ser adicionados como locais e serão tratados como casos especiais, pois alguns desses locais abrangem diferentes unidades administrativas. Portanto, uma localidade de tipo POINT, PLOT ou TRANSECTpode pertencer a umA UC, umA TI e muitas camadas ambientais se estas estiverem armazenadas no banco de dados. Essas localidades relacionadas são detectadas automaticamente a partir da geometria da localidade.
Verifique a POST API de localidades para entender quais colunas podem ser declaradas ao importar localidades.
Adm_level define o tipo de localidade
O nível administrativo (adm_level) de um local é um número:
2para país;3à10como outras como ‘áreas administrativas’, seguindo a convenção OpenStreeMap para facilitar a importação de dados externos e traduções locais (À SER IMPLEMENTADO) . Portanto, para o Brasil, os códigos são (Estados = 4, Municípios = 8);999para locais de ‘POINT’ como waypoints GPS;101para transectos100é o código para PARCELAS e SUBPARCELAS;99é o código para Unidades de Conservação98para Territórios Indígenas97para polígonos ambientais (por exemplo, Floresta Ombrofila Densa ou Bioma Amazônia)
Importando polígonos espaciais
Limites administrativos do GADM
Limites administrativos também podem ser importados sem sair de R, obtendo dados de GDAM e usando as funções odb_import*
library(raster)
library(opendatabio)
#download áreas administrativas do GADM para um país
#get country codes
crtcodes = getData('ISO3')
bra = crtcodes[crtcodes$NAME%in%"Brazil",]
#define a path where to save the downloaded spatial data
path = "GADMS"
dir.create(path,showWarnings = F)
#o número de admin_levels em cada país varia
#obter todos os níveis existentes em seu computador
runit =T
level = 0
while(runit) {
ocrt <- try(getData('GADM', country=bra, level=level,path=path),silent=T)
if (class(ocrt)=="try-error") {
runit = FALSE
}
level = level+1
}
#read downloaded data and format to odb
files = list.files(path, full.name=T)
locations.to.odb = NULL
for(f in 1:length(files)) {
ocrt <- readRDS(files[f])
#class(ocrt)
#convert the SpatialPolygonsDataFrame to OpenDataBio format
ocrt.odb = opendatabio:::sp_to_df(ocrt) #only for GADM data
locations.to.odb = rbind(locations.to.odb,ocrt.odb)
}
#see without geometry
head(locations.to.odb[,-ncol(locations.to.odb)])
#you may add a note to location
locations.to.odb$notes = paste("Source gdam.org via raster::get_data()",Sys.Date())
#adjust the adm_level to fit the OpenStreeMap categories
ff = as.factor(locations.to.odb$adm_level)
(lv = levels(ff))
levels(ff) = c(2,4,8,9)
locations.to.odb$adm_level = as.vector(ff)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=locations.to.odb,odb_cfg=cfg)
Atenção: você pode querer verificar se há exclusividade de nome + pai em vez de apenas nome, já que nome + pai é uma combinação única. Você não pode salvar dois locais com o mesmo nome dentro do mesmo pai.
Example usando um shapefile
library(rgdal)
#read your shape file
path = 'mymaps'
file = 'myshapefile.shp'
layer = gsub(".shp","",file,ignore.case=TRUE)
data = readOGR(dsn=path, layer= layer)
#you may reproject the geometry to standard of your system if needed
data = spTransform(data,CRS=CRS("+proj=longlat +datum=WGS84"))
#convert polygons to WKT geometry representation
library(rgeos)
geom = rgeos::writeWKT(data,byid=TRUE)
#prep import
names = data@data$name #or the column name of the data
shape.to.odb = data.frame(name=names,geom=geom,stringsAsFactors = F)
#need to add the admin_level of these locations
shape.to.odb$admin_level = 2
#and may add parent and note if your want
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=shape.to.odb,odb_cfg=cfg)
Example importando de um KML
#read file as SpatialPolygonDataFrame
file = "myfile.kml"
file.exists(file)
mykml = readOGR(file)
geom = rgeos::writeWKT(mykml,byid=TRUE)
#prep import
names = mykml@data$name #or the column name of the data
to.odb = data.frame(name=names,geom=geom,stringsAsFactors = F)
#need to add the admin_level of these locations
to.odb$admin_level = 2
#and may add parent or any other valid field
#import
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=to.odb,odb_cfg=cfg)
Importar Parcelas e SubParcelas
Parcelas e transectos são casos especiais no OpenDataBio:
- Eles podem ser definidas com uma geometria do tipo Polygon ou LineString, respectivamente;
- Ou eles podem ser registrados apenas como localidaes de tipo POINT. Nesse caso, o OpenDataBio criará o polígono ou linestring para você;
- Dimensões (x e y) são armazenadas em metros para PARCELAS. Portanto elas devem ser quadradas ou retangulares.
- SubParcelas são localidades do tipo PARCELA tendo outra localidade do tipo PARCELA como pai e também devem ter posições cartesianas (startX, startY) dentro da localidade pai além das dimensões. A posição cartesiana refere-se às posições X e Y dentro da PARCELA pai e, portanto, DEVE ser menor do que o pai X e Y.
- SubParcela é o único tipo de localidade que pode ser registrado sem uma coordenada geográfica ou geometria, que será calculada a partir da geometria da PARCELA pai usando os valores startx e starty.
Parcela e SubParcela - exemplo 01
Você precisa de pelo menos uma coordenada geográfica para registrar uma localidade do tipo PLOT. A geometria (ou latitude e longitude) não pode estar vazia.
Este exemplo registra uma parcela em Manaus de 100x100m, informando sua geometria e depois importa algumas subparcelas sem especificação de geometria.
#geometry of a plot in Manaus
southWestCorner = c(-59.987747, -3.095764)
northWestCorner = c(-59.987747, -3.094822)
northEastCorner = c(-59.986835,-3.094822)
southEastCorner = c(-59.986835,-3.095764)
geom = rbind(southWestCorner,northWestCorner,northEastCorner,southEastCorner)
library(sp)
geom = Polygon(geom)
geom = Polygons(list(geom), ID = 1)
geom = SpatialPolygons(list(geom))
library(rgeos)
geom = writeWKT(geom)
to.odb = data.frame(name='A 1ha example plot',x=100,y=100,notes='a fake plot',geom=geom, adm_level = 100,stringsAsFactors=F)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=to.odb,odb_cfg=cfg)
Aguarde alguns segundos e, em seguida, importe subtramas para esta plotagem.
#importar subparcelas de 20x20m para a PARCELA acima sem indicar uma geometria.
(parent = odb_get_locations(params = list(name='A 1ha example plot',fields='id,name',adm_level=100),odb_cfg = cfg))
sub1 = data.frame(name='sub plot 40x40',parent=parent$id,x=20,y=20,adm_level=100,startx=40,starty=40,stringsAsFactors=F)
sub2 = data.frame(name='sub plot 0x0',parent=parent$id,x=20,y=20,adm_level=100,startx=0,starty=0,stringsAsFactors=F)
sub3 = data.frame(name='sub plot 80x80',parent=parent$id,x=20,y=20,adm_level=100,startx=80,starty=80,stringsAsFactors=F)
dt = rbind(sub1,sub2,sub3)
#import
odb_import_locations(data=dt,odb_cfg=cfg)
Capturas de tela das parcelas importadas
Abaixo capturas de tela para as parcelas importadas com o código acima
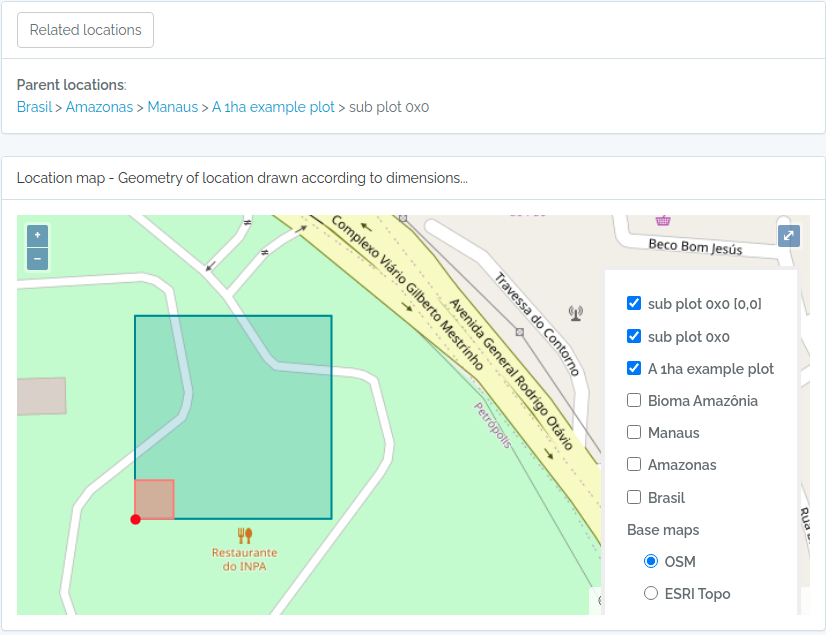
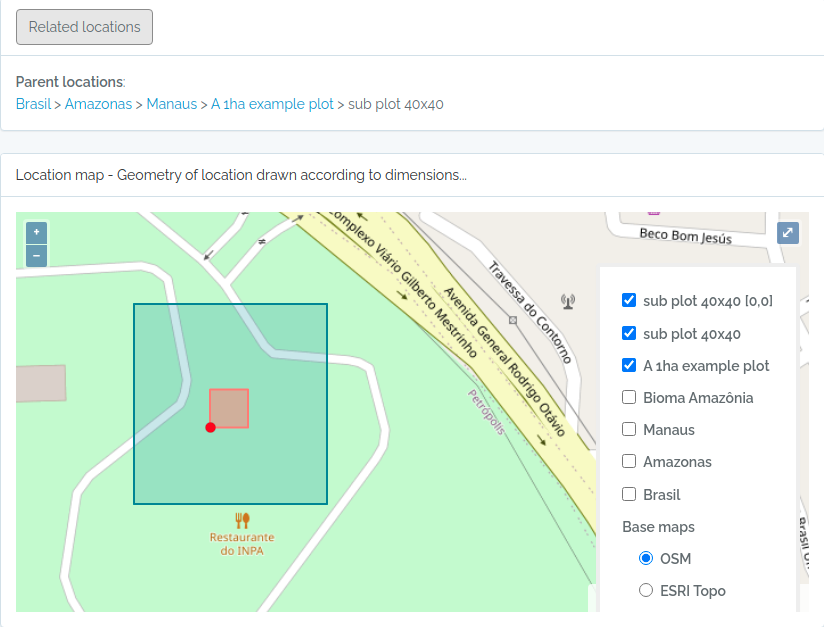
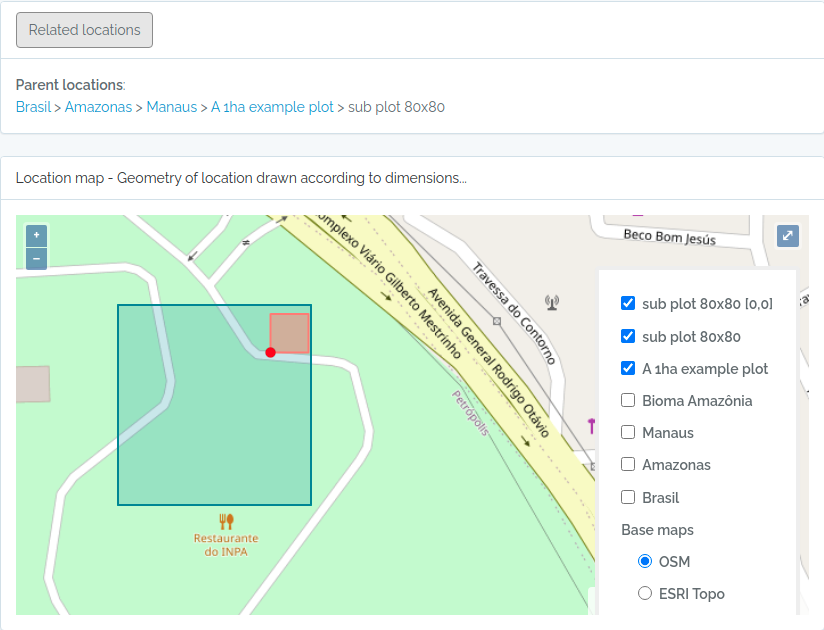
Parcela e SubParcela - exemplo 02
Importe uma parcela e suas subparcelas tendo apenas:
- a coordenada geográfica de um único ponto, representando a coordenada [0,0] da parcela.
- um azimute ou ângulo da direção da parcela (se não informar, Norte será usado
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
#the plot
geom = "POINT(-59.973841 -2.929822)"
to.odb = data.frame(name='Example Point PLOT',x=100, y=100, azimuth=45,notes='OpenDataBio point plot example',geom=geom, adm_level = 100,stringsAsFactors=F)
odb_import_locations(data=to.odb,odb_cfg=cfg)
#define 20x20 subplots cartesian coordinates
x = seq(0,80,by=20)
xx = rep(x,length(x))
yy = rep(x,each=length(x))
names = paste(xx,yy,sep="x")
#importar esses subplots sem ter uma geometria, mas especificando a localidade pai
parent = odb_get_locations(params = list(name='Example Point PLOT',adm_level=100),odb_cfg = cfg)
to.odb = data.frame(name=names,startx=xx,starty=yy,x=20,y=20,notes="OpenDataBio 20x20 subplots example",adm_level=100,parent=parent$id)
odb_import_locations(data=to.odb,odb_cfg=cfg)
#obter os locais importados usando o parâmetro root
locais = odb_get_locations(params=list(root=parent$id),odb_cfg = cfg)
locais[,c('id','locationName','parentName')]
colnames(locais)
for(i in 1:nrow(locais)) {
geom = readWKT(locais$footprintWKT[i])
if (i==1) {
plot(geom,main=locais$locationName[i],cex.main=0.8,col='yellow')
axis(side=1,cex.axis=0.7)
axis(side=2,cex.axis=0.7,las=2)
} else {
plot(geom,add=T,border='red')
}
}
A figura gerada acima:
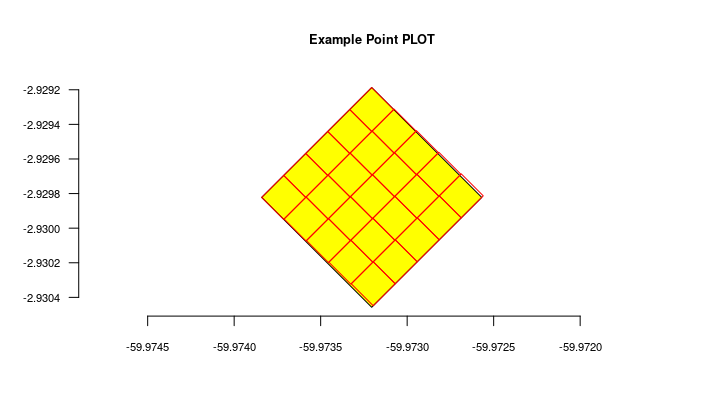
Importar transectos
Este código importará dois transectos, um definido por uma geometria (LINESTRING), e o outro apenas por uma única coordenada geográfica (POINT). Veja as figuras abaixo para o resultado importado.
#geometry of transect in Manaus
#read trail from a kml file
#library(rgdal)
#file = "acariquara.kml"
#file.exists(file)
#mykml = readOGR(file)
#library(rgeos)
#geom = rgeos::writeWKT(mykml,byid=TRUE)
#above will output:
geom = "LINESTRING (-59.9616459699999993 -3.0803612500000002, -59.9617394400000023 -3.0805952900000002, -59.9618530300000003 -3.0807376099999999, -59.9621049400000032 -3.0808563200000001, -59.9621949100000009 -3.0809758500000002, -59.9621587999999974 -3.0812666800000001, -59.9621092399999966 -3.0815010400000000, -59.9620656999999966 -3.0816403499999998, -59.9620170600000009 -3.0818584699999998, -59.9620740699999999 -3.0819864099999998)";
#prep data frame
#o valor y refere-se a um buffer em metros aplicado à trilha
#y é usado para validar a inserção de indivíduos relacionados
to.odb = data.frame(name='A trail-transect example',y=20, notes='OpenDataBio transect example',geom=geom, adm_level = 101,stringsAsFactors=F)
#import
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=to.odb,odb_cfg=cfg)
#IMPORTA UM SEGUNDO TRANSECTO SEM GEOMETRIA
# então você precisa informar o valor x, que é o comprimento do transecto
#ODB irá mapear este transecto orientado pelo parâmetro azimute (sul no exemplo abaixo)
#point geometry = ponto inicial
geom = "POINT(-59.973841 -2.929822)"
to.odb = data.frame(name='A transect point geometry',x=300, y=20, azimuth=180,notes='OpenDataBio point transect example',geom=geom, adm_level = 101,stringsAsFactors=F)
odb_import_locations(data=to.odb,odb_cfg=cfg)
locais = odb_get_locations(params=list(adm_level=101),odb_cfg = cfg)
locais[,c('id','locationName','parentName','levelName')]
O código acima resultará nas duas localidades a seguir:
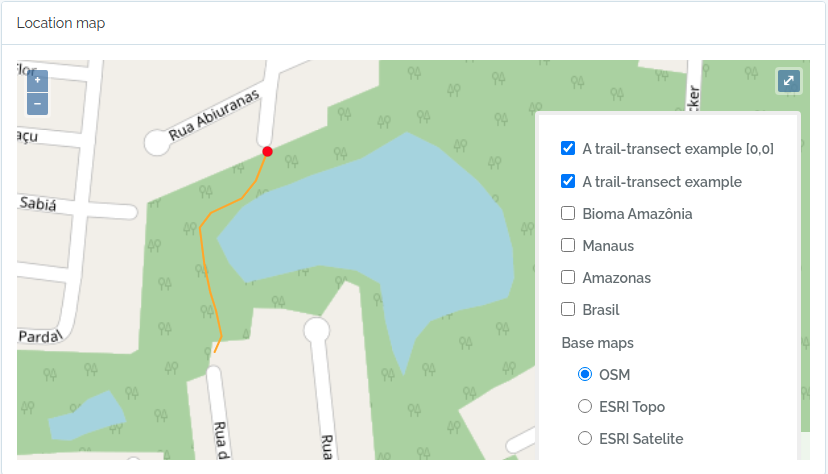
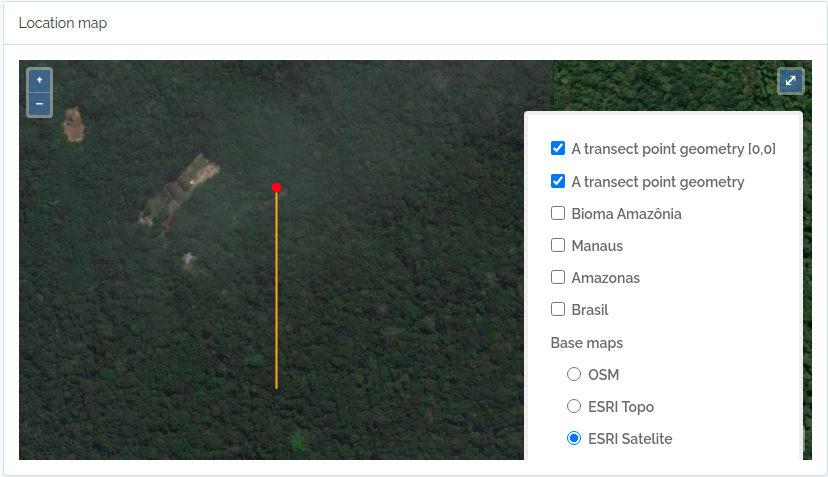
2.2 - Importar Taxons
Atenção
- Para uma importação bem-sucedida de nomes taxomicos publicados você só precisa fornecer um
name, sendo o nome completo para espécies e infra-espécies (por exemplo, Licaria canella tenuicarpa, Ocotea delicata), da mesma forma quecanonicalNamedo GBIF, ou seja, não inclui os ranks infra-específicos; - Não há necessidade de importar táxons pais para nomes publicados, OpenDataBio faz isso para você. O processo de importação usará o
nameque você informou para recuperar informações de táxons de um repositório de nomenclatura: atualmente Tropicos, GBIF BackBone Taxonomy e ZOOBANK. Se o nome do táxon for encontrado, ele obterá automaticamente as informações necessárias e também todos os táxons pais (ou nomes aceitos se estiver marcado como sinônimo) conforme necessário, e armazenará no banco de dados para você. Se onamefor inválido, ou seja, é sinônimo de outro nome, ele será importado, mas marcado como inválido. - Se informar
parente for diferente do detectado pela API o pai informado tem preferência. - Para espécies não publicadas
level,parent, e umapersonouauthor_idtambém devem ser fornecidos; - A API externa usará uma consulta difusa durante a pesquisa de GBIF se não encontrar uma correspondência exata, permitindo detectar até mesmo quando está com erros ortográficos. No entanto, erros de grafia nos nomes podem impedir que um táxon seja armazenado.
- A taxonomia é hierárquica e a estrutura dentro do OpenDataBio pode ser semelhante a uma árvore. Portanto, você pode adicionar, se quiser, qualquer nó da árvore da vida na tabela Taxon. Para isso, você precisa usar o nível do táxon
Clade. Mas observe que isso pode não ser relevante se você não vincular dados a esses nomes de clados. - Como a verificação da nomenclatura é demorada, seja paciente.
OpenDataBio é distribuído com uma tabela semente de Taxons, que inclui a raiz de todos os reinos e alguns nós da filogenia das plantas, ou seja, o os nós da àrvore no nível de ordem para Angiospermas - APWeb Tree.
Um exemplo simples de nome publicado
Os scripts abaixo foram testados, estando a base já populada com até o nível de Ordem para Angiospermas.
Na tabela de táxons, as famílias Moraceae, Lauraceae e Solanaceae ainda não estavam registradas:
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
exists = odb_get_taxons(params=list(root="Moraceae,Lauraceae,Solanaceae"),odb_cfg=cfg)
Retornou:
data frame with 0 columns and 0 rows
Agora importe algumas espécies e uma infraespécie para as famílias acima, especificando seu nome completo (canonicalName):
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
spp = c("Ficus schultesii", "Ocotea guianensis","Duckeodendron cestroides","Licaria canella tenuicarpa")
splist = data.frame(name=spp)
odb_import_taxons(splist, odb_cfg=cfg)
Agora verifique se foi importado (note o argumento root):
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
exists = odb_get_taxons(params=list(root="Moraceae,Lauraceae,Chrysobalanaceae"),odb_cfg=cfg)
head(exists[,c('id','scientificName', 'taxonRank','taxonomicStatus','parentNameUsage')])
Retorno:
id scientificName taxonRank taxonomicStatus parentName
1 252 Moraceae Family accepted Rosales
2 253 Ficus Genus accepted Moraceae
3 254 Ficus schultesii Species accepted Ficus
4 258 Solanaceae Family accepted Solanales
5 259 Duckeodendron Genus accepted Solanaceae
6 260 Duckeodendron cestroides Species accepted Duckeodendron
7 255 Lauraceae Family accepted Laurales
8 256 Ocotea Genus accepted Lauraceae
9 257 Ocotea guianensis Species accepted Ocotea
10 261 Licaria Genus accepted Lauraceae
11 262 Licaria canella Species accepted Licaria
12 263 Licaria canella subsp. tenuicarpa Subspecies accepted Licaria canella
Observe que embora tenhamos especificado apenas os nomes das espécies e infra-espécies, a API importou também toda a hierarquia parental necessária até a família, porque as ordens já estavam registradas.
Um exemplo de nome publicado inválido
O nome Licania octandra pallida (Chrysobalanaceae) foi recentemente movido com sinônimo de Leptobalanus octandrus pallidus.
O roteiro a seguir exemplifica o que acontece nesses casos.
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
#lets check
exists = odb_get_taxons(params=list(root="Chrysobalanaceae"),odb_cfg=cfg)
exists
#in this test returns an empty data frame
#data frame with 0 columns and 0 rows
#now import
spp = c("Licania octandra pallida")
splist = data.frame(name=spp)
odb_import_taxons(splist, odb_cfg=cfg)
#see the results
exists = odb_get_taxons(params=list(root="Chrysobalanaceae"),odb_cfg=cfg)
exists[,c('id','scientificName', 'taxonRank','taxonomicStatus','parentName')]
Retorno:
id scientificName taxonRank taxonomicStatus parentName
1 264 Chrysobalanaceae Family accepted Malpighiales
2 265 Leptobalanus Genus accepted Chrysobalanaceae
3 267 Leptobalanus octandrus Species accepted Leptobalanus
4 269 Leptobalanus octandrus subsp. pallidus Subspecies accepted Leptobalanus octandrus
5 266 Licania Genus accepted Chrysobalanaceae
6 268 Licania octandra Species invalid Licania
7 270 Licania octandra subsp. pallida Subspecies invalid Licania octandra
Observe que, embora tenhamos especificado apenas um nome de infra-espécie, a API importou também toda a hierarquia pai necessária até a família e, como o nome é inválido, também importou o nome aceito para esta infra-espécie e seus pais.
Uma espécie ou morfotipo não publicado
É comum ter nomes de espécies locais não publicados (morfotipos) para plantas em parcelas, ou ainda trabalhos taxonômicos ainda não publicados. As designações não publicadas são específicas do projeto e, portanto, DEVEM também fornecer um autor, pois diferentes projetos podem usar o mesmo código ‘sp.1’ ou ‘sp.A’ para seus táxons não publicados.
Você pode vincular um nome não publicado como qualquer nível de táxon e não precisa usar a lógica de gênero + espécie para atribuir um morfotipo para o qual o gênero ou taxonomia de nível superior é indefinida. Por exemplo, você pode armazenar um nível de ’espécie’ com o nome ‘Indet sp.1’ e parent_name ‘Laurales’, se a determinação formal de nível mais baixo que você tem é o nível de ordem. Neste exemplo, não há necessidade de armazenar um gênero Indet e táxons da família Indet apenas para contabilizar este morfotipo não identificado.
##assign an unpublished name for which you only know belongs to the Angiosperms and you have this node in the Taxon table already
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
#check that angiosperms exist
odb_get_taxons(params=list(name='Angiosperms'),odb_cfg = cfg)
#if it is there, start creating a data.frame to import
to.odb = data.frame(name='Morphotype sp.1', parent='Angiosperms', stringsAsFactors=F)
#get species level numeric code
to.odb$level=odb_taxonLevelCodes('species')
#you must provide an author that is a Person in the Person table. Get from server
odb.persons = odb_get_persons(params=list(search='João Batista da Silva'),odb_cfg=cfg)
#found
head(odb.persons)
#add the author_id to the data.frame
#NOTE it is not author, but author_id or person)
#this makes odb understand it is an unpublished name
to.odb$author_id = odb.persons$id
#import
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
odb_import_taxons(to.odb,odb_cfg = cfg)
Verifique o registro importado:
exists = odb_get_taxons(params=list(name='Morphotype sp.1'),odb_cfg = cfg)
exists[,c('id','scientificName', 'taxonRank','taxonomicStatus','parentName','scientificNameAuthorship')]
Algumas colunas para o registro importado:
id scientificName taxonRank taxonomicStatus parentName scientificNameAuthorship
1 276 Morphotype sp.1 Species unpublished Angiosperms João Batista da Silva - Silva, J.B.D.
Importar um clado publicado
Você pode adicionar um clado Taxon e pode referenciar uma publicação usando a entrada bibkey. Portanto, é possível armazenar de fato todos os nós relevantes de qualquer filogenia na hierarquia do Taxon.
#parent já deve estar armazenado
odb_get_taxons(params=list(name='Pagamea'),odb_cfg = cfg)
#define o clado a ser armazenado
to.odb = data.frame(name='Guianensis core', parent_name='Pagamea', stringsAsFactors=F)
to.odb$level = odb_taxonLevelCodes('clade')
#adicione uma referência à publicação onde está publicado
#importe a referência do bib para o banco de dados de antemão
odb_get_bibreferences(params(bibkey='prataetal2018'),odb_cfg=cfg)
to.odb$bibkey = 'prataetal2018'
#então adicione nomes de espécies válidos como filhos deste clado em vez do nível de gênero
children = data.frame(name = c('Pagamea guianensis','Pagamea angustifolia','Pagamea puberula'),stringsAsFactors=F)
children$parent_name = 'Guianensis core'
children$level = odb_taxonLevelCodes('species')
children$bibkey = NA
#merge
to.odb = rbind(to.odb,children)
#import
odb_import_taxons(to.odb,odb_cfg = cfg)
2.3 - Importar Pessoas
Ver o POST Persons API docs para entender quais colunas podem ser declaradas ao importar Pessoas.
A API verificará por abreviações idênticas, que é a única restrição da classe Pessoa. Abreviações são exclusivas e duplicações não são permitidas. Isso não impede que os dados baixados de repositórios tenham abreviações ou nomes completos diferentes para a mesma pessoa. Portanto, você deve padronizar os dados secundários antes de importá-los para o servidor para minimizar esses erros comuns.
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
one = data.frame(full_name='Adolpho Ducke',abbreviation='DUCKE, A.',notes='Grande botânico da Amazônia',stringsAsFactors = F)
two = data.frame(full_name='Michael John Gilbert Hopkins',abbreviation='HOPKINKS, M.J.G.',notes='Curador herbário INPA',stringsAsFactors = F)
to.odb= rbind(one,two)
odb_import_persons(to.odb,odb_cfg=cfg)
#pode adicionar um email
Pegar os dados
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
persons = odb_get_persons(odb_cfg=cfg)
persons = persons[order(persons$id,decreasing = T),]
head(persons,2)
resultado:
id full_name abbreviation email institution notes
613 1582 Michael John Gilbert Hopkins HOPKINKS, M.J.G. <NA> NA Curador herbário INPA
373 1581 Adolpho Ducke DUCKE, A. <NA> NA Grande botânico da Amazônia
2.4 - Importar Variáveis
Atenção
- Recomendamos fortemente que você use a interface da web para inserir variáveis uma por uma. Use importações em lote somente se você tiver muitas variáveis.
- Antes de importar qualquer variável certifique-se de que ela já não esteja cadastrada no sistema com um nome diferente. Prevenir a duplicação é importante porque a classe Trait é compartilhada entre todos os usuários de uma instalação OpenDataBio. Isso também significa que, uma vez que uma variável é usada para Medições, você só poderá alterar a definição da variável, por exemplo, um nome de uma categoria, se você for o único usuário que inseriu medições para a variável. Caso contrário, outra pessoa usou essa definição e você não poderá alterá-la.
- export_name deve ser exclusivo em uma única instalação ODB. Este nome é usado ao exportar dados e em formulários, e você deve considerar torná-lo o mais curto e informativo (de definição) possível. Além disso, a API validará o nome da exportação - deve ser camelCase, PascalCase ou snake_case, e você não pode adicionar espaços ou qualquer caractere especial (acentos). Ex. ‘dbh’, ‘dbhPom’, ’leafLength’, ‘LeafLength’ ou ’leaf_length’, respectivamente.
As características podem ser importadas usando odb_import_traits().
Leia atentamente o Traits POST API.
Tipos de variáveis
Veja odb_traitTypeCodes() para os códigos numéricos possíveis tipos de variáveis
Traduções Nome da Variável e de Categorias
Os campos name e description podem ter um dos seguintes conteúdos:
- usando o código do idioma como chaves:
list("en" = "Diameter at Breast Height","pt-br" ="Diâmetro a Altura do Peito") - ou usando os nomes dos idiomas como chaves:
list("English" ="Diameter at Breast Height","Portuguese" ="Diâmetro a Altura do Peito").
O campo categories deve incluir para cada categoria + classificação + idioma os seguintes campos: lang= misto - obrigatório, o id, código ou nome do idioma da traduçãoname= string - obrigatório, o nome da categoria traduzido obrigatório (name + rank + lang deve ser único)rank= número - obrigatório, a classificação é importante para indicar a mesma categoria entre os idiomas e define variáveis ordinais;description= string - opcional para categorias, uma definição da categoria.
Isso pode ser formatado como um data.frame e colocado na coluna categories de outro data.frame:
data.frame(
rbind(
c("lang"="en","rank"=1,"name"="small","description"="smaller than 1 cm"),
c("lang"="pt-br","rank"=1,"name"="pequeno","description"="menor que 1 cm"),
c("lang"="en","rank"=2,"name"="big","description"="bigger than 10 cm"),
c("lang"="pt-br","rank"=2,"name"="grande","description"="maior que 10 cm")
),
stringsAsFactors=FALSE
)
Variável quantitativa
Para variáveis quantitativas para valores integers ou real (type 0 ou 1).
odb_traitTypeCodes()
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#do this first to build a correct data.frame as it will include translations list
to.odb = data.frame(type=1,export_name = "dbh", unit='centimeters',stringsAsFactors = F)
#add translations (note double list)
#format is language_id = translation (and the column be a list with the translation lists)
to.odb$name[[1]]= list('1' = 'Diameter at breast height', '2' = 'Diâmetro à altura do peito')
to.odb$description[[1]]= list('1' = 'Stem diameter measured at 1.3m height','2' = 'Diâmetro do tronco medido à 1.3m de altura')
#measurement validations
to.odb$range_min = 10 #this will restrict the minimum measurement value allowed in the trait
to.odb$range_max = 400 #this will restrict the maximum value
#measurements can be linked to (classes concatenated by , or a list)
to.odb$objects = "Individual,Voucher,Taxon" #makes no sense link such measurements to Locations
to.odb$notes = 'this is quantitative trait example'
#import
odb_import_traits(to.odb,odb_cfg=cfg)
Variável categórica
- Deve incluir categorias. A única diferença entre as características ordinais e categóricas é que as categorias ordinais terão um rank, ou ordem. Observe que as variáveis ordinais são semiquantitativas e, portanto, se você tiver categorias, pergunte-se se elas não são realmente ordinais e registre de acordo.
- Assim como o
namee adescriptionda variável, as categorias também podem ter traduções em diferentes idiomas, e você DEVE inserir as traduções para os idiomas disponíveis (odb_get_languages ()) para que a variável esteja acessível em todos os idiomas. O inglês é obrigatório, portanto, pelo menos o nome em inglês deve ser informado. As categorias podem ter uma descrição associada, mas às vezes o nome da categoria é autoexplicativo, portanto, as descrições das categorias não são obrigatórias.
odb_traitTypeCodes()
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#do this first to build a correct data.frame as it will include translations list
#do this first to build a correct data.frame as it will include translations list
to.odb = data.frame(type=3,export_name = "specimenFertility", stringsAsFactors = F)
#trait name and description
to.odb$name = data.frame("en"="Specimen Fertility","pt-br"="Fertilidade do especímene",stringsAsFactors=F)
to.odb$description = data.frame("en"="Kind of reproductive stage of a collected plant","pt-br"="Estágio reprodutivo de uma amostra de planta coletada",stringsAsFactors=F)
#categories (if your trait is ORDINAL, the add categories in the wanted order here)
categories = data.frame(
rbind(
c('en',1,"Sterile"),
c('pt-br',1,"Estéril"),
c('en',2,"Flowers"),
c('pt-br',2,"Flores"),
c('en',3,"Fruits"),
c('pt-br',3,"Frutos"),
c('en',4,"Flower buds"),
c('pt-br',4,"Botões florais")
),
stringsAsFactors =FALSE
)
colnames(categories) = c("lang","rank","name")
#descriptions not included for categories as they are obvious,
# but you may add a 'description' column to the categories data.frame
#objects for which the trait may be used for
to.odb$objects = "Individual,Voucher"
to.odb$notes = 'a fake note for a multiselection categorical trait'
to.odb$categories = list(categories)
#import
odb_import_traits(to.odb,odb_cfg=cfg)
Variável tipo LINK
Um variável de tipo LINK permite vincular um Táxon ou Voucher como uma medição de outro objeto. Por exemplo, você pode conduzir um inventário de plantas para o qual possui apenas contagens para o táxon associado a uma localidade. Portanto, você pode criar uma variável do tipo LINK, que permitirá que você armazene os valores de contagem para qualquer Táxon como medições para um local específico (PONTO, POLÍGONO).
Use a interface ou siga o exemplo para as demais variáveis.
Variáveis do tipo texto ou cor
Variáveis de texto permitem o armazenamento de observações textuais. A cor permitirá apenas códigos de cores.
odb_traitTypeCodes()
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
to.odb = data.frame(type=5,export_name = "taxonDescription", stringsAsFactors = F)
#trait name and description
to.odb$name = data.frame("en"="Taxonomic descriptions","pt-br"="Descrições taxonômicas",stringsAsFactors=F)
to.odb$description = data.frame("en"="Taxonomic descriptions from the literature","pt-br"="Descrições taxonômicas da literatura",stringsAsFactors=F)
#will only be able to use this trait for a measurment associated with a Taxon
to.odb$objects = "Taxon"
#import
odb_import_traits(to.odb,odb_cfg=cfg)
Variáveis espectrais
Variáveis espectrais são específicos para dados espectrais. Você deve especificar a amplitude dos números de ondas para os quais você pode ter dados de absorbância ou refletância e o comprimento dos espectros a serem armazenados como medições para permitir a validação durante a entrada. Portanto, para cada intervalo e espaçamento dos valores espectrais que você tem, uma variável ESPECTRAL diferente deve ser criada.
Use a interface ou siga o exemplo para as demais variáveis.
2.5 - Importar Indivíduos & Vouchers
Attention
- Você pode importar Vouchers juntamente com o registro do Indivíduo. Use o endpoint POST-Voucher apenas para indivíduos já registrados, caso contrário, siga abaixo para importar indivíduos e seus vouchers de uma só vez.
- Os indivíduos podem ter uma identificação taxonômica própria ou uma identificação taxonômica dependente
- Os indivíduos podem ter vários locais, mas ao registrar o indivíduo, um único local é necessário. Você pode então importar locais adicionais usando o Individual-location POST API.
Indivíduos podem ser importados usando odb_import_individuals() e vouchers com odb_import_vouchers().
Leia atentamente o Individual POST API e o Voucher POST API.
Exemplo simples
Inventando dados para 1 registro de um indivíduo:
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#the number in the aluminium tag in the forest
to.odb = data.frame(tag='3405.L1', stringsAsFactors=F)
#the collectors (get ids from the server)
(joao = odb_get_persons(params=list(search='joao batista da silva'),odb_cfg=cfg)$id)
(ana = odb_get_persons(params=list(search='ana cristina sega'),odb_cfg=cfg)$id)
#ids concatenated by | pipe
to.odb$collector = paste(joao,ana,sep='|')
#tagged date (lets use an incomplete).
to.odb$date = '2018-07-NA'
#lets place in a Plot location imported with the Location post tutorial
plots = odb_get_locations(params=list(name='A 1ha example plot'),odb_cfg=cfg)
head(plots)
to.odb$location = plots$id
#relative position within parent plot
to.odb$x = 10.4
to.odb$y = 32.5
#or could be
#to.odb$relative_position = paste(x,y,sep=',')
#taxonomic identification
taxon = 'Ocotea guianensis'
#check that exists
(odb_get_taxons(params=list(name='Ocotea guianensis'),odb_cfg=cfg)$id)
#person that identified the individual
to.odb$identifier = odb_get_persons(params=list(search='paulo apostolo'),odb_cfg=cfg)$id
#or you also do to.odb$identifier = "Assunção, P.A.C.L."
#the used form only guarantees the persons is there.
#may add modifers as well [may need to use numeric code instead]
to.odb$modifier = 'cf.'
#or check with to see you spelling is correct
odb_detModifiers()
#and submit the numeric code instaed
to.odb$modifier = 3
#an incomplete identification date
to.odb$identification_date = list(year=2005)
#or to.odb$identification_date = "2005-NA-NA"
Importando esse indivíduo
odb_import_individuals(to.odb,odb_cfg = cfg)
#lets import this individual
odb_import_individuals(to.odb,odb_cfg = cfg)
#check the job status
odb_get_jobs(params=list(id=130),odb_cfg = cfg)
Ops, esqueci de informar um dataset e meu usuário não tem um dataset default definido.
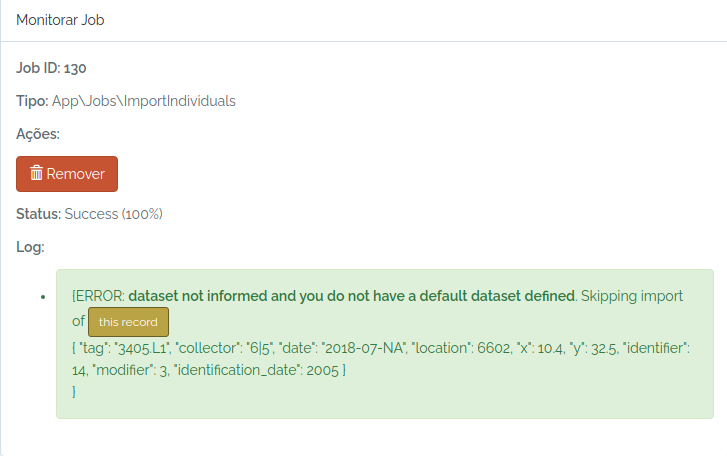
Então, eu apenas informo um conjunto de dados existente e tento novamente:
dataset = odb_get_datasets(params=list(name="Dataset test"),odb_cfg=cfg)
dataset
to.odb$dataset = dataset$id
odb_import_individuals(to.odb,odb_cfg = cfg)
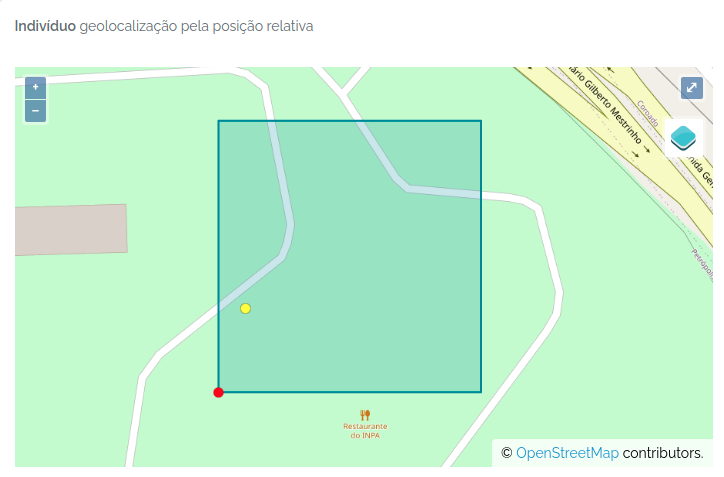
Importando Indivíduos e Vouchers de uma vez
Indivíduos são o objeto real que possui a maior parte das informações relacionadas aos Vouchers, que são amostras em uma Biocoleção. Portanto, você pode importar um registro de um indivíduo com a especificação de um ou mais vouchers.
#a fake plant record somewhere in the Amazon
aplant = data.frame(taxon="Duckeodendron cestroides", date="2021-09-09", latitude=-2.34, longitude=-59.845,angle=NA,distance=NA, collector="Oliveira, A.A. de|João Batista da Silva", tag="3456-A",dataset=1)
#a fake set of vouchers for this individual
herb = data.frame(biocollection=c("INPA","NY","MO"),biocollection_number=c("12345A","574635","ANOTHER FAKE CODE"),biocollection_type=c(2,3,3))
#add this dataframe to the object
aplant$biocollection = NA
aplant$biocollection = list(herb)
#another fake plant
asecondplant = data.frame(taxon="Ocotea guianensis", date="2021-09-09", latitude=-2.34, longitude=-59.89,angle=240,distance=50, collector="Oliveira, A.A. de|João Batista da Silva", tag="3456",dataset=1)
asecondplant$biocollection = NA
#merge the fake data
to.odb = rbind(aplant,asecondplant)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
odb_import_individuals(to.odb, odb_cfg=cfg)
Verifique os dados importados
O script acima criou registros para o modelo Indivíduo e Voucher:
#get the imported individuals using a wildcard
inds = odb_get_individuals(params = list(tag='3456*'),odb_cfg = cfg)
inds[,c("basisOfRecord","scientificName","organismID","decimalLatitude","decimalLongitude","higherGeography") ]
Retorna:
basisOfRecord scientificName organismID decimalLatitude decimalLongitude higherGeography
1 Organism Ocotea guianensis 3456 - Oliveira - UnnamedPoint_5989234 -2.3402 -59.8904 Brasil | Amazonas | Rio Preto da Eva
2 Organism Duckeodendron cestroides 3456-A - Oliveira - UnnamedPoint_5989234 -2.3400 -59.8900 Brasil | Amazonas | Rio Preto da Eva
E Vouchers
#get the vouchers imported with the first plant data
vouchers = odb_get_vouchers(params = list(individual=inds$id),odb_cfg = cfg)
vouchers[,c("basisOfRecord","scientificName","organismID","collectionCode","catalogNumber") ]
Retorna:
basisOfRecord scientificName occurrenceID collectionCode catalogNumber
1 PreservedSpecimens Duckeodendron cestroides 3456-A - Oliveira -INPA.12345A INPA 12345A
2 PreservedSpecimens Duckeodendron cestroides 3456-A - Oliveira -MO.ANOTHER FAKE CODE MO ANOTHER FAKE CODE
3 PreservedSpecimens Duckeodendron cestroides 3456-A - Oliveira -NY.574635 NY 574635
Importar Vouchers para Indivíduos registrados
Atenção
- Vouchers são amostras de Indivíduos depositados em BioColeções. Se a Biocoleção for um recurso genético, o Voucher pode representar apenas uma amostra de DNA ou tecido.
- Portanto, os Vouchers devem pertencer a um Indivíduo e à uma Biocoleção, que são as informações obrigatórias para o cadastro de Vouchers. Coletores, data de coleta, localização e identidade taxonômica podem simplesmente ser extraídos do cadastro do Indivíduo.
- O Voucher pode ter coletor, número de coletor e data de coleta próprios, distintos do Indivíduo a que pertence, quando necessário.
Os campos obrigatórios são:
individual= id do indivíduo ou nome completo (organismID);biocollection= sigla ou id da Biocoleção - useodb_get_biocollections()para verificar se está registrado, caso contrário, primeiro registre a Biocoleção no banco de dados;
Para campos adicionais veja Voucher POST API.
Uma importação de voucher simples
#a holotype voucher with same collector and date as individual
onevoucher = data.frame(individual=1,biocollection="INPA",biocollection_number=1234,biocollection_type=1,dataset=1)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
odb_import_vouchers(onevoucher, odb_cfg=cfg)
#get the imported voucher
voucher = odb_get_vouchers(params=list(individual=1),cfg)
vouchers[,c("basisOfRecord","scientificName","occurrenceID","collectionCode","catalogNumber") ]
Voucher diferente para um indivíduo
Dois vouchers para o mesmo Indivíduo, um com o mesmo coletor e data de coleta do Indivíduo, outro em data diferente e por outros cobradores.
#one with same date and collector as individual
one = data.frame(individual=2,biocollection="INPA",biocollection_number=1234,dataset=1,collector=NA,number=NA,date=NA)
#this one with different collector and date
two= data.frame(individual=2,biocollection="INPA",biocollection_number=4435,dataset=1,collector="Oliveira, A.A. de|João Batista da Silva",number=3456,date="1991-08-01")
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
to.odb = rbind(one,two)
odb_import_vouchers(to.odb, odb_cfg=cfg)
#get the imported voucher
voucher = odb_get_vouchers(params=list(individual=2),cfg)
voucher[,c("basisOfRecord","scientificName","occurrenceID","collectionCode","catalogNumber") ]
Registros importados:
basisOfRecord scientificName occurrenceID collectionCode catalogNumber recordedByMain
1 PreservedSpecimens Unidentified plot tree - Vicentini -INPA.1234 INPA 1234 Vicentini, A.
2 PreservedSpecimens Unidentified 3456 - Oliveira -INPA.4435 INPA 4435 Oliveira, A.A. de
2.6 - Importar Medições
Atenção
- A medição tem uma restrição: valores ou categorias de medição duplicados para o mesmo object_id e mesma data não serão importados, a menos que você especifique
duplicated = 1para o registro. - Para importar medições você precisa coletar algumas informações de outros modelos. Você precisa fornecer um
datasetpara o qual você tem permissões,trait_id(export_name ou id),value(o conteúdo varia dependendo do tipo de variável),date(uma data completa ou incompleta),person(pessoa que mediu),object_type(uma de taxon, individual, voucher ou location) eobject_id(o id do object_type). Para o tipo de variável LINK você deve fornecer uma colunalink_id, e ‘value’ torna-se opcional para este caso específico. - DICA: Para relacionar medições de diferentes variáveis, você pode adicionar palavaras chave campo de medição
notes. Por exemplo, você mede LeafLength e LeafWidth para as mesmas folhas, portanto, em ambas as medições, adicione “leaf1”, “leaf2” ao campo de nota para vinculá-los. Além disso, se você medir o DAP e o DAP.POM em um inventário de parcela florestal e se tiver vários troncos para uma planta, você pode adicionar “caule1”, “caule2” para permitir a correspondência de um DAP do caule com sua altura específica de medição. Esse tipo de link pode ser útil em análises. - Várias validações acontecerão durante a importação da medição. Considere verificar os dados localmente antes de enviar um trabalho (job) para facilitar a solução de problemas e garantir que os valores sejam consistentes com suas expectativas. Você não poderá ATUALIZAR nem EXCLUIR medidas com a API, portanto, evite importar medições que precisarão ser corrigidas posteriormente (uma por uma).
- Para características quantitativas, OpenDataBio não pode validar se suas medições estão na mesma unidade que a definição de variável. Portanto, verifique qual unidade a variável possui e converta seus dados de acordo antes de enviar.
As medições podem ser importadas usando odb_import_measurements(). Leia atentamente o Measurements POST API.
Variáveis Quantitativas
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#obter o id do trait do servidor (verifique se o trait existe)
#gerar alguns dados falsos para 10 medições
dbhs = sample(seq(10,100,by=0.1),10)
object_ids = sample(1:3,length(dbhs),replace=T)
dates = sample(as.Date("2000-01-01"):as.Date("2000-03-31"),length(dbhs))
dates = lapply(dates,as.Date,origin="1970-01-01")
dates = lapply(dates,as.character)
dates = unlist(dates)
to.odb = data.frame(
trait_id = 'dbh',
value = dbhs,
date = dates,
object_type = 'Individual',
object_id=object_ids,
person="Oliveira, A.A. de",
dataset = 1,
notes = "some fake measurements",
stringsAsFactors=F)
#isso só funcionará se a pessoa existir, os ids individuais existirem
#e se a característica com export_name = dbh existe
odb_import_measurements(to.odb,odb_cfg=cfg)
Baixar os dados importados:
dad = odb_get_measurements(params = list(dataset=1),odb_cfg=cfg)
dad[,c("id","basisOfRecord", "measured_type", "measured_id", "measurementType",
"measurementValue", "measurementUnit", "measurementDeterminedDate",
"datasetName", "license")]
id basisOfRecord measured_type measured_id measurementType measurementValue measurementUnit measurementDeterminedDate
1 1 MeasurementsOrFact App\\Models\\Individual 3 dbh 86.8 centimeters 2000-02-19
2 2 MeasurementsOrFact App\\Models\\Individual 2 dbh 84.8 centimeters 2000-03-25
3 3 MeasurementsOrFact App\\Models\\Individual 2 dbh 65.7 centimeters 2000-03-15
4 4 MeasurementsOrFact App\\Models\\Individual 3 dbh 88.0 centimeters 2000-03-05
5 5 MeasurementsOrFact App\\Models\\Individual 3 dbh 35.3 centimeters 2000-01-04
6 6 MeasurementsOrFact App\\Models\\Individual 2 dbh 36.0 centimeters 2000-03-23
7 7 MeasurementsOrFact App\\Models\\Individual 2 dbh 78.6 centimeters 2000-03-22
8 8 MeasurementsOrFact App\\Models\\Individual 2 dbh 69.7 centimeters 2000-03-09
9 9 MeasurementsOrFact App\\Models\\Individual 3 dbh 12.3 centimeters 2000-01-30
10 10 MeasurementsOrFact App\\Models\\Individual 3 dbh 14.7 centimeters 2000-01-18
datasetName license
1 Dataset test CC-BY 4.0
2 Dataset test CC-BY 4.0
3 Dataset test CC-BY 4.0
4 Dataset test CC-BY 4.0
5 Dataset test CC-BY 4.0
6 Dataset test CC-BY 4.0
7 Dataset test CC-BY 4.0
8 Dataset test CC-BY 4.0
9 Dataset test CC-BY 4.0
10 Dataset test CC-BY 4.0
Medidas categóricas
As categorias DEVEM ser informadas por seus ids ou nome no campo value. Para características CATEGORICAL ou ORDINAL, value deve ser um valor único. Para CATEGORICAL_MULTIPLE, value pode ser um ou vários ids de categorias ou nomes separados por um de | ou ; ou,.
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#a categorical trait
(odbtraits = odb_get_traits(params=list(name="specimenFertility"),odb_cfg = cfg))
#base line
to.odb = data.frame(trait_id = odbtraits$id, date = '2021-07-31', stringsAsFactors=F)
#the plant was collected with both flowers and fruits, so the value are the two categories
value = c("Flowers","Fruits")
#get categories for this trait if found
(cats = odbtraits$categories[[1]])
#check that your categories are registered for the trait and get their ids
value = cats[match(value,cats$name),'id']
#make multiple categories ids a string value
value = paste(value,collapse=",")
to.odb$value = value
#this links to a voucher
to.odb$object_type = "Voucher"
#get voucher id from API (must be ID).
#Search for collection number 1234
odbspecs = odb_get_vouchers(params=list(number="3456-A"),odb_cfg=cfg)
to.odb$object_id = odbspecs$id[1]
#get dataset id
odbdatasets = odb_get_datasets(params=list(name='Dataset test'),odb_cfg=cfg)
head(odbdatasets)
to.odb$dataset = odbdatasets$id
#person that measured
odbperson = odb_get_persons(params=list(search='ana cristina sega'),odb_cfg=cfg)
to.odb$person = odbperson$id
#import'
odb_import_measurements(to.odb,odb_cfg=cfg)
#get imported
dad = odb_get_measurements(params = list(voucher=odbspecs$id[1]),odb_cfg=cfg)
dad[,c("id","basisOfRecord", "measured_type", "measured_id", "measurementType",
"measurementValue", "measurementUnit", "measurementDeterminedDate",
"datasetName", "license")]
id basisOfRecord measured_type measured_id measurementType measurementValue measurementUnit
1 11 MeasurementsOrFact App\\Models\\Voucher 1 specimenFertility Flowers, Fruits NA
measurementDeterminedDate datasetName license
1 2021-07-31 Dataset test CC-BY 4.0
Cores
Para valores de cor, você deve inserir cor como seus códigos de strings RGB hexadecimal, para que possam ser renderizados graficamente e na interface da web. Portanto, qualquer valor de cor é permitido e seria mais fácil usar as cores da paleta na interface da web para inserir tais medidas. O pacote gplots permite que você converta nomes de cores em códigos RGB hexadecimais se você quiser fazer isso por meio da API.
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#get the trait id from the server (check that trait exists)
odbtraits = odb_get_traits(odb_cfg=cfg)
(m = match(c("fruitColor"),odbtraits$export_name))
#base line
to.odb = data.frame(trait_id = odbtraits$id[m], date = '2014-01-13', stringsAsFactors=F)
#get color value
#install.packages("gplots",dependencies = T)
library(gplots)
(value = col2hex("red"))
to.odb$value = value
#this links to a specimen
to.odb$object_type = "Individual"
#get voucher id from API (must be ID). Search for collection number 1234
odbind = odb_get_individuals(params=list(tag='3456'),odb_cfg=cfg)
odbind$scientificName
to.odb$object_id = odbind$id[1]
#get dataset id
odbdatasets = odb_get_datasets(params=list(name='Dataset test'),odb_cfg=cfg)
head(odbdatasets)
to.odb$dataset = odbdatasets$id
#person that measured
odbperson = odb_get_persons(params=list(search='ana cristina sega'),odb_cfg=cfg)
to.odb$person = odbperson$id
odb_import_measurements(to.odb,odb_cfg=cfg)

Medições de tipo de LINK de banco de dados
O tipo de variável LINK permite registrar dados de contagem, como por exemplo o número de indivíduos de uma espécie em um determinado local. Você deve fornecer o objeto vinculado (link_id), que pode ser um Táxon ou um Voucher, dependendo da definição da variável, e então o value recebe a contagem numérica.
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#get the trait id from the server (check that trait exists)
odbtraits = odb_get_traits(odb_cfg=cfg)
(m = match(c("taxonCount"),odbtraits$export_name))
#base line
to.odb = data.frame(trait_id = odbtraits$id[m], date = '2014-01-13', stringsAsFactors=F)
#the taxon to link the count value
odbtax = odb_get_taxons(params=list(name='Ocotea guianensis'),odb_cfg=cfg)
to.odb$link_id = odbtax$id
#now add the count value for this trait type
#this is optional for this measurement,
#however, it would make no sense to include such link without a count in this example
to.odb$value = 23
#a note to clarify the measurement (optional)
to.odb$notes = 'No voucher, field identification'
#this measurement will link to a location
to.odb$object_type = "Location"
#get location id from API (must be ID).
#lets add this to a transect
odblocs = odb_get_locations(params=list(adm_level=101,limit=1),odb_cfg=cfg)
to.odb$object_id = odblocs$id
#get dataset id
odbdatasets = odb_get_datasets(params=list(name='Dataset test'),odb_cfg=cfg)
head(odbdatasets)
to.odb$dataset = odbdatasets$id
#person that measured
odbperson = odb_get_persons(params=list(search='ana cristina sega'),odb_cfg=cfg)
to.odb$person = odbperson$id
odb_import_measurements(to.odb,odb_cfg=cfg)

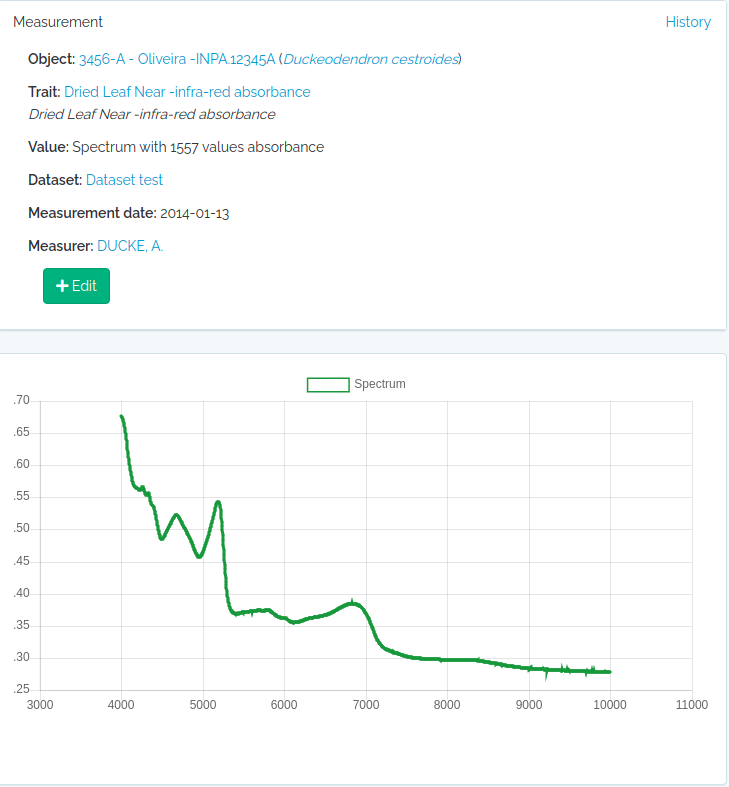
Medições espectrais
value deve ser uma string de valores do espectro separados por “;”. O número de valores concatenados deve corresponder ao atributo value_length da variável, que é extraído da especificação do intervalo de número de onda para a variável. Portanto, você pode verificar isso facilmente antes de importar com odb_get_traits(params=list (fields = 'all', type = 9),cfg)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#read a spectrum
spectrum = read.table("1_Sample_Planta-216736_TAG-924-1103-1_folha-1_abaxial_1.csv",sep=",")
#second column are NIR leaf absorbance values
#the spectrum has 1557 values
nrow(spectrum)
#[1] 1557
#collapse to single string
value = paste(spectrum[,2],collapse = ";")
substr(value,1,100)
#[1] "0.6768057;0.6763237;0.6755353;0.6746023;0.6733549;0.6718447;0.6701176;0.6682984;0.6662288;0.6636459;"
#get the trait id from the server (check that trait exists)
odbtraits = odb_get_traits(odb_cfg=cfg)
(m = match(c("driedLeafNirAbsorbance"),odbtraits$export_name))
#see the trait
odbtraits[m,c("export_name", "unit", "range_min", "range_max", "value_length")]
#export_name unit range_min range_max value_length
#6 driedLeafNirAbsorbance absorbance 3999.64 10001.03 1557
#must be true
odbtraits$value_length[m]==nrow(spectrum)
#[1] TRUE
#base line
to.odb = data.frame(trait_id = odbtraits$id[m], value=value, date = '2014-01-13', stringsAsFactors=F)
#this links to a voucher
to.odb$object_type = "Voucher"
#get voucher id from API (must be ID).
#search for a collection number
odbspecs = odb_get_vouchers(params=list(number="3456-A"),odb_cfg=cfg)
to.odb$object_id = odbspecs$id[1]
#get dataset id
odbdatasets = odb_get_datasets(params=list(name='Dataset test'),odb_cfg=cfg)
to.odb$dataset = odbdatasets$id
#person that measured
odbperson = odb_get_persons(params=list(search='adolpho ducke'),odb_cfg=cfg)
to.odb$person = odbperson$id
#import
odb_import_measurements(to.odb,odb_cfg=cfg)
Texto e notas
Basta adicionar o texto ao campo value e proceder como para os outros tipos de variável.