This the multi-page printable view of this section. Click here to print.
Documentação
- 1: Visão geral
- 2: Primeiros passos
- 2.1: Primeira vez?
- 2.2: Instalação padrão
- 2.3: Instalação com Docker
- 2.4: Personalizar a instalação
- 3: Serviços de API
- 3.1: Referência rápida
- 3.2: Obter dados - GET
- 3.3: Inserir dados - POST
- 3.4: Atualizar dados - PUT
- 4: Modelo conceitual
- 4.1: Objetos Centrais
- 4.2: Objetos de Atributos
- 4.3: Objetos de Acesso
- 4.4: Objetos Auxiliares
- 5: Como contribuir
- 6: Tutoriais
- 6.1: Obter dados via R
- 6.2: Importar dados via R
- 6.2.1: Importar Localidades
- 6.2.2: Importar Taxons
- 6.2.3: Importar Pessoas
- 6.2.4: Importar Variáveis
- 6.2.5: Importar Indivíduos & Vouchers
- 6.2.6: Importar Medições
1 - Visão geral
OpenDataBio é uma plataforma-web de código aberto projetada para ajudar pesquisadores e organizações que estudam a biodiversidade em regiões tropicais a coletar, armazenar, relacionar e servir dados. Ele é projetado para acomodar muitos tipos de dados usados em ciências biológicas e suas relações, particularmente para estudos de ecologia e biodiversidade e serve como um repositório de dados que permite a usuários baixar ou solicitar dados de pesquisa bem organizados e documentados.
Porque?
Estudos de biodiversidade freqüentemente requerem a integração de uma grande quantidade de dados, exigindo padronização para uso e compartilhamento. Pesquisadores também precisam gerenciar e manter os seus dados de forma contínua e para além de depositá-los de forma estática em repositórios públicos ou data papers.
O OpenDataBio foi desenhado com base na necessidade de organizar e integrar dados históricos e atuais coletados na região amazônica, levando em consideração as práticas de campo e os tipos de dados utilizados por ecologistas e taxonomistas.
O OpenDataBio visa facilitar a padronização e normalização dos dados, utilizando diferentes serviços disponíveis online, dando flexibilidade aos usuários e grupos de usuários, e criando os links necessários entre Localidades, Táxons, Indivíduos, Vouchers e as Medições e os Arquivos de Mídia associados a eles, ao mesmo tempo em que oferece acessibilidade aos dados por meio de um serviço de API próprio, facilitando a distribuição e análise dos dados.
Características relevantes
- Variáveis personalizadas - flexibilidade para o usuário definir variáveis incluindo alguns casos especiais como variáveis Espectrais, Cores, Links e GeneBank. Essa variávies são compartilhadas entre usários e requerem definições extadas como metadados. Medições para tais características podem ser registradas para Indivíduos, Vouchers, Taxons ou Localidades.
- Taxons podem ser nomes publicados ou não publicados (por exemplo, um morfotipo), sinônimos ou nomes válidos e qualquer nó da árvore da vida pode ser armazenado. A inserção de táxons é verificada em diferentes bases nomenclaturais (Tropicos, IPNI, MycoBank, ZOOBANK, GBIF), minimizando sua busca por erros ortográficos, autores, e validação de sinônimos
- Localidades são armazenados com suas geometrias espaciais, permitindo consultas espaciais. Localidades como Parcelas e Transectos podem ser definidos, facilitando a organização de dados de métodos comumente usados em estudos de biodiversidade
- Controle de acesso aos dados - os dados são organizados em Conjuntos de dados que permite definir uma política de acesso (público, não público) e uma licença para distribuição de conjuntos de dados públicos, tornando-se uma publicação de dados dinâmicos, cuja versão é a data da última edição.
- Diferentes grupos de pesquisa podem usar uma única instalação OpenDataBio, tendo total controle sobre a edição e acesso de seus dados de pesquisa particulares, enquanto compartilham bibliotecas comuns como Taxonomia, Localidades, Referências bibliográficas e Váriáveis.
- API para acessar dados programaticamente - Ferramentas para exportação e importação de dados são fornecidas por meio de serviços de API junto com um cliente API na linguagem R, o pacote OpenDataBio-R.
- Auditoria - o modelo de atividade audita alterações em qualquer registro e downloads de conjuntos de dados completos, que são registrados para rastreamento.
- O modelo BioColeção permite que uma Coleções Biológica gerencie seus registros de Vouchers e solicitações realizados por usuários, facilitando a interação com os usuários que fornecem amostras e dados;
- Um coletor de dados móveis está planejado com ODK ou ODK-X
Saiba mais
- Primeiros passos: instale o OpenDataBio
2 - Primeiros passos
OpenDataBio é um web-software para Linux nas distribuições Debian, Ubuntu e Arch-Linux e pode ser implementado em qualquer máquina baseada em Linux. Não temos planos de suporte ao Windows, mas pode ser fácil de instalar em uma máquina Windows usando o Docker.
Opendatabio é escrito em PHP e desenvolvido com o framework Laravel. Requer um servidor web (apache ou nginx), PHP e um banco de dados SQL - testado apenas com MySQL e MariaDB .
Você pode instalar o OpenDataBio facilmente usando os arquivos Docker incluídos na distribuição, mas esses arquivos docker fornecidos destinam-se apenas ao desenvolvimento e precisam de ajuste para implementar um site em produção.
Se você deseja testar o OpenDataBio, ajudar no desenvolvimento ou ou ter uma instalação individual no seu computador, siga a instalação do Docker.
Prepare para instalação
- Você pode solicitar uma chave API Tropicos.org para que o OpenDataBio possa recuperar dados taxonômicos do banco de dados Tropicos.org. Se não for fornecido, principalmente o serviço de nomenclatura do GBIF será usado;
- OpenDataBio envia e-mails para usuários registrados, seja para informar sobre um job que foi concluído, para enviar solicitações de dados para administradores de Conjuntos de Dados, ou para recuperação de senha. Você pode usar um e-mail do Google para isso, mas precisará alterar as opções de segurança da conta para permitir que o OpenDataBio use a conta para enviar e-mails (você precisa ativar a opção de
Acesso a aplicativos menos segurosnas configurações da conta do gmail). Portanto, crie um endereço de e-mail dedicado para sua instalação. Verifique o arquivo “config/mail.php” para mais opções sobre como enviar e-mails.
2.1 - Primeira vez?
OpenDataBio é um software para ser usado online. As instalações locais são para teste ou desenvolvimento, embora possam ser usadas para como ambiente local de produção de um único usuário.
Tipos de usuário
Atenção
Se você se registrou, alguém precisa atribuir você como usuário completo para que você possa inserir dados.- Se você estiver instalando, o primeiro login para uma instalação do OpenDataBio deve ser feito com o usuário super-administrador padrão:
admin@example.orgepassword1. Essas configurações devem ser alteradas, senão a instalação fica aberta à qualquer pessoa lendo essas instruções. - Quando um usuário se auto-cadastra numa instalação do OpenDataBio, ele não tem permissão de edição ou inserção de dados no banco de dados, ele apenas ganha um cadastro e acesso aos conjuntos de dados que são abertos apenas para
usuários registradose permite que o usuário faça downloads pela interface, os quais exigem login. - Apenas usuários completos podem contribuir com dados
- E apenas super-administradores podem atribuir o papel de
usuário completopara usuários registrados - diferentes instalações do OpenDataBio podem ter políticas diferentes sobre como você pode obter acesso de usuário completo.
Ver mais em Usuários.
Prepare seu perfil de usuário-completo
- Criar uma Pessoa com os seus dados e ligar ela ao seu perfil de usuário através das configurações. Assim, os formulários da interface utilizarão essa Pessoa como padrão, facilitando a entrada de dados.
- Você precisará de pelo menos 1 conjunto de dados para entrar seus dados
- isso não é necessário para importar dados de biblitecas compartilhadas, como Pessoas, Localidades, Taxons e Referências Bibliográficas (saiba mais aqui).
- quando receber atribuição de usuário-completo, um Conjunto de Dados de acesso restrito e Projeto serão automaticamente criados para você com o nome de Workspace SEU-NOME. Você pode modificar as configurações desses objetos da forma que desejar.
- Você poderá criar tantos projetos e conjuntos de dados que precisar. Entenda bem esses Objetos de Controle de Acesso do OpenDataBio.
Entrando dados
Existem três maneiras principais de importar dados para o OpenDataBio:
- Um por um por meio da interface web, usando um navegador.
- Usando os [serviços OpenDataBio de POST API] (/docs/api):
- importar de um arquivo CSV, XLXS ou ODS usando a interface (Menu Importar)
- através do R, usando OpenDataBio R package, que um cliente para essa API.
- criando o seu próprio cliente em outra linguagem
- Ao usar os [serviços da API OpenDataBio] (/docs/api), você deve preparar seus dados ou arquivo para importação de acordo com as opções de campo do [verbo POST] (/docs/post-data) para o ’endpoint’ específico que você está tentando importar. Verifique também a referência rápida da API.
R Tutorials
Veja o tutorial Importar dados via R para exemplos de como usar o pacote do R (e API) para importar dados.Dicas para inserir dados
- Se for inserir dados pela primeira vez, recomendamos que você utilize a interface pelo seu navegador e crie pelo menos um registro para cada objeto que atenda às suas necessidades. Em seguida, experimente as configurações de privacidade do seu espaço de trabalho conjunto de dados e verifique se você pode acessar os dados quando estiver conectado ou não. Ou seja, entenda como os dados ficam organizados e veja como obtê-los.
- Use Conjunto de dados para um conjunto independente de dados que deve ser distribuído como um grupo. Os conjuntos de dados são publicações dinâmicas, têm autor, dados e título.
- Embora o ODB tente minimizar a redundância, dar flexibilidade aos usuários tem um custo em algumas definições, como por exemplo, Variáveis e [Pessoas](/docs/concepts/objetos auxiliares/#person) que podem receber registros duplicados. Portanto, deve-se ter cuidado ao criar tais registros. Os administradores podem criar um código de conduta para os usuários de uma instalação ODB para minimizar tal redundância.
- Siga uma ordem de importação de novos dados, a partir das bibliotecas de uso comum. Por exemplo, você deve primeiro registrar Localidades, Taxons, Pessoas, Variáveis e qualquer outra biblioteca comum antes de importar Indivíduos ou Medições
- OpenDataBio pode ser instalado com dados previamente organizados para Localidades do Brasil [unidades administrativas (Município, Estado, País), Unidades de Conservação federais e Terras Indígenas] e uma base da árvore da vida para Taxons complementada pela filogenia das plantas com sementes até o nível de Ordem conforme a árvore do Angiosperm Phylogeny WebSite, version 14.
- Não há nenhuma necessidade de importar localidades do tipo
POINTpara importar Indivíduos porque o ODB cria a localidade para você se informar latitude e longitude, e detectará para você a qual são as localidades registradas à qual o indivíduo pertence. Ou seja, ODB detecta as unidades administrativas (Município, Estados, Pais), as Unidades de Conservação e Terras Indígenas e também classes ambientais, dependendo das Localidades registradas na base de dados. Você pode usar a API de localidades com o parâmetroquerytypepara validar previamente as coordenadas dos seus indivíduos. - Existem diferentes maneiras de criar localidades do tipo PARCELA e TRANSECTOS - saiba mais em Localidades.
- A criação de taxons exige apenas a especificação de um
nome- ODB pesquisará os serviços de nomenclatura para você, encontrará o nome, metadados e taxons pais ou taxons aceitos, se o seu nome for um sinônimo, e importará todos os eles, se necessário até encontrar na base algum taxon na hierarquia ancestral. Por exemplo, se informar Ocotea guianensis, mas apenas Laurales já está cadastrado na base, ODB irá buscar e cadastrar para você todo o caminhoLauraceae >> Ocotea >> Ocotea guianensis, com autoria, referencia bibliográfica e link ao repositório nomenclatural onde encontrou os dados. Se você estiver importando nomes publicados, basta informar este único atributo. Caso contrário, se o nome não for publicado, é necessário informar campos adicionais. Portanto, separe a importação em lote de nomes publicados e não publicados em dois conjuntos. - O campo
notesde qualquer modelo é para texto simples ou para dados formatados como objectos JSON. A opção Json permite que você armazene dados estruturados personalizados em qualquer modelo que tenha o camponotes. Você pode, por exemplo, armazenar como notas alguns campos secundários de fontes originais ao importar dados, mas pode armazenar quaisquer dados adicionais que não sejam fornecidos pela estrutura do banco de dados do OpenDataBio. Esses dados não serão validados pelo OpenDataBio e a padronização de tags e valores depende de você. As notas Json serão importadas e exportadas como um texto JSON e serão apresentadas na interface como uma tabela formatada; URLs em seu Json serão apresentados como links.
2.2 - Instalação padrão
Estas instruções são para uma instalação baseada em apache, mas podem ser facilmente ajustadas para funcionar com nginx.
Requisitos do servidor
- A versão mínima do PHP é 8.0
- O servidor da web pode ser apache ou nginx. Para nginx, verifique a configuração nos arquivos do docker para ajustar essas instruções, que são para o apache.
- Requer um banco de dados SQL, MySQL e MariaDB foram testados, mas também pode funcionar com Postgres. Testado com MYSQL.v8 e MariaDB.v15.1.
- Extensões PHP necessárias ‘openssl’, ‘pdo’, ‘pdo_mysql’, ‘mbstring’, ’tokenizer’, ‘xlm’, ‘dom’, ‘gd’, ’exif’, ‘bcmath’
- Pandoc é usado para traduzir o código LaTeX usado nas referências bibliográficas. Não é necessário para a instalação, mas é sugerido para uma melhor experiência do usuário.
- Requer Supervisor, que é necessário para os jobs de usuário
Criar usuário dedicado
A maneira recomendada de instalar o OpenDataBio para produção é usando um usuário de sistema dedicado. Nestas instruções, esse usuário é odbserver.
Baixar OpenDataBio
Faça login como seu Usuário dedicado e baixe ou clone este software para onde deseja instalá-lo.
Aqui assumimos que é /home/odbserver/opendatabio para que os arquivos de instalação residam neste diretório. Se este não for o seu caminho, altere abaixo sempre que aplicável.
Baixar OpenDataBio
Prepare o Servidor
Primeiro, instale os softwares Apache, MySQL, PHP, Pandoc e Supervisor. Em um sistema Debian, você também precisa instalar algumas extensões PHP e ativá-las:
#EXEMPLO EM UBUNTU 22.04
#repositórios
apt-get install software-properties-common
add-apt-repository ppa:ondrej/php
add-apt-repository ppa:ondrej/php ppa:ondrej/apache2
add-apt-repository ppa:ondrej/php
add-apt-repository ppa:ondrej/apache2
apt update
#instala o php
apt install php8.2 -y
apt update
apt upgrade
#instala extensoes (modulos) do php
php --version
#quais os modulos instalados?
php -m
#se algum desses nao estive, instale
apt install php8.2-{bcmath,xml,mysql,zip,intl,gd,cli,curl,mbstring,sqlite3}
#install apache
apt install libapache2-mod-php8.2
#install pandoc
apt install pandoc
#install supervisor (needed for jobs)
apt-get install supervisor -y
a2enmod php8.2
phpenmod mbstring
phpenmod xml
phpenmod dom
phpenmod gd
a2enmod rewrite
a2ensite
systemctl restart apache2.service
#To check if they are installed:
php -m | grep -E 'mbstring|cli|xml|gd|mysql|pandoc|supervisord|bcmath|pcntl|zip'
Adicione o seguinte à sua configuração do Apache.
- Mude
/home/odbserver/opendatabiopara o seu caminho (os arquivos devem estar acessíveis pelo apache) - Você pode criar um novo arquivo na pasta sites-available:
/etc/apache2/sites-available/opendatabio.confe colocar o seguinte código nele.
<IfModule alias_module>
Alias /opendatabio /home/odbserver/opendatabio/public/
Alias /fonts /home/odbserver/opendatabio/public/fonts
Alias /images /home/odbserver/opendatabio/public/images
<Directory "/home/odbserver/opendatabio/public">
Require all granted
AllowOverride All
</Directory>
</IfModule>
Isso fará com que o Apache redirecione todas as solicitações de / para a pasta correta, e também permitirá que o arquivo .htaccess fornecido controle as regras de reescrita, de forma que os URLs sejam bonitos. Se desejar acessar o arquivo apontando o navegador para a raiz do servidor, adicione também a seguinte diretiva:
RedirectMatch ^/$ /
Configure seu arquivo php.ini. O instalador pode reclamar da falta de extensões do PHP, então lembre-se de ativá-las nos arquivos cli (/etc/php/8.0/cli/php.ini e web ini (/etc/php/8.0/fpm/php.ini) para PHP!
Atualize os valores para as seguintes variáveis:
Encontre os arquivos
php -i | grep 'Configuration File'
Mudar:
memory_limit should be at least 512M
post_max_size should be at least 30M
upload_max_filesize should be at least 30M
Algo como:
[PHP]
allow_url_fopen=1
memory_limit = 512M
post_max_size = 100M
upload_max_filesize = 100M
Habilite os módulos Apache ‘mod_rewrite’ e ‘mod_alias’ e reinicie o servidor:
sudo a2enmod rewrite
sudo a2ensite
sudo systemctl restart apache2.service
Mysql Charset e Collation
- Você deve adicionar o seguinte ao seu arquivo de configuração do SQL (mariadb.cnf ou my.cnf), ou seja, o conjunto de caracteres e o agrupamento que você escolher para sua instalação devem corresponder aos do
config/database.php
[mysqld]
character-set-client-handshake = FALSE #without this, there is no effect of the init_connect
collation-server = utf8mb4_unicode_ci
init-connect = "SET NAMES utf8mb4 COLLATE utf8mb4_unicode_ci"
character-set-server = utf8mb4
log-bin-trust-function-creators = 1
sort_buffer_size = 4294967295 #this is needed for geometry (bug in mysql:8)
[mariadb]
max_allowed_packet=100M
innodb_log_file_size=300M #no use for mysql
- Se estiver usando MariaDB e você ainda tiver problemas do tipo #1267 Illegal mix of collations, então verifique aqui sobre como consertar isso.
Configurar o supervisord
Configure o Supervisor, que é necessário para trabalhos. Crie um nome de arquivo opendatabio-worker.conf na pasta de configuração do Supervisor /etc/supervisor/ conf.d/opendatabio-worker.conf com o seguinte conteúdo, ajustando o caminho conforme a sua instalação:
touch /etc/supervisor/conf.d/opendatabio-worker.conf
echo ";--------------
[program:opendatabio-worker]
process_name=%(program_name)s_%(process_num)02d
command=php /home/odbserver/opendatabio/artisan queue:work --sleep=3 --tries=1 --timeout=0 --memory=512
autostart=true
autorestart=true
user=odbserver
numprocs=8
redirect_stderr=true
stdout_logfile=/home/odbserver/opendatabio/storage/logs/supervisor.log
;--------------" > /etc/supervisor/conf.d/opendatabio-worker.conf
Permissões de arquivos e pastas
Segurança
As permissões de pasta e arquivo são importantes para proteger a instalação em um servidor aberto publicamente. Se você não configurar corretamente, seu site poderá estar em risco.- As pastas
storageebootstrap/cacheprecisam ter permissão de escrita para o usuário do servidor (geralmente www-data). Defina0775para esses diretórios. - O arquivo de configuração
.envprecisa ter permissão0640pois contém senhas. - Este link mostra diferentes métodos de definir permissões para um aplicativo Laravel.
Este é o método recomendado:
cd /home/odbserver
#note que odbserver e www-data podem mudar na sua configuracao
#defia as permissões tanto par ao seu usuário (aqui odbserver) como para o do apache (aqui www-data)
sudo chown -R odbserver:www-data opendatabio
sudo find ./opendatabio -type f -exec chmod 644 {} \;
sudo find ./opendatabio -type d -exec chmod 755 {} \;
cd /home/odbserver/opendatabio
sudo chgrp -R www-data storage bootstrap/cache
sudo chmod -R ug+rwx storage bootstrap/cache
#arquivo de configuracao .env para permissao 640
sudo chmod 640 ./.env
Instale OpenDataBio
-
Muitas distribuições Linux (Ubuntu e Debian) têm arquivos php.ini diferentes para a interface de linha de comando e para o Apache. Recomenda-se usar o arquivo de configuração do Apache ao executar o script de instalação, para que ele possa apontar corretamente as extensões ou configurações ausentes. Para fazer isso, encontre o caminho correto para o arquivo .ini e exporte-o **antes de usar o comando de instalação
php install**.Por exemplo,
export PHPRC=/etc/php/8.0/fpm/php.ini -
O script de instalação baixará o gerenciador de dependências Composer e todas as bibliotecas PHP necessárias listadas no arquivo
composer.json. No entanto, se o seu servidor estiver atrás de um proxy, você deve instalar e configurar o Composer independentemente. Implementamos a configuração do PROXY, mas não a estamos mais usando e não testamos corretamente (se você precisar de ajustes, coloque um issue no GitLab). -
O script solicitará opções de configuração, que são armazenadas no arquivo de ambiente
.envna pasta raiz do aplicativo.Você pode, opcionalmente, configurar este arquivo antes de executar o instalador:
- Crie um arquivo
.envcom o conteúdo docp .env.example .envfornecido - Leia os comentários neste arquivo e ajuste de acordo
- Crie um arquivo
-
Execute o instalador:
cd /home/odbserver/opendatabio
php install
- Seed data - o script irá pergunar se você quer instalar Localidades e Taxons distribuídos com aplicativo. Esses dados são específicos de cada versão do OpenDataBio. Ver as notas das versões no repositório desses dados.
Pronto para usar
Se o script de instalação for concluído com sucesso, você está pronto para prosseguir! Aponte seu navegador parahttp://localhost/opendatabio. As migrações de banco de dados incluem uma conta de administrador, com login admin@example.org e senha password1. Altere a senha após a instalação.
Problemas de instalação
Existem inúmeras maneiras possíveis de instalar o aplicativo, mas podem envolver mais etapas e configurações.
- se o navegador retornar 500|SERVER ERROR , você deve olhar para o último error em
storage/logs/laravel.log. Se você tiver ERROR: No application encryption key has been specified execute:
chave artesanal php: gerar
php artisan config: cache
- Se você receber o erro failed to open stream: Connection timed out durante a execução do instalador, isso indica uma configuração incorreta do seu roteamento IPv6. A correção mais fácil é desabilitar o roteamento IPv6 no servidor.
- Se você receber erros durante alimentação aleatória do banco de dados, você pode tentar remover o banco de dados inteiramente e reconstruí-lo. Claro, não execute isso em uma instalação de produção.
php artisan migrate: fresh
- Você pode substituir as tabelas Locations e Taxons usando o seed data depois de reconstruir a base:
php seedodb
Configurações pós-instalação
- Se seus Jobs de importação/exportação não estão sendo processados, certifique-se de que o Supervisor esteja executando
systemctl start supervisord && systemctl enable supervisorde verifique os arquivos de log emstorage/logs/supervisor.log. - Você pode alterar várias variáveis de configuração para o aplicativo. O mais importante deles provavelmente está definido pelo instalador, mas há outras variáveis em
.enve no arquivoconfig/app.phpque você pode alterar. Em particular, você pode querer alterar as configurações de idioma, fuso horário e e-mail. Executephp artisan config: cacheapós atualizar os arquivos de configuração. - Para impedir que os rastreadores do mecanismo de pesquisa indexem seu banco de dados, adicione o seguinte ao seu “robots.txt” na pasta raiz do servidor (no Debian, /var/www/html):
User-agent: *
Disallow: /
- As pastas
storageebootstrap/cachedevem ser graváveis pelo usuário do Apache (geralmente www-data). Veja este link para um exemplo de como fazer isso. Defina a permissão0775para esses diretórios.
Armazenamento e backups
Você pode alterar as configurações de armazenamento em config/filesystem.php, onde pode definir o armazenamento baseado em nuvem, que pode ser necessário se muitos usuários enviarem arquivos de mídia, exigindo muito espaço em disco.
- Downloads de dados são colocados em fila como Jobs e um arquivo é gravado em uma pasta temporária,sendo excluído quando o trabalho é excluído pelo usuário. Esta pasta é definida como
download diskno arquivo de configuração filesystem.php, que aponta parastorage/app/public/downloads. Apagar esses arquivos temporários depende dos usuários, portanto, um trabalho de limpeza do cron pode ser aconselhável para implementar em sua instalação; - Arquivos de mídia são armazenados por padrão no
media disk, que coloca os arquivos na pastastorage/app/ public/media; - Para configuração regular crie ambos os diretórios
storage/app/public/downloadsestorage/app/public/mediacom permissões graváveis pelo usuário do servidor - Lembre-se de incluir a pasta de mídia em um plano de backup;
2.3 - Instalação com Docker
A maneira mais fácil de instalar e executar o OpenDataBio é usando o Docker e os arquivos de configuração do docker fornecidos, que contêm todas as configurações necessárias para executar o ODB. Usa nginx e mysql e supervisor.
Arquivos Docker incluídos
laraverl-app/
----docker/*
----./env.docker
----docker-compose.yml
----Dockerfile
----Makefile
Eles foram adaptados deste link, onde você também encontra uma configuração de produção.
Instalação
Baixar OpenDataBio
- Certifique-se de ter Docker e Docker-compose instalados em seu sistema operacional.
- Verifique se o seu usuário está no grupo docker criado durante a instalação do docker;
- Baixe ou clone o OpenDataBio em sua máquina;
- Certifique-se de que o seu usuário é o proprietário da pasta e do conteúdo criados, caso contrário, altere o proprietário e o grupo recursivamente para o seu usuário
- Entre no diretório criado
opendatabio - Opcionalmente, pode editar e ajustar o nome do arquivo contendo as configurações de ambiente
.env.docker - Para instalar localmente para desenvolvimento, basta ajustar as seguintes variáveis no arquivo Dockerfile, que são necessárias para mapear os proprietários dos arquivos para um usuário do docker;
UIDo usuário numérico que você está logado e que é o dono de todos os arquivos e diretórios no diretório opendatabio (em geral 1000 ou 1001).GDIo grupo numérico ao qual o usuário pertence, geralmente o mesmo que UID.
- O arquivo
Makefilecontém atalhos para os comandos docker-compose usados para construir os serviços configurados nodocker-compose.ymle arquivos auxiliares na pastadocker. - Crie os contêineres do docker usando os atalhos (leia o Makefile para compreender os comandos)
make build
- Inicie os serviços docker implementados
make start
- Veja os contêineres e tente entrar no contêiner laravel
docker ps
make ssh #para entrar no container do app
make ssh-mysql #para entrar no container do mysql container onde você pode acessar a base de dados usando `mysql -uroot -p`
- Instale as dependencias
make composer-install
- Criar a base de dados usando Laravel Migration
make migrate
- Você pode alimentar as tabelas Locations e Taxons usando o seed data para sua versão do OpenDataBio:
make seed-odb
- Se funcionou, então Opendatabio estará disponível em seu navegador http::/localhost:8080.
- O banco de dados estará disponível através do phpmyadmin em http://localhost:8082/
- Faça login com o super-usuário
admin@example.orge a senhapassword1 - Configurações adicionais nesses arquivos são necessárias para um ambiente de produção e implantação;
Persistência de dados
As containers criados pelo Docker podem ser excluídos e regerados sem perder os dados As tabelas mysql são armazenadas em um volume, que se apagado irá excluir a base de dados completamente.
docker volume list
Usando
Leia o conteúdo do arquivo Makefile
make stop
make start
make restart
docker ps
...
Se você tiver problemas e alterou os arquivos do docker, pode ser necessário reconstruir:
#apaga todas as imagens sem apagr a base de dados
docker system prune -a #aceitar com Yes
make build
make start
2.4 - Personalizar a instalação
Mudanças simples que podem ser implementadas no layout de um site OpenDataBio
Logo e imagem de fundo
Para substituir o logotipo da barra de navegação e a imagem da página inicial,
apenas coloque seus arquivos de imagem substituindo os arquivos em /public/custom/ sem alterar seus nomes.
Textos e informações
Para alterar o texto de boas-vindas da página inicial, altere os valores para cada entrada nos arquivos:
/resources/lang/en/customs.php/resources/lang/pt/customs.php- Não remova as chaves de entrada. Defina como
nullpara suprimir a exibição no rodapé e na página inicial.
NavBar e Rodapé
- Se você deseja alterar a cor da barra de navegação superior e do rodapé,
basta substituir a classe css Boostrap 5 nas tags e arquivos correspondentes na pasta
/resources/view/layout. - Você pode adicionar html adicional ao rodapé e barra de navegação, alterar o tamanho do logotipo, etc… como desejar.
3 - Serviços de API
Cada instalação do OpenDataBio fornece um serviço de API, permitindo aos usuários OBTER, INSERIR, ATUALIZAR dados programaticamente. O serviço é de acesso aberto a dados públicos, e requer autenticação do usuário para INSERIR e ATUALIZAR dados, ou para OBTER dados de acesso restrito.
O OpenDataBio Application Programming Interface -API permite que os usuários interajam com um banco de dados OpenDataBio para exportar, importar e atualizar dados sem usar a interface web.
 O pacote OpenDataBio R é um cliente para esta API, permitindo a interação com o repositório de dados diretamente do R.
O pacote OpenDataBio R é um cliente para esta API, permitindo a interação com o repositório de dados diretamente do R.
A API OpenDataBio permite a consulta ao banco de dados e a importação/edição de dados por meio de uma interface inspirada em REST. Todas as solicitações e respostas da API são formatadas em JSON.
A interação com a API do OpenDataBio
Uma simples chamada para a API OpenDataBio possui quatro partes independentes:
- HTTP-verbo -
GETpara exportações,POSTpara importações ePUTpara atualizações. - URL-base - o URL usado para acessar seu servidor OpenDataBio + mais
/ api / v0. Por exemplo,http:// opendatabio.inpa.gov.br/api/v0 - endpoint - representa o objeto ou coleção de objetos que você deseja acessar, por exemplo, para consultar nomes taxonômicos, o endpoint é “taxons”
- parâmetros de solicitação - representam a filtragem e o processamento que devem ser feitos com os objetos e são representados na chamada da API após um ponto de interrogação. Por exemplo, para recuperar apenas nomes taxonômicos válidos finalize a solicitação com
?valid = 1.
A chamada API acima pode ser inserida em um navegador para OBTER dados de acesso público. Por exemplo, para obter a lista de táxons válidos de uma instalação do OpenDataBio, a solicitação da API poderia ser:
https://opendb.inpa.gov.br/api/v0/taxons?valid=1&limit=10
Quando usar o OpenDataBio R package essa mesma chamada seria algo como odb_get_taxons(list(valid=1,limit=10)).
A resposta será algo como:
{
"meta":
{
"odb_version":"0.9.1-alpha1",
"api_version":"v0",
"server":"http://opendb.inpa.gov.br",
"full_url":"https://opendb.inpa.gov.br/api/v0/taxons?valid=1&limit1&offset=100"},
"data":
[
{
"id":62,
"parent_id":25,
"author_id":null,
"scientificName":"Laurales",
"taxonRank":"Ordem",
"scientificNameAuthorship":null,
"namePublishedIn":"Juss. ex Bercht. & J. Presl. In: Prir. Rostlin: 235. (1820).",
"parentName":"Magnoliidae",
"family":null,
"taxonRemarks":null,
"taxonomicStatus":"accepted",
"ScientificNameID":"http:\/\/tropicos.org\/Name\/43000015 | https:\/\/www.gbif.org\/species\/407",
"basisOfRecord":"Taxon"
}]}
Autenticação da API
- Não é obrigatória para obter quaisquer dados de acesso público numa base de dados ODB, que por padrão inclui Localidades, Taxons, Referências Bibliográficas, Pessoas e Variáveis.
- Autenticação é necessária para OBTER quaisquer dados que não sejam de acesso público e para INSERIR e ATUALIZAR dados.
- A autenticação é feita usando um
token API, que pode ser encontrado no seu perfil de usuário na interface do aplicativo. O token é atribuído a um único usuário do banco de dados e não deve ser compartilhado, exposto, enviado por e-mail ou armazenado em controles de versão. - Para autenticar na API OpenDataBio, use o token no cabeçalho “Authorization” da solicitação da API. Ao usar o cliente R, passe o token para a função
odb_config`` cfg = odb_config (token = "seu-token-aqui").
Os usuários somente terão acesso aos dados para os quais o usuário tem permissão e para quaisquer dados com acesso público na base de dados. O acesso à Medições, Indivíduos, Vouchers e Mídia depende das permissões compreendidas pelo token do usuário.
Versões da API
A API OpenDataBio segue seu próprio número de versão. Isso significa que o cliente pode esperar usar o mesmo código e obter as mesmas respostas, independentemente da versão do OpenDataBio que o servidor está executando. Todas as alterações feitas na mesma versão da API devem ser compatíveis com versões anteriores. Nosso controle de versão da API é controlado pelo URL, portanto, para solicitar uma versão específica da API, use o número da versão entre o URL base e o endpoint:
http://opendatabio.inpa.gov.br/opendatabio/api/v1/taxons
http://opendatabio.inpa.gov.br/opendatabio/api/v2/taxons
Version v0
A versão 0 da API (v0) é uma versão instável. A primeira versão estável será a versão 1 da API.3.1 - Referência rápida
Formato da API
base-URL + ‘/api/v0/’ + endpoint + ‘?’ + request-parametersExemplo para o Taxons endpoint
https://opendb.inpa.gov.br/api/v0/taxons?valid=1&limit=2&offset=10OBTER DADOS - GET
Parâmetros GET compartilhados
Todos os endpoints compartilham estes parâmetros GET:
idretorna apenas o recurso especificado. Pode ser um número ou uma lista delimitada por vírgulas, comoapi/v0/locations? Id = 1,50,124limit: limita o número de itens que devem ser retornados (deve ser maior que 0). Exemplo:api/v0/taxons?limit=10offset: o registro inicial a partir do qual deseja extrair, para ser usado com limite ao tentar baixar uma grande quantidade de dados. Exemplo:api/v0/taxons?offset=1000&limit=10retorna 10 registros começando da posição 1K da consulta realizada.fields: o campo ou campos que devem ser retornados. Cada EndPoint possui seus próprios campos, mas há duas palavras especiais, simple (padrão) e all, que retornam diferentes coleções de campos.fields = allpode retornar sub-objetos para cada objeto.fields ='raw'irá retornar a tabela bruta, e isso torna a busca muito mais rápida, embora será mais difícil interpretar alguns valores. Exemplo:api/v0/taxons?fields='id,scientificName,valid'save_job: usandosave_job=1na lista de parâmetros, será gerado um job com a sua busca e os dados poderão ser baixados após a finalização do job através da API userjobs api.
Baixar muitos dados?
Os parâmetroslimit e offset podem ser usados para dividir sua busca em partes. Alternativamente, use a opção save_job=T e depois baixe os dados com o parametro get_file=T da API userjobs.
wildcards
Alguns parâmetros aceitam um asterisco como curinga, entãoapi/v0/taxons?name=Euterpe retornará táxons com nome exatamente como “Euterpe”, enquanto api/v0/taxons?name=Eut* retornará nomes começando com “Eut”.
Parâmetros GET específicos
| Endpoint | Descrição | Parâmetros possíveis |
|---|---|---|
| / | Testa o acesso | nenhum |
| bibreferences | Obter referências bibliográficas | id, bibkey |
| biocollections | Lista de BioColeções registradas | id |
| datasets | Lista Conjuntos de Dados ou Baixa os arquivos de versões de conjuntos de dados | id, list_versions, file_name |
| individuals | Obter dados de Indivíduos (organisms) | id, location, location_root,taxon, taxon_root, tag,project, dataset |
| individual-locations | Obter localizações (ocorrências) de indivíduos | individual_id, location, location_root,taxon, taxon_root, dataset |
| languages | Lista de Idiomas | |
| measurements | Obter Medições | id, taxon,dataset,trait,individual,voucher,location |
| locations | Obter localidades | root, id, parent_id,adm_level, name, limit, querytype, lat, long,project,dataset |
| persons | Lista de Pessoas | id, search, name, abbrev, email |
| projects | Lista de projects | id |
| taxons | Lista de Taxons | root, id, name,level, valid, external, project,dataset |
| traits | Lista Variáveis (traits) | id, name |
| vouchers | Lista Vouchers | id, number, individual, location, collector, location_root,taxon, taxon_root,project, dataset |
| userjobs | Lista Jobs de Usuário | id, status |
Importar dados - POST
Atenção
- A importação de dados de arquivos por meio da interface da web requer a especificação dos parâmetros do verbo POST da API listados abaixo
- As importações em lote de Referências bibliográficas e Arquivos de Mídia só são possíveis por meio da interface no navegador.
| Endpoint | Descrição | Parâmetros POST |
|---|---|---|
| biocollections | Importar BioColeções | name, acronym |
| individuals | Importar indivíduos | collector, tag, dataset, date, (location or latitude + longitude)**, altitude, location_notes, location_date_time, x, y, distance , angle , notes , taxon , identifier , identification_date , modifier, identification_notes , identification_based_on_biocollection, identification_based_on_biocollection_id , identification_individual |
| individual-locations | Importar Localidades de Indivíduos | individual, (location or latitude + longitude), altitude, location_notes, location_date_time, x, y, distance, angle |
| locations | Importar Localidades | name, adm_level, (geom or lat+long)** , parent, altitude, datum, x, y , startx, starty, notes, ismarine |
| measurements | Importar Medições | dataset, date, object_type, object_type, person, trait_id, value**, link_id, bibreference, notes, duplicated,location, parent_measurement |
| persons | Importar Pessoas | full_name**, abbreviation, email, institution, biocollection |
| traits | Importar Variáveis | export_name, type, objects, name, description**, units, range_min, range_max, categories, wavenumber_min and wavenumber_max, value_length, link_type, bibreference, tags |
| taxons | Importar Taxons | name**, level, parent, bibreference, author, author_id or person, valid, mobot, ipni, mycobank, zoobank, gbif |
| vouchers | Importar Vouchers | individual, biocollection, biocollection_type, biocollection_number, number, collector, date, dataset, notes |
** indica campos obrigatórios.
Atualizar dados - PUT
Atenção
Somente os endpoints listados abaixo podem ser atualizados usando a API e somente os campos PUT listados podem ser atualizados em cada endpoint. Os valores dos campos são os mesmos como explicados para a API POST, exceto que em todos os casos oid do registro a ser atualizado também deve ser fornecido.
| Endpoint | Description | PUT Fields |
|---|---|---|
| individuals | Atualizar Indivíduos | (id or individual_id),collector, tag, dataset, date, notes , taxon , identifier , identification_date , modifier, identification_notes , identification_based_on_biocollection, identification_based_on_biocollection_id , identification_individual |
| individual-locations | Atualizar Localidades de Indivíduos | (id or individual_location_id), individual, (location or latitude + longitude), altitude, location_notes, location_date_time, x, y, distance, angle |
| locations | Atualizar Localidades | (id or location_id), name, adm_level, (geom or lat+long) , parent, altitude, datum, x, y , startx, starty, notes, ismarine |
| measurements | Atualizar Medições | (id or measurement_id), dataset, date, object_type, object_type, person, trait_id, value, link_id, bibreference, notes, duplicated,location, parent_measurement |
| persons | Atualizar Pessoas | (id or person_id),full_name, abbreviation, email, institution, biocollection |
| vouchers | Atualizar Vouchers | (id or voucher_id),individual, biocollection, biocollection_type, biocollection_number, number, collector, date, dataset, notes |
Nomenclature Types
| Tipo Nomenclatural : código numérico | |
|---|---|
| NotType : 0 | Isosyntype : 8 |
| Type : 1 | Neotype : 9 |
| Holotype : 2 | Epitype : 10 |
| Isotype : 3 | Isoepitype : 11 |
| Paratype : 4 | Cultivartype : 12 |
| Lectotype : 5 | Clonotype : 13 |
| Isolectotype : 6 | Topotype : 14 |
| Syntype : 7 | Phototype : 15 |
Níveis taxonômicos (Ranks)
| Nível | Nível | Nível | Nível |
|---|---|---|---|
-100 clade |
60 cl., class |
120 fam., family |
210 section, sp., spec., species |
0 kingdom |
70 subcl., subclass |
130 subfam., subfamily |
220 subsp., subspecies |
10 subkingd. |
80 superord., superorder |
150 tr., tribe |
240 var., variety |
30 div., phyl., phylum, division |
90 ord., order |
180 gen., genus |
270 f., fo., form |
40 subdiv. |
100 subord. |
190 subg., subgenus, sect. |
3.2 - Obter dados - GET
- O pacote OpenDataBio-R é um cliente para esta API.
- Não é necessária autenticação para acessar dados com uma política de acesso público
- Token de autenticação necessário apenas para obter dados com uma política de acesso não pública
Parâmetros GET compartilhados
Os endpoints compartilham estes parâmetros GET:
idretorna apenas o recurso especificado. Pode ser um número ou uma lista delimitada por vírgulas, comoapi/v0/locations? Id = 1,50,124limit: limita o número de itens que devem ser retornados (deve ser maior que 0). Exemplo:api/v0/taxons?limit=10offset: o registro inicial a partir do qual deseja extrair, para ser usado com limite ao tentar baixar uma grande quantidade de dados. Exemplo:api/v0/taxons?offset=1000&limit=10retorna 10 registros começando da posição 1K da consulta realizada.fields: o campo ou campos que devem ser retornados. Cada EndPoint possui seus próprios campos, mas há duas palavras especiais, simple (padrão) e all, que retornam diferentes coleções de campos.fields = allpode retornar sub-objetos para cada objeto.fields ='raw'irá retornar a tabela bruta, e isso torna a busca muito mais rápida, embora será mais difícil interpretar alguns valores. Exemplo:api/v0/taxons?fields='id,scientificName,valid'save_job: usandosave_job=1na lista de parâmetros, será gerado um job com a sua busca e os dados poderão ser baixados após a finalização do job através da API userjobs api.
Baixar muitos dados?
Os parâmetroslimit e offset podem ser usados para dividir sua busca em partes. Alternativamente, use a opção save_job=T e depois baixe os dados com o parametro get_file=T da API userjobs.
wildcards
Alguns parâmetros aceitam um asterisco como curinga, entãoapi/v0/taxons?name=Euterpe retornará táxons com nome exatamente como “Euterpe”, enquanto api/v0/taxons?name=Eut* retornará nomes começando com “Eut”.
BibReferences Endpoint
O endpoint
bibreferencesinterage com a tabela [bibreferences](/docs/concepts/auxiliary-objects/# bibreferences). Seu uso básico é obter as referências bibliográficas registradas.
Parâmetros GET
id = listretorna apenas referências com o id ou ids fornecidos (exid = 1,2,3,10)bibkey = listretorna apenas referências com bibkey ou bibkeys (exbibkey = ducke1953, mayr1992)taxon = lista de idsretorna apenas referências vinculadas ao táxon informado.limiteoffset. consulte Parâmetros comuns.
Campos de resposta
id- o id de BibReference na tabela bibreferences (um id de banco de dados local)bibkey- a bibkey usada para pesquisar e usar a referência no sistema webyear- o ano de publicaçãoauthor- os autores da publicaçãotitle- o título da publicaçãodoi- a publicação DOI, se presenteurl- um url externo para a publicação, se presentebibtex- o registro de citação de referência no formato [BibTex] (http://www.bibtex.org/)
Datasets Endpoint
O endpoint
datasetsinterage com a tabela [Conjuntos de Dados] (/docs/concepts/data-access/#datasets) e com versões estáticas dos Conjuntos de Dados. Seu uso básico é obter uma lista dos Conjuntos de dados registrados. Útil para obter dataset_ids para importar medições, indivíduos, vouchers e midia. Além disso você pode baixar arquivos de versões estáticas dos dados, se alguma foi gerada. Isso permite um acesso aos dados de conjuntos de dados. O acesso aos dados vai depender se a versão tem licensa pública ou se o usuário é colaborador, administrador ou pode ver o conjunto de dados.
Parâmetros GET
id = listretorna apenas referências com o id ou ids fornecidos (exid = 1,2,3,10)list_versions = booleanretorna a lista de arquivos e versões para um ou mais conjuntos de dadosid(exid='1,2,3,4',list_versions=1)file_name = stringretorna os dados do arquivo indicado por esse parâmetro, conforme indicado na lista de versões retornada (exid='1',file_name='2_Organisms.csv')
Campos de resposta
Com list_versions=1
dataset_id- o id do conjunto de dados na tabela de conjuntos de dados (um id de banco de dados local)dataset- o nome do conjunto de dadosversion- o nome da versão estática do conjunto de dadoslicense- a licensa CreativeCommons da versão dos dadosaccess- inicando se é OpenAccess ou se o usuário tem acessoget_params- como informar o argumentoparamsna função odb_get_datasets() do pacote do R para pegar os dados
Com file_name
- Retorna os dados do arquivo indicado
Sem list_versions ou file_name
id- o id do conjunto de dados na tabela de conjuntos de dados (um id de banco de dados local)name- o nome do conjunto de dadosprivacyLevel- o nível de acesso para o conjunto de dadoscontactEmail- o e-mail do administrador do conjunto de dadosdescription- uma descrição do conjunto de dadospolicy- a política de dados, se especificadameasurements_count- o número de medições no conjunto de dadostaggedWidth- a lista de tags aplicadas ao conjunto de dados
Biocollections Endpoint
O endpoint
biocollectionsinterage com a tabela BioColeções. Seu uso básico é obter a lista das Coleções Biológicas cadastradas no banco de dados. Usando para obterbiocollection_idou validar seus códigos para importar dados com os pontos de extremidade Vouchers ou Indivíduos.
Parâmetros GET
id = listretorna apenas as Biocoleções tendo o id ou ids fornecidos (exid = 1,2,3,10)acronymretorna apenas Biocoleções tendo o acrônimo ou acrônimo fornecido (exacronym = INPA, SP, NY)
Campos de resposta
id- o id do repositório ou museu na tabela de Biocoleções (um id de banco de dados local)name- o nome do repositório ou museuacronym- o acrônimo do repositório ou museuirn- apenas para Herbarios, o número do herbário no Index Herbariorum
Individuals Endpoint
O endpoint
individualsinterage com a modelo Indivíduos. Seu uso básico é obter uma lista de indivíduos e dados associados.
Parâmetros GET
id = número ou lista- retorna indivíduos que têm id ou ids ex:id = 2345,345location = mixed- retorna indivíduos por localidade, pode ser id ou nome ex:location = 24,25,26location = Parcela 25ha(ligações diretas)location_root- igual a location, mas retorna também dos descendentes das localidades informadas (ligações espaciais, tudo o que está dentro da(s) localidade(s))taxon = mixed- o id ou ids, ou nomes de táxons (nomes completos, canonicalName) ex:taxon = Aniba, Ocotea guianensis, Licaria cannela tenuicarpaoutaxon = 456.789,3,4taxon_root- igual a taxon mas retorna também todos os indivíduos identificados como qualquer um dos descendentes dos táxons informados (por exemplo,taxon=Lauraceaeretorn todos os indivíduos da família)project = mixed- o id ou ids ou nomes do projeto, ex:project = 3ouproject = OpenDataBiotag = list- um ou mais códigos de identificação do individual, ex:tag = individuala1,2345,2345Adataset = mixed- o id ou ids ou nomes dos conjuntos de dados, retorna os indivíduos que têm medições nos conjuntos de dados informadoslimiteoffset- Consulte Parâmetros comuns.
Note
Observe que todos os campos de pesquisa (táxon, localidades e conjunto de dados) podem ser especificados como nomes (por exemplo, “taxon = Euterpe edulis”) ou como ids de banco de dados. Se uma lista for especificada para um desses campos, todos os itens da lista devem ser do mesmo tipo, ou seja, você não pode pesquisar por ’taxon = Euterpe, 24’. Além disso, location e taxon têm prioridade sobre location_root e taxon_root se ambos forem informados.Campos de resposta
id- o id do indivíduo na tabela de individuals (um id de banco de dados local)basisOfRecordDWC - será sempre DWC organismorganismIDDWC - uma combinação única local de informações de registro, composta de recordNumber,recordedByMain,locationNamerecordedByDWC - lista separada de abreviações de pessoas registradas (usando pipe " | “)recordedByMain- a primeira pessoa listada em recordedByrecordNumberDWC - um identificador para o indivíduo, pode ser o código em uma etiqueta de alumínio de árvore, um código num anilha de uma ave, um número de coletorrecordedDateDWC - a data de registroscientificNameDWC - a identificação taxonômica atual do indivíduo (sem autores) ou “unidentified”scientificNameAuthorshipDWC - a autoria do táxon. Para taxonomicStatus = não publicado será um nome de pessoa registrado ODBfamilyDWCgenusDWCidentificationQualifierDWC - modificador do nome identificado cf. aff. s.l., etc.identifiedByDWC - pessoa que atribuiu o scientificNamedateIdentifiedDWC - data da identificaçãoidentificationRemarksDWC - notas da identificaçãolocationName- nome da localidadelocationParentName- nome da localidade pai da localidade do indivíduohigherGeographyDWC - a hierarquia completa separada por pipe (’|’) - e.g. Brasil | Amazonas | Rio Preto da Eva | Fazenda Esteio | Reserva km37 | Manaus ForestGeo-PDBFF Plot | Quadrat 100x100 );decimalLatitudeDWC - a latitude em graus decimais. O valor vai depender do tipo de localidade e da posição relativa do indivíduo (i.e. se houver atributos X e Y, ou Angle e Distance eles serão usados para calcular este valor). Se o indivíduo tiver multiplas localidades registradas (e.g. uma ave monitorada), esta será a coordenada da última localidade registrada.decimalLongitudeDWC - mesmo que decimalLatitude, mas para longitude.x- a posição X do indivíduo dentro de uma localidade do tipo Parcelay- a posição Y do indivíduo dentro de uma localidade do tipo Parcelagx- a posição X do indivíduo dentro da localidade pai, quando a localidade for uma subparcela (e.g. protocolo ForestGeo)gy- a posição Y do indivíduo dentro da localidade pai, quando a localidade for uma subparcela (e.g. protocolo ForestGeo)angle- a direção do azimute da posição do indivíduo em relação uma coordenada geográfica (POINT location)distance- a distância do indivíduo a coordenada geográfica (POINT location), quandoangleé informado.organismRemarksDWC - notas associadas ao registro do IndivíduoassociatedMediaDWC - urls para arquivos de media ligados ao indivíduo quando for o casodatasetName- nome do Conjunto de Dados ao qual o indivíduo pertence DWCaccessRights- a política de acesso do Conjunto de dados - DWCbibliographicCitation- a citação para Conjunto de Dados - DWClicense- a licensa para o Conjunto de Dados - DWC
Individual-locations Endpoint
O endpoint
individual-locationsinterage com a tabela individual_location. Seu uso básico é obter dados de ocorrência de indivíduos. Projetado para ocorrências de organismos que se movem e têm vários locais, caso contrário, a mesma informação é recuperada com o Endpoint Individuals.
Parâmetros GET
individual_id = número ou lista- retorna indivíduos que têm id ou ids ex:id = 2345,345location = mixed- retorna indivíduos por localidade, pode ser id ou nome ex:location = 24,25,26location = Parcela 25ha(ligações diretas)location_root- igual a location, mas retorna também dos descendentes das localidades informadas (ligações espaciais, tudo o que está dentro da(s) localidade(s))taxon = mixed- o id ou ids, ou nomes de táxons (nomes completos, canonicalName) ex:taxon = Aniba, Ocotea guianensis, Licaria cannela tenuicarpaoutaxon = 456.789,3,4taxon_root- igual a taxon mas retorna também todos os indivíduos identificados como qualquer um dos descendentes dos táxons informados (por exemplo,taxon=Lauraceaeretorn todos os indivíduos da família)dataset = mixed- o id ou ids ou nomes dos conjuntos de dados, retorna os indivíduos que têm medições nos conjuntos de dados informadoslimiteoffset- Consulte Parâmetros comuns.
Note
Observe que todos os campos de pesquisa (táxon, localidades e conjunto de dados) podem ser especificados como nomes (por exemplo, “taxon = Euterpe edulis”) ou como ids de banco de dados. Se uma lista for especificada para um desses campos, todos os itens da lista devem ser do mesmo tipo, ou seja, você não pode pesquisar por ’taxon = Euterpe, 24’. Além disso, location e taxon têm prioridade sobre location_root e taxon_root se ambos forem informados.Campos de resposta
-
individual_id- o id do indivíduo na tabela de individuals (um id de banco de dados local) -
location_id- o id do local na tabela de locations (um id de banco de dados local) -
basisOfRecord- será sempre ‘occurrence’ - DWC e DWC occurrence; -
occurrenceID- o identificador único para este registro, o indivíduo + local + date_time - DWC -
organismID- o identificador exclusivo para o indivíduo DWC -
recordedDate- a data e hora do registro da ocorrência [DWC] (https://dwc.tdwg.org/terms/#dwc:recordedDate) -
locationName- o nome da localidade -
higherGeographyDWC - a hierarquia completa separada por pipe (’|’) - e.g. Brasil | Amazonas | Rio Preto da Eva | Fazenda Esteio | Reserva km37 | Manaus ForestGeo-PDBFF Plot | Quadrat 100x100 ); -
decimalLatitudeDWC - a latitude em graus decimais. O valor vai depender do tipo de localidade e da posição relativa do indivíduo (i.e. se houver atributos X e Y, ou Angle e Distance eles serão usados para calcular este valor). Se o indivíduo tiver multiplas localidades registradas (e.g. uma ave monitorada), esta será a coordenada da última localidade registrada. -
decimalLongitudeDWC - mesmo que decimalLatitude, mas para longitude. -
x- a posição X do indivíduo dentro de uma localidade do tipo Parcela -
y- a posição Y do indivíduo dentro de uma localidade do tipo Parcela ForestGeo) -
angle- a direção do azimute da posição do indivíduo em relação uma coordenada geográfica (POINT location) -
distance- a distância do indivíduo a coordenada geográfica (POINT location), quandoangleé informado. -
organismRemarksDWC - notas associadas ao registro do Indivíduo -
associatedMediaDWC - urls para arquivos de media ligados ao indivíduo quando for o caso -
datasetName- nome do Conjunto de Dados ao qual o indivíduo pertence DWC -
accessRights- a política de acesso do Conjunto de dados - DWC -
bibliographicCitation- a citação para Conjunto de Dados - DWC -
license- a licensa para o Conjunto de Dados - DWC -
scientificNameDWC - a identificação taxonômica atual do indivíduo (sem autores) ou “unidentified” -
familyDWC -
datasetName- nome do Conjunto de Dados ao qual o indivíduo pertence DWC -
accessRights- a política de acesso do Conjunto de dados - DWC -
bibliographicCitation- a citação para Conjunto de Dados - DWC -
license- a licensa para o Conjunto de Dados - DWC -
minimumElevation- a altitude da ocorrência, se registrado - DWC -
occurrenceRemarks- qualquer nota associada ao registro de ocorrência do indivíduo - DWC
Measurements Endpoint
O endpoint
measurementsinterage com o modelo Medições. Seu uso básico é obter dados vinculados a indivíduos, táxons, localidades ou vouchers, independentemente dos conjuntos de dados, por isso é útil quando você deseja obter medições específicas de diferentes conjuntos de dados aos quais você tem acesso. Se você quiser um conjunto de dados completo, mais fácil usar a interface da web, pois ela prepara um conjunto completo de medições do conjunto de dados e todas as tabelas de dados associados.
Parâmetros GET
id = lista de idsretorna apenas a medição ou medições tendo o id ou ids fornecidos (exid = 1,2,3,10)taxon = lista de ids ou nomesretorna apenas as medições relacionadas aos táxons, tanto medidas diretas quanto medidas indiretas de seus indivíduos e vouchers. Não considera os táxons descendentes para esse uso, em vez disso, usetaxon_root. Por exemplo,taxon = Aniba, Licariairá retornar apenas medições diretamente ligadas ao gênero e vouchers identificados em nível de gênero.taxon_root = lista de ids ou nomessemelhantes ataxon, mas obtém também medições para táxons descendentes da consulta informada (extaxon = Lauraceaeobterá medições vinculadas a Lauraceae e qualquer táxon que pertença a ela);dataset = lista de idsretorna apenas as medições pertencentes aos datasets informados (exdataset = 1,2)trait = lista de ids ou export_namesretorna apenas as medições para as variáveis informadas (extrait=DBH, DBHpomoudataset=2&trait=DBH)individual = lista de ids indivíduosretorna apenas as medições para os indivíduos com esses id informados (exindividual = 1000,1200)voucher = lista de ids de vouchersretorna apenas as medições para os vouchers com esses id informados (exvoucher = 345,321)location = lista de ids de localidadesretorna apenas medições para as localidades informados (ex:location = 4,3,2,1) - não recupera medições para indivíduos e vouchers nessas locais, apenas medições relacionados às próprias localidaes, como, por exemplo, dados de levantamentos de solo de parcelas.limiteoffset- Consulte Parâmetros comuns.
Campos de resposta
id- o id da medição na tabela de measurements (id do banco de dados local)basisOfRecordDWC - será sempre ‘MeasurementsOrFact’ [dwc measureorfact](DWC measureorfactmeasured_type- o objeto medido, um de ‘Individual’, ‘Location’, ‘Taxon’ ou ‘Voucher’measured_id- o id do objeto medido na respectiva tabela de objetos (individuals.id, locations.id, taxons.id, vouchers.id)measurementIDDWC - um identificador único para a mediçãomeasurementTypeDWC - o export_name para a Variável medidameasurementValueDWC - o valor da medição - dependerá do tipo de medição ver VariáveismeasurementUnitDWC - a unidade de medida para variáveis quantitativasmeasurementDeterminedDateDWC - a data da mediçãomeasurementDeterminedByDWC - Pessoa responsável pela mediçãomeasurementRemarksDWC - nota de texto associada a este registro de mediçãoresourceRelationshipDWC - o objeto medido (recurso) - um de ’location’,’taxon’,‘organism’,‘preservedSpecimen’resourceRelationshipIDDWC - o id do resourceRelationshiprelationshipOfResourceDWC - será sempre ‘measurement of’scientificNameDWC - a identificação taxonômica relacionada à medição, se for o casofamilyDWC - a família taxonômica, se for o caso.datasetName- nome do Conjunto de Dados ao qual a medição pertence DWCaccessRights- a política de acesso do Conjunto de dados - DWCbibliographicCitation- a citação para Conjunto de Dados - DWClicense- a licensa para o Conjunto de Dados - DWCmeasurementLocationId- o ODB id da localidade associada à mediçãomeasurementParentId- o ODB id de outra medição relacionada (a medição pai da qual esta depende)decimalLatitudeDWC - a latitude em graus decimais da medição ou do objeto medido.decimalLongitudeDWC - a longitude em graus decimais da medição ou do objeto medido.
Media Endpoint
O endpoint
mediainterage com a tabela media. Seu uso básico é obter os metadados associados aos arquivos de mídia, incluindo a URL dos arquivos.
Parâmetros GET
individual = número ou lista- retorna mídia associada aos indivíduos com id ou ids, por exemplo:id = 2345,345voucher = number or list- retorna mídia associada aos vouchers com id ou ids, por exemplo:id = 2345,345location = mixed- retorna mídia associada a localidades, pode ser id ou nome ex:location = 24,25,26location = Parcela 25ha(ligações diretas)location_root- igual a location, mas retorna também dos descendentes das localidades informadas (ligações espaciais, tudo o que está dentro da(s) localidade(s))taxon = mixed- o id ou ids, ou nomes de táxons (nomes completos, canonicalName) ex:taxon = Aniba, Ocotea guianensis, Licaria cannela tenuicarpaoutaxon = 456.789,3,4taxon_root- igual a taxon mas retorna também todos os indivíduos identificados como qualquer um dos descendentes dos táxons informados (por exemplo,taxon=Lauraceaeretorn todos os indivíduos da família)dataset = mixed- o id ou ids ou nomes dos conjuntos de dados, retorna mídia pertencentes aos conjuntos de dados informadoslimiteoffset- Consulte Parâmetros comuns.
Note
Observe que todos os campos de pesquisa (táxon, localidades e conjunto de dados) podem ser especificados como nomes (por exemplo, “taxon = Euterpe edulis”) ou como ids de banco de dados. Se uma lista for especificada para um desses campos, todos os itens da lista devem ser do mesmo tipo, ou seja, você não pode pesquisar por ’taxon = Euterpe, 24’. Além disso, location e taxon têm prioridade sobre location_root e taxon_root se ambos forem informados.Response fields
id- o id da medição na tabela measurements (local database id)basisOfRecordDWC - será sempre ‘MachineObservation’ DWCmodel_type- o objeto relacionado ‘Individual’, ‘Location’, ‘Taxon’ or ‘Voucher’model_id- o id do objeto relacionado na respectiva tabela (individuals.id, locations.id, taxons.id, vouchers.id)resourceRelationshipDWC - o objeto relacionado - one of ’location’,’taxon’,‘organism’,‘preservedSpecimen’resourceRelationshipIDDWC - o id não numérico do resourceRelationshiprelationshipOfResourceDWC - será um ‘dwcType’recordedByDWC - lista separada por pipe “|” das Pessoas autoresrecordedDateDWC - a data do arquivo de mídiascientificNameDWC - a identificação taxonômica do objeto na mídia, quando for o caso.familyDWCdwcTypeDWC - um de StillImage, MovingImage, SoundfamilyDWC - a família taxonômica, se for o caso.datasetName- nome do Conjunto de Dados ao qual a mídia pertence DWCaccessRights- a política de acesso do Conjunto de dados - DWCbibliographicCitation- a citação para Conjunto de Dados - DWClicense- a licensa para o Conjunto de Dados - DWCfile_name- nome do arquivofile_url- o endereço do arquivo
Languages EndPoint
O endpoint
languagesinterage com a tabela Language. Seu uso básico é obter uma lista de idiomas registrados para importar Traduções do usuário como é o caso dos nomes de Variáveis e categorias, ou descrições de arquivos de mídia.
Campos de resposta
id- o id do idioma na tabela de idiomas (um id de banco de dados local)code- o código do idioma;name- o nome do idioma;
Locations Endpoint
O endpoint
locationsinterage com a modelo Localidades. Seu uso básico é obter uma lista de localidades registradas
Parâmetros GET
id = listretorna apenas localidades com o id ou ids fornecidos (ex:id = 1,2,3,10)adm_level = numberretorna apenas localidades para o nível ou tipo especificado:- 2 para o país; 3 para a primeira divisão dentro do país (província, estado); 4 para a segunda divisão (por exemplo, município) … até adm_level 10 como áreas administrativas (Geometria: polígono, multipolygon);
- 97 é o código para Camadas Ambientais (Geometria: polygon, multipolygon);
- 98 é o código para Áreas Indígenas (Geometria: polygon, multipolygon);
- 99 é o código para Unidades de Conservação (Geometria: polygon, multipolygon);
- 100 é o código para PARCELAS e SUB-PARCELAS (Geometria: polygon ou point);
- 101 para transectos (geometria: point ou linestring)
- 999 para qualquer localidades de ‘PONTO’ como waypoints GPS (Geometria: point);
name = stringretorna apenas as localidades cujo nome corresponde à string de pesquisa. Você pode usar asterisco como curinga. Exemplo:nome=Manausounome=*Ducke*para encontrar o nome que possui a palavra Ducke;parent_id = listretorna as localidades cujo pai imediato na lista (ex: parent_id = 2,3)root = numberid da localidade para pesquisar, retorna as localidades para a id especificada junto com todas as localidades descendentes; exemplo: encontre o id para o Brasil e use seu id como root para obter todos as localidades pertencentes ao Brasil;querytypeum de “exact”,“parent” ou “closest” e deve ser fornecido comlatelong:- quando
querytype = exactencontrará uma localidade do tipo POINT que tem a correspondência exata delatelong; - quando
querytype = parentencontrará a localidade pai mais inclusiva dentro da qual as coordenadas fornecidas porlatelongcaem; - quando
querytype = closestirá encontrar a localidade mais próxima das coordenadas dadas porlatelong; Ele pesquisará apenas os locais mais próximos comadm_level>99, veja acima. latelongdevem ser coordenadas válidas em graus decimais (negativo para Sul e Oeste);
- quando
fields = listespecifica quais campos você deseja obter com sua consulta (veja abaixo os nomes dos campos), ou use as opções ‘all’ ou ‘simple’, para obter o conjunto completo e as colunas mais importantes, respectivamenteproject = mixed- id ou nome do projeto (pode ser uma lista) retorna as localidades utilizadas em um ou mais projetosdataset = mixed- id ou nome de um conjunto de dados (pode ser uma lista) retorna as localidades utilizadas a um ou mais conjuntos de dadoslimiteoffset- Consulte Parâmetros comuns.
Note que id, search, parent e root não devem ser combinadas na mesma busca;
Response fields
id- o id da localidade na tabela locations (um id de banco de dados local)basisOfRecordDWC - sempre conterá ’location’ (DWC locationlocationName- o nome do local (se o país for o nome do país, se indicar o nome do estado, etc …)adm_level- o valor numérico para o nível administrativo ODB (2 para países, etc)levelName- o nome do nível administrativo ODBparent_id- o id da localidade pai na tabela locationsparentName- o nome do pai imediato da localidadehigherGeography[DWC] (https://dwc.tdwg.org/terms/#dwc:higherGeography) - o caminho completo das localidades pais,(por exemplo, Brasil | São Paulo | Cananéia);footprintWKTDWC - a representação geométrica da localidade em formato WKT; para localidades comadm_level==100(parcelas) ouadm_level==101mas informadas como POINT, as geometrias do polygon ou linestring, respectivamente, serão geradas a partir das dimensões especificadas para essas localidades.xey- (metros) para localidades do tipo Parcela (100 == adm_level), as dimensões X e Y. Para localidades do tipo Transecto (101 == adm_level), x pode ser o comprimento do transecto e Y um valor de buffer para inclusão de indivíduosstartxestarty- (meters) se a localidde for uma subparcela (100 == adm_level com localidade pai também adm_level==100), esses valores representam a posição X e Y da subparcela em relação as coordenadas 0,0 da parcela pai;distance- apnas quando querytype=closest este valor representa a distância em metros da localidade pesquisada a localidade obtidalocationRemarksDWC - quaquer nota associada ao registro da localidadedecimalLatitudeDWC - depende do adm_level: se adm_level<=99, a latitude do centroid; se adm_level == 999 (point), a latitude do ponto; se adm_level==100 (parcela) or 101 (transecto), mas estes informados com uma geometria do tipo POINT, a latitude do POINT, caso contrário, se geometria POLYGON ou LINESTRING, o a coordenada do primeiro ponto da geometria.decimalLongitudeDWC - mesmo para decimalLatitudegeoreferenceRemarksDWC - explicação sobre como decimalLatitude e decimalLongitude form calculadosgeodeticDatum-DWC - o geodeticDatum informado para a geometria (ODB não faz reprojeções espaciais, assumes que dados espaciais são sempre WSG84)
Persons Endpoint
O endpoint
personsinterage com a tabela Person. O uso básico é obter uma lista de pessoas cadastradas (coletores de dados, especialistas taxonômicos, etc).
Parâmetros GET
id = listretorna apenas pessoas com o id ou ids fornecidos (ex:id = 1,2,3,10)name = string- retorna pessoas cujo nome corresponde à string especificada. Você pode usar asterisco como curinga. Ex:name =*ducke*abbrev = string, retorna pessoas cuja abreviação corresponde à string especificada. Você pode usar asterisco como curinga.email = string, retorna pessoas cujo e-mail corresponde à string especificada. Você pode usar asterisco como curinga.search = string, retorna pessoas cujo nome, abreviatura ou e-mail corresponde à string especificada. Você pode usar asterisco como curinga.limiteoffset- Consulte Parâmetros comuns.
Campos de resposta
id- o id da pessoa na tabela de pessoas (um id de banco de dados local)full_name- o nome da pessoa;abbreviation- o nome da pessoa (estes são valores ÚNICOS em um banco de dados OpenDataBio)email- o e-mail, se registrado ou a pessoa é o usuárioinstitution- a instituição de pessoas, se registradanotes- quaisquer notas registradas;biocollection- o nome da Coleção Biológica (Biocollections, etc) à qual a pessoa está associada
Projects EndPoint
O endpoint
projectsinterage com a tabela [projects](/docs /concepts /data-access /#projects). O uso básico é obter os projetos registrados.
Parâmetros GET
id = listretorna apenas projetos com o id ou ids fornecidos (exid = 1,2,3,10)
Campos de resposta
id- o id do projeto na tabela de projetos (um id de banco de dados local)fullname- nome do projetocontactEmail- o e-mail do administrador do projetoindividual_count- o número de indivíduos no projetovouchers_count- o número de vouchers no projeto
Taxons Endpoint
O endpoint
taxonsinterage com a tabela [taxons](/docs /concepts /core-objects/#taxons). O uso básico é obter uma lista de nomes taxonômicos registrados.
Parâmetros GET
id = listretorna apenas táxons com o id ou ids fornecidos (ex:id = 1,2,3,10)name = searchretorna apenas táxons com fullname (sem autores) correspondendo à string de pesquisa. Você pode usar asterisco como curinga.root = numberretorna o táxon para o id especificado junto com todos os seus descendenteslevel = numberretorna apenas táxons para o taxonRank.valid = 1retorna apenas nomes válidosexternal = 1retorna os números de referência Tropicos, IPNI, MycoBank, ZOOBANK ou GBIF. Você precisa especificarexternalrefsna lista de campos para retorná-los!project = mixed- id ou nome do projeto (pode ser uma lista) retorna os táxons pertencentes a um ou mais Projetosdataset = mixed- id ou nome de um conjunto de dados (pode ser uma lista) retorna os táxons pertencentes a um ou mais conjuntos de dadoslimiteoffset- Consulte Parâmetros comuns.
Note que id, name e root não devem ser combinadas na mesma busca;
Campos de resposta
id- este id ODB para este registro de táxons na tabela de táxonssenior_id- se inválido, o id do nome aceito para este taxon (acceptedNameUsage) - somente quando taxonomicStatus == ‘invalid’parent_id- o id do táxon paiauthor_id- o id da pessoa que definiu o táxon para nomes não publicados (ter um author_id significa que o táxon não foi publicado)scientificNameDWC - o nome taxonômico completo sem autores (ou seja, incluindo o nome do gênero e epíteto do nome da espécie)ScientificNameIDDWC - IDs de bancos de dados nomenclaturais, se qualquer referência externa for armazenada para este registro de táxontaxonRankDWC - o nível taxonômico do taxon registradolevel- o valor numérico do taxonRank para o OpenDataBioscientificNameAuthorshipDWC - a autoria do táxon. Para não publicado será um nome de pessoa registrado ODBnamePublishedIn- referência bibliográfica unificada (ou seja, o formato curto ou um trecho da referência do bibtex atribuído). Isso será recuperado principalmente de bancos de dados nomenclaturais; As referências vinculadas a táxons podem ser extraídas com o endpoint BibReferences.taxonomicStatusDWC - um de ‘accepted’, ‘invalid’ ou ‘unpublished’; se invalid, os campos senior_id and acceptedNameUsage serão preenchidos.parentNameUsage[DWC] (https://dwc.tdwg.org/terms/#dwc:parentNameUsage) - o nome do táxon pai, se espécie, gênero, se gênero, família e assim por diantefamily[DWC] (https://dwc.tdwg.org/terms/#dwc:family) - o nome da família taxonômica quando for o caso.higherClassification[DWC] (https://dwc.tdwg.org/terms/#dwc:higherClassification) - a classificação hierárquica taxonômica completa, separada por “|” (incluirá apenas táxons registrados neste banco de dados)acceptedNameUsage[DWC] (https://dwc.tdwg.org/terms/#dwc:acceptedNameUsage) - se taxonomicStatus for inválido, o nome científico válido para este táxonacceptNameUsageID[DWC] (https://dwc.tdwg.org/terms/#dwc:acceptedNameUsageID) - se taxonomicStatus for inválido os ids ScientificNameID do táxon válidotaxonRemarks[DWC] (https://dwc.tdwg.org/terms/#dwc:taxonRemarks) - qualquer nota que o registro do táxon possa terbasisOfRecord[DWC] (https://dwc.tdwg.org/terms/#dwc:basisOfRecord) - será sempre ’taxon’externalrefs- os números de referência Tropicos, IPNI, MycoBank, ZOOBANK ou GBIF
Traits Endpoint
O endpoint
traitsinterage com o modelo Variáveis. O uso básico é obter uma lista de variáveis e categorias de variáveis para importar Medições.
Parâmetros GET
id = listaretorna apenas variáveis tendo o id ou ids fornecidos (exid = 1,2,3,10);name = stringretorna apenas variáveis com oexport_nameindicado (ex:name = DBH) - para mais opções de busca, baixe toda a biblioteca e filtre localmente.categories- se verdadeiro, retorna as categorias para variáveis categóricaslanguage = mixedidioma para retorno dos nomes e descrições da variável e das categorias para variáveis categóricas. Os valores podem ser ’language_id’, ’language_code’ ou ’language_name’;bibreference = boolean- se verdadeiro, inclui a [BibReference](/docs/concepts/auxiliary-objects/# bibreference) associado à definição da variável, se houver.limiteoffset- Consulte Parâmetros comuns.
Campos de resposta
id- o id do Trait na tabela odbtraits (um id de banco de dados local)type- o código numérico que define o tipo de característicatypename- o nome do tipo de Traitexport_name- o valor do nome de exportaçãomeasurementTypeDWC - o mesmo que export_name para compatibilidade DWCmeasurementMethodDWC - combine nome, descrição e categorias se aplicável (incluído na API Measurement GET, para compatibilidade DWC)measurementUnit- a unidade de medida para características quantitativasmeasurementTypeBibkeys- os bibkeys das referências bibliográficas associadas à definição do trait, separados por barra vertical ‘|’taggedWith- o nome das tags ou palavras-chave associadas à definição da característica, separados por barra vertical ‘|’range_min- o valor mínimo permitido para características quantitativasrange_max- o valor máximo permitido para características quantitativaslink_type- se o tipo de link trait, a classe do objeto ao qual o traço se vincula (atualmente apenas Taxon)name- o nome do trait no idioma solicitado ou no idioma padrãodescription- a descrição do traço no idioma solicitado ou no idioma padrãovalue_length- o comprimento dos valores permitidos para variáveis Espectrais (juntamente com range_min e range_max, define o espaçamento entre os valores que fazem o espectro medido.objects- os tipos de objeto para os quais o traço pode ser usado, separados por barra vertical ‘|’categories- para variáveis categóricas e ordinais, um sub conjunto com os seguintes campos para cada categoria:id,name,descriptionerank. O rank é informativo apenas para variáveis ORDINAIS, mas é reportado para todas as variáveis categóricas.
Vouchers Endpoint
O endpoint
vouchersinterage com o modelo Vouchers. Seu uso básico é obter dados de espécimes de Coleções Biológicas
Parâmetros GET
id = listretorna apenas vouchers com o id ou ids fornecidos (exid = 1,2,3,10)numero = stringretorna apenas vouchers para o número do coletor informado (mas é uma string e pode conter caracteres não numéricos)collector = mixedum dos id ou ids ou abreviações, retorna apenas vouchers para o coletor principal informadodataset = list- lista de id ou ids, ou nomes do conjuntos de dados, retorna todos os vouchers relacionados aos conjuntos de dados informados, incluindo vouchers associados indiretamente (por exemplo, vouchers de medições ou de arquivos de mídia nos conjuntos de dados informados)project = mixedum de ids ou nomes, retorna apenas os vouchers para o projeto informado.location = mixedum de ids ou nomes; filtra vouchers diretamente vinculados à(s) localidades(s)location_root = mixed- igual a location, mas inclui também os vouchers para as localides descendentes das localidades informadas. por exemplo. “location_root = Manaus” para obter qualquer voucher coletado dentro da área administrativa de Manaus, enquanto “location=Manaus” retorna apenas indivíduos cuja localidade é Manaus, mas indivíduos ligados a outras localidades dentro do município..;individual=mixedid ou lista de ids, ou organismID(s) de indivíduos, retorna apenas vouchers para os indivíduos informadostaxon = mixedum de ids ou nomes, retorna apenas vouchers para os táxons informados.taxon_root = mixed- igual a taxon, mas incluirá vouchers para os descendentes dos táxons informados. por exemplo. “taxon_root = Lauraceae” para obter qualquer voucher de Lauraceae e “taxon=Lauraceae” para obter vouchers identificados no nível de família.
Note
Observe que todos os campos de pesquisa (táxon, localidades e conjunto de dados) podem ser especificados como nomes (por exemplo, “taxon = Euterpe edulis”) ou como ids de banco de dados. Se uma lista for especificada para um desses campos, todos os itens da lista devem ser do mesmo tipo, ou seja, você não pode pesquisar por ’taxon = Euterpe, 24’. Além disso, location e taxon têm prioridade sobre location_root e taxon_root se ambos forem informados.Campos de resposta
id- o id do voucher na table vouchers (id local da instalação)basisOfRecordDWC - será sempre ‘preservedSpecimen’ [dwc location](DWCoccurrenceIDDWC - o identificador único do voucher que combina collectionCode+catalogNumber+organismIDorganismIDDWC -o identificador textual do Indivíduo ao qual o Voucher pertenceindividual_id- o id do Indivíduo na tabela individuals (id local)collectionCodeDWC - o acrônimo da Coleção Biológica onde o Voucher está depositadocatalogNumberDWC - o número de tombo (id) do Voucher na Coleção BiológicatypeStatusDWC - se o Voucher representa um tipo nomenclaturalrecordedByDWC - abbreviações de coletores separados por pipe “|”recordedByMain- a primeira pessoa listada em recordedBy, o coletor ao qual pertence o recordNumberrecordNumberDWC - o número do coletor ou identificador do Voucher.recordedDateDWC - a data de coleta (pode estar incompleta)scientificNameDWC - o nome científico do Indivíduo ao qual o voucher pertence.scientificNameAuthorshipDWC - o autor do nome taxonomico. Para taxonomicStatus unpublished será o nome da Pessoa que criou o nome não publicado.familyDWCgenusDWCidentificationQualifierDWC - modificadores do nome para a identificação atribuída: cf. aff. s.l., etc.identifiedByDWC - Pessoa que identificoudateIdentifiedDWC - data da identificaçãoidentificationRemarksDWC - notas de identificacaolocationName- nome da localidade em que o indivíduo estava quando foi coletado (quando o indivíduo tem mais de uma localidade, a localidade da data mais próxima)higherGeographyDWC - o caminho completo até LocationName ‘|’ separated (e.g. Brasil | Amazonas | Rio Preto da Eva | Fazenda Esteio | Reserva km37);decimalLatitudeDWC - depende do tipo de localidade e dos atributos de posição relativa do Indivíduo ao qual o Voucher pertence. Se o indivíduo tiver vários locais (uma ave monitorado), o local mais próximo da data em que o voucher foi obtidodecimalLongitudeDWC - mesmo que para decimalLatitudeoccurrenceRemarksDWC - notas para este registroassociatedMediaDWC - urls para arquivos de mídia associados a este registrodatasetName- nome do Conjunto de Dados ao qual o indivíduo pertence DWCaccessRights- a política de acesso do Conjunto de dados - DWCbibliographicCitation- a citação para Conjunto de Dados - DWClicense- a licensa para o Conjunto de Dados - DWC
Jobs Endpoint
O endpoint
jobsinterage com o modelo [UserJobs] (/docs/concepts/data-access/#user-jobs). O uso básico é obter uma lista de trabalhos de importação, junto com uma mensagem de status e logs. Você também pode obter dados solicitados usando o parâmetrosave_job=1ou através da interface da web.
Parâmetros GET
id- id do job (id local)status = stringretorna apenas jobs para o status especificado: “Submitted”, “Processing”, “Success”, “Failed” ou “Cancelled”;get_file- seget_file=1, então você pode obter o arquivo de dados salvo por um job. Isso também precisa de um único parâmetroid. Útil, ao obter dados com a opçãosave_job=1.
Campos de resposta
id- id do job (id local)status- o status do trabalho:” Enviado “,” Processando “,” Sucesso “,” Falha “ou” Cancelado “;dispatcher- o tipo de trabalho, por exemplo, ImportTaxons;log- as mensagens de log do job, geralmente indicando se os recursos foram importados com sucesso ou se ocorreram erros; outros.
Possíveis erros
Esta é uma pequena lista de códigos de erro que você pode receber ao usar a API. Se você receber qualquer outro código de erro, envie um issue para o repositório.
- Error 401 - Unauthenticated. Currently not implemented. You may receive this error if you attempt to access some protected resources but didn’t provide an API token.
- Error 403 - Unauthorized. You may receive this error if you attempt to import or edit some protected resources, and either didn’t provide an API token or your user does not have enough privileges.
- Error 404 - The resource you attempted to see is not available. Note that you can receive this code if your user is not allowed to see a given resource.
- Error 413 - Request Entity Too Large. You may be attempting to send a very large import, in which case you might want to break it down in smaller pieces.
- Error 429 - Too many attempts. Wait one minute and try again.
- Error 500 - Internal server error. This indicates a problem with the server code. Please file a bug report with details of your request.
3.3 - Inserir dados - POST
- O pacote OpenDataBio-R é um cliente para esta API.
- Importar dados de arquivos (planilhas) pela interface também requer utilizar os Parâmetros POST listados nesta página
- Importação em lote de Referências Bibliográficas e Arquivos de Mídia é possível apenas através da interface.
POST Individuals
Campos para importar indivíduos:
collector = mixed- obrigatório - pessoas - pode ser ‘id’, ‘abbreviation’, ‘full_name’, ’email’; se houver várias pessoas, separe os valores em sua lista com a barra vertical|ou;porque vírgulas podem estar presentes nos nomes. O coletor principal é o primeiro da lista;tag = string- obrigatório - o seu código ou número para o indivíduo (número da placa, número de coletor, número de anilha, etc)dataset = mixed- obrigatório - nome ou id do Conjunto de Dados para colocar o Indivíduodate = AAAA-MM-DD ou matriz- data em que o indivíduo foi coletado/etiquetado - para registros históricos pode-se informar uma string incompleta no formato “1888-05-NA” ou “1888-NA-NA"quando dia ou mês são desconhecidos. Pode-se informar também como array no formato “data = {‘year’: 1888, ‘month’: 5}”. OpenDataBio lida com datas incompletas, consulte o Modelo de dados incompletos. Pelo menosyearé obrigatório.notes=string- qualquer anotação para o Indivíduo, um texto simples ou dados em JSON;
Campos de localidade (uma ou mais localidades podem ser registradas para 1 indivíduo). Os campos possíveis são:
location- o nome ou id do local do Indivíduo obrigatório se a longitude e a latitude não forem informadaslatitudeelongitude- coordenadas geográficas em graus decimais; obrigatório se location não for informadoaltitude- a elevação do local onde o Indivíduo foi observado (em metros acima do nível do mar). Precisa ser um número inteiro;location_notes- qualquer nota para a localidade do Indivíduo, um texto simples ou dados em JSON;location_date_time- se diferente da data do indivíduo, uma data completa ou um valor de data + hora para a localidade do indivíduo. Obrigatório quando importando várias localidades.x- se a localidade for do tipo Parcela (adm_level=100) ou Transecto (adm_level=101), a coordenada x do indivíduo na localidade;y- se a localidade for do tipo Parcela (adm_level=100) ou Transecto (adm_level=101), a coordenada y do indivíduo na localidade;distance- se o localidade for do tipo PONTO, uma distância para a posição do indivíduo em relação ao PONTO (em metros). Útil, comanglepara, por exemplo, dados de Ponto Quadrante;angle- se a localidade for do tipo PONTO, o azimute da posição do Indivíduo em relação à localidade;
Campos de identificação. A identificação taxonômica não é obrigatória, podendo ser informada de duas formas distintas: (1) identificação-própria - o indivíduo pode ter sua própria identificação; ou (2) identificação-dependente a identificação é a mesma que a de outro indivíduo (por exemplo, quando múltiplos indivíduos tem sua identificação associada a um único indivíduo para o qual existe um voucher num Biocoleção)
- Os seguintes campos podem ser usados para a
identificação-própriado indivíduo:taxon = mixed- nome ou id do táxon identificado, por ex. ‘Ocotea delicata’ ou seu ididentifier = mixed- uma ou mais pessoas responsável(is) pela identificação taxonômica. Pode informar ‘id’, ‘abbreviation’, ‘full_name’ ou ’email’. Se houver várias pessoas, separe os valores em sua lista com a barra vertical|ou;porque vírgulas podem estar presentes nos nomes.identification_dateouidentification_date_year,identification_date_month, e/ouidentification_date_day- completo ou incompleto. Se vazio, a data de coleta do indivíduo será usada.modifier- nome ou número do modificador do nome da identificação. Valores possíveis ’s.s.’ = 1, ’s.l.’ = 2, ‘cf.’ = 3, ‘aff.’ = 4, ‘vel aff.’ = 5, o padrão é 0 (nenhum).identification_notes- quaisquer notas de identificação, um texto simples ou dados em JSON;identification_based_on_biocollection- o nome ou id da Biocoleção se a identificação for baseada em um espécime de referência depositado em uma Biocoleção (registrada ou não na base)identification_based_on_biocollection_id- somente preencher seidentification_based_on_biocollectionestiver preenchido;
- Para uma
identificação-dependenteidentification_individual- id ou nome completo (organimsID) do Indivíduo que possui a identificação.
Se o Indivíduo tiver Vouchers com os mesmos Coletores, Data e Número do Coletor (Tag) que os do Indivíduo, os seguintes campos e opções permitem armazenar os vouchers durante a importação do registro do Indivíduo (como alternativa, você pode importar o voucher após a importação indivíduos através do Voucher EndPoint. Os vouchers para o indivíduo podem ser informados de duas maneiras:
- Ou como campos separados:
biocollection- uma string com um único valor ou uma lista de valores separados por vírgulas. Os valores podem ser os valoresidouacronymdo Modelo de Biolcoleções. Ex: “{‘biocollection’: ‘INPA; MO; NY’}” ou “{‘biocollection’: ‘1,10,20’}”;biocollection_number- uma string com um único valor ou uma lista de valores separados por vírgulas com o BiocollectionNumber para o voucher individual. Se for uma lista, então deve ter o mesmo número de valores quebiocollection;biocollection_type- Uma string com um único valor de código numérico ou uma lista de valores separados por vírgulas para Tipo Nomenclatural para os Vouchers. O valor padrão é 0 (não é um tipo).
- Como um único campo
biocollectioncontendo uma matriz com cada elemento tendo os campos acima para uma única BioColeção: “{ { ‘biocollection_code’ : ‘INPA’, ‘biocollection_number’ : 59786, ‘biocollection_type’ : 0}, { ‘biocollection_code’ : ‘MG’, ‘biocollection_number’ : 34567, ‘biocollection_type’ : 0} }”
POST Individual-locations
O endpoint individual-locations permite a importação de várias localidades para Indivíduos previamente registrados. Projetado para importação de múltiplas ocorrências de organismos que se movem.
Possible fields are:
individual- o id do Indivíduo obrigatóriolocation- o nome ou id do local do Indivíduo obrigatório se a longitude e a latitude não forem informadaslatitudeelongitude- coordenadas geográficas em graus decimais; obrigatório se location não for informadoaltitude- a elevação do local onde o Indivíduo foi observado (em metros acima do nível do mar);location_notes- qualquer nota para a localidade do Indivíduo, um texto simples ou dados em JSON;location_date_time- se diferente da data do indivíduo, uma data completa ou um valor de data + hora para a localidade do indivíduo. Obrigatório quando importando várias localidades.x- se a localidade for do tipo Parcela (adm_level=100) ou Transecto (adm_level=101), a coordenada x do indivíduo na localidade;y- se a localidade for do tipo Parcela (adm_level=100) ou Transecto (adm_level=101), a coordenada y do indivíduo na localidade;
POST Locations
O endpoint
locationsinterage com o modelo Localidades. Use para importar novas localidades.
Atenção
As localidades são armazenadas com um relacionamento pai-filho, garantindo validações e facilitando consultas. Os pais serão adivinhados usando a geometria da localidade sendo importada. Se o pai não for informado, o local importado deve estar completamente contido por um pai registrado (a função sql ST_WITHIN será usada para detectar o pai). No entanto, se um pai for informado, a importação também pode testar se a geometria se ajusta a uma versão em buffer da geometria da localidade pai, ignorando assim a sobreposição de geometrias secundárias e bordas compartilhadas. Os países podem ser importados sem relações com os pais. Qualquer outro local deve ser registrado em pelo menos um ‘país’ como pai. Se o registro for marinho e estiver fora do polígono de um país registrado, um argumento ‘ismarine’ deve ser indicado para aceitar o relacionamento não espacial com a localidade.
Subparcelas é a única situação em que a geometria não é necessária. Se não for informado, a geometria será calculada com base na geometria da Parcela pai e com as coordenadas startx e starty da subparcela.
Certifique-se de que sua projeção geométrica seja EPSG: 4326 WGS84. Use este padrão!
Campos POST disponíveis:
nome- o nome da localidade - obrigatório (pai + nome deve ser único no banco de dados)adm_level- deve ser numérico, veja aqui - obrigatório- geometria obrigatório, pode usar:
geompara uma representação WKT da geometria, POLYGON, MULTIPOLYGON, POINT OU LINESTRING permitido;latelongpara latitude e longitude em graus decimais (use números negativos para sul/oeste).
altitude- em metros, se for o caso.datum- o padrão é ‘EPSG: 4326-WGS 84’ e você é fortemente encorajado a importar apenas dados nesta projeção. Você pode informar uma projeção diferente aqui;parent- o id ou o nome do local pai. A API detectará o pai com base na geometria informada e o pai detectado tem prioridade se for diferente do parent informado. No entanto, apenas quando os pais são informados, a validação também testará se sua localização cai dentro de uma versão em buffer do pai informado, permitindo importar locais que têm um relacionamento pai-filho, mas suas fronteiras se sobrepõem de alguma forma (fronteiras compartilhadas ou diferenças no georreferenciamento) ;- quando a localização é PARCELA (
adm_level = 100), os campos opcionais são:xeypara as dimensões da parcela em metros (define as coordenadas cartesianas)startxestartypara a posição inicial de uma sub-parcela em relação à localização da parcela pai;
notes- qualquer nota que você deseja adicionar à sua localidade, um texto simples ou dados em JSON;azimuth- aplica-se apenas para Parcelas e Transectos e quando registrados com uma geometria de tipo PONTO - o azimute será usado para construir a geometria. Para Parcelas, as coordenadas do ponto referem-se ao vértice 0,0 do polígono que será construído no sentido horário a partir do ponto informado, do azimute e a dimensão y. Para transectos, as coordenadas do ponto informado são o ponto inicial e uma linha será construída usando este azimute e a dimensão x.ismarine- para permitir a importação de registros de localidade que não se enquadram em nenhum registro de localidade pai, você pode adicionarismarine=1. Observe, no entanto, que isso permite importar locais sem validação espacial. Use somente se sua localização for realmente um local marítimo que esteja fora da fronteira de qualquer país registrado.
alternatively: you may just submit a single column named geojson containing a Feature record, with its geometry and having as ‘properties’ at least tags name and adm_level (or admin_level). See geojson.org. This is usefull, for example, to import country political boundaries (https://osm-boundaries.com/).
alternativamente: você pode apenas enviar uma única coluna chamada geojson contendo um registro Feature, com sua geometria e tendo como ‘propriedades’ pelo menos as tags name e adm_level (ou admin_level). Consulte geojson.org. Isso é útil, por exemplo, para importar as fronteiras políticas e administrativas de um país usando (https://osm-boundaries.com/).
POST Measurements
O endpoint measurements permite importar medições.
Os seguintes campos são permitidos para importar medições
dataset = number- o id do Conjunto de Dados onde a medição deve ser colocada obrigatóriodata = AAAA-MM-DD- a data de observação para a medição, deve ser passada como AAAA-MM-DD obrigatórioobject_type = string- ‘Individual’,‘Location’,‘Taxon’ or ‘Voucher’, ou seja, o objeto ao qual a medição pertence obrigatórioobject_type = number- o id do objeto medido, seja (individuals.id, locations.id, taxons.id, vouchers.id) obrigatórioperson = números ou strings- uma ou mais pessoas que mediram o objeto. Pode ser ‘id’, ‘abbreviation’, ‘full_name’, ’email’; se houver várias pessoas, separe os valores em sua lista com a barra vertical|ou;porque vírgulas podem estar presentes nos nomes. obrigatóriotrait_id = número ou string- o id ou export_name para a variável medida. obrigatóriovalue = número, lista de strings- isso dependerá do tipo de variável obrigatório, opcional para variável de tipo LINK**link_id = number- o id do objeto vinculado para uma variável do tipo Link obrigatório para variáveis do tipo Link DEPRECADO SUBSTITUIDO POR locationlocation ou location_id- o id ou nome da Localidade de uma medição relacionada a um Taxon. Apenas Taxons podem ter um location também associado. Isso substitui a lógica do Trait de tipo Link.bibreference = number- o id ou bibkey de uma referência para a medição. Deve ser usado quando a medição foi tirada de uma publicaçãonotes- qualquer nota que você desejar. Este é um local útil para armazenar informações relacionadas à medição. Por exemplo, ao medir 3 folhas de um voucher, você pode indicar aqui a qual folha a medição pertence, folha1, folha2, etc. permitindo vincular medidas de diferentes variáveis por este campo. Pode ser um texto simples ou dados em JSON;duplicated- por padrão, a API de importação evitará medições duplicadas para a mesma variável, mesmo objeto medido e mesma data. Pode importar especificandoduplicado=#, onde # é o número armazenado de registros+1. Por exemplo, se já existe um registro, deve-se informarduplicado=2se houver dois, um terceiro pode ser armazenado utilizandoduplicado=3e assim por diante.parent_measurement = números apenas- o ‘id’ de outra medição com a qual a medição sendo importada tem dependência. Isso cria uma relação entre as medições. O id será validado e deve ser de uma medição pertencente ao mesmo objeto, mesma data e variável diferente. Por exemplo. a ’largura da folha’ pode estar ligada a uma medida do ‘comprimento da folha’.
POST Persons
O endpoint persons interage com a tabela Person.
Os seguintes campos são permitidos ao importar pessoas usando a API:
full_name- nome completo da pessoa, obrigatórioabreviação- nome abreviado, conforme usado pela pessoa nas publicações, como coletor, etc. (se deixado em branco, uma abreviação padrão será gerada usando o atributo full_name - a abreviação deve ser única dentro de uma instalação OpenDataBio);email- um endereço de e-mail,instituição- a que instituição esta pessoa está associada;biocollection- nome ou acrônimo da BioColeção à qual esta pessoa está associada (se for um taxonomista, por exemplo)
POST Taxons
Use para importar novos nomes taxonômicos.
- O POST API requer APENAS o nome completo do táxon a ser importado, ou seja, para espécies ou infra-espécies, o nome completo deve ser informado, por exemplo, Ocotea guianensis ou Licaria cannela aremeniaca, sem indicação dos ranks.
- O script validará o nome, recuperando as informações necessárias restantes através de serviços de API de repositórios nomenclaturais. Ele pesquisará GBIF, Tropicos.org, Zoobank e se encontrar recuperará os dados do nome, os ids nesses repositórios e também o caminho de classificação completo até Reino. Se o esta busca indicar que o nome é um sinônimo, os nomes válidos e aceitos para o seu nome serão também detectados.E tudo isso será importando na base de dados juntamente com seu nome. Se seu nome for reconhecido com sinônimo, ele será inserido como nome
invalid. O caminho da classificação hierárquica será importado até que um Taxon pai já registrado seja encontrado. Note, no entanto, que você pode não querer a classificação hierárquica, ou querer uma diferente. Neste caso indiqueleveleparent. - Portanto, a menos que você esteja tentando importar nomes não publicados, apenas envie o parâmetro de
nameda lista abaixo.
Campos possíveis:
name- nome completo do táxon obrigatório, por exemplo “Ocotea floribunda” ou “Pagamea plicata glabrescens”level- pode ser o id numérico ou uma string que descreve o taxonRank recomendado para nomes não publicadosparent- o nome completo ou id do pai do táxon - nota - se você informar um pai válido e o sistema detectar um pai diferente através da API aos repositórios nomenclaturais, será dada preferência ao pai informado; obrigatório para nomes não publicadosbibreference- a referência bibliográfica em que o táxon foi publicado;author- o nome do autor do táxon;author_idouperson- o nome da pessoa registrada, abreviatura, e-mail ou id, representando o autor de nomes não publicados - obrigatório para nomes não publicadosvalid- booleano, verdadeiro se o nome do táxon for válido; 0 ou 1mobot- id do Tropicos.org para este táxonipni- id do IPNI para este táxonmycobank- id do MycoBank para este táxonzoobank- id do ZOOBANK para este táxongbif- nubKey de GBIF para este táxon
POST Traits
Ao inserir algumas variáveis, recomendamos fortemente que você insira variáveis uma a uma usando o formulário da Interface Web, o que reduz a chance de duplicar definições de variáveis, que são objetos compartilhados com todos os usuários da base e devem ter uma definição clara.
O endpoint traits interage com o modelo Variáveis. O método POST permite que você importe variáveis em lote para o banco de dados e é projetado para transferir Ontologias para o OpenDataBio de outros sistemas
- Conforme observado na descrição do modelo Variáveis, é importante verificar se uma Variável que você precisa já não está cadastrada e evitar a multiplicação de características redundantes. A interface no navegador facilita esse processo. Por meio da API, OpenDataBio verifica por duplicações apenas em
export_name, que deve ser único no banco de dados. Observe, no entanto, que as Variáveis também devem ser o mais específicas possível para anotações detalhadas de metadados. - Variáveis usam Traduções de Usuário para nomes e descrições, permitindo traduções nos idiomas disponíveis na sua instalação.
Campos permitidos para POST traits:
export_name = string- um nome curto para a variável, que será usado durante as exportações de dados e nos formulários na interface. export_name deve ser único na base de dados e não devem ter tradução. Nomes de exportação curtos no estilo [CamelCase](https://en.wikipedia.org/wiki/Camel_case) são recomendados. Evite sinais diacríticos (acentos), caracteres especiais, pontos e até mesmo espaços em branco obrigatóriotype = number- um código numérico que especifica o tipo de Variável. Veja o modelo Variáveis para uma lista completa. obrigatórioobjects = list- uma lista dos Core objects para os quais a variável pode ser medida. Os valores possíveis são ‘Individual’, ‘Voucher’, ‘Location’ e/ou ‘Taxon’, singular e sensível a maiúsculas e minúsculas. Ex: “{‘object’: ‘Individual,Voucher’}”; obrigatórioname = json- veja abaixo sobre traduções; obrigatóriodescription = json- veja abaixo sobre traduções; obrigatório Campos específicos para tipos de variáveis:unit=string- necessário apenas para características quantitativas (a unidade de medida). Deve ser um código ou um nome (em qualquer idioma) de uma unidade já armazenada no banco de dados. As unidades só podem ser definidas através da interface web.range_min = number- opcional para variáveis quantitativas. especifique o valor mínimo permitido para uma medição.range_max = number- opcional para quantitativo. valor máximo permitido para a variável.categories = json- obrigatório para variáveis categóricas e ordinais; veja abaixo sobre traduçõeswavenumber_minewavenumber_max- obrigatório para variáveis espectrais = número de onda mínimo e máximo dentro do qual os valores de absorbância ou refletância de ‘value_length’ estão igualmente distribuídos. Pode ser informado emrange_minerange_max, prioridade para o prefixo wavenumber sobre range se ambos informados.value_length- obrigatório para características espectrais = número de valores no espectrolink_type- obrigatório para características de link - a classe do tipo de link, nome completo ou nome de base: por exemplo. ‘Taxon’ ou ‘App\Models\Taxon’.
bibreference=mix- o(s) id(s) ou bibkey(s) de uma BibReference já armazenada(s) no banco de dados. Se mais de uma, separados por ‘|’ ou ‘;’parent- id ou export_name de outra variável da qual a variável depende. Se você indicar uma variável aqui, isso significa que você adiciona uma RESTRIÇÃO na validação das medições. Adicionar uma Medida para a variável sendo importada DEPENDERÁ de o banco de dados possuir uma medida para a variável aqui indicada, para o mesmo objeto e mesma data. Exemplo, você cria uma característica chamada POM (ponto de medição) para registrar a altura em uma árvore onde você mede um DAP (diâmetro à altura do peito). Adicionar DAP como uma característica da qual o POM depende significa que você só pode adicionar POM se houver um valor de DAP para a mesma árvore na mesma data.
Categorias e Traduções
-
Os campos
name,descriptiondevem ter a seguinte estrutura para incorporar Traduções do usuário. Eles devem ser uma lista com o idioma como ‘chaves’. -
Idiomas válidos podem ser obtidos pelo Language API.
-
Por exemplo, um campo
namepode receber a seguinte informação:- usando o código de idioma como chaves:
[ {"en": "Diâmetro na altura do peito"," pt-br": "Diâmetro a Altura do Peito"} ]- ou usando os ids de idioma como chaves:
[ {"1":"Diâmetro à altura do peito","2":"Diâmetro a Altura do Peito"} ]- ou usando os nomes dos idiomas como chaves:
[ {"English":"Diameter at Breast Height","Portuguese": "Diâmetro a Altura do Peito"} ] -
Alternativamente, você pode adicionar as informaçõe como parâmetros separados. Ao invés de
namepode usarname.IDIOMA_CODIGO_ou_ID, por exemploname.enouname.1para o nome em inglês ename.pt-brouname.2para o nome em português. Da mesma forma paradescription:description.enoudescription.1, etc. -
O campo
categoriesdeve incluir para cada categoria + ordem + idioma os seguintes campos:lang = mixed- o id, código ou nome do idioma da tradução, obrigatórioname = string- o nome da categoria no idioma obrigatório (name + rank + lang deve ser único)rank = number- a ordem para variáveis ordinais, semi-quantitativas; para não ordinal, oranktambém é importante, mas apenas para indicar a mesma categoria em todos os idiomas obrigatóriodescription = string- opcional para categorias, uma definição da categoria.- Exemplo para categoria:
[ {"lang":"en","rank":1,"name":"small","description":"smaller than 1 cm"}, {"lang":"pt-br","rank":1,"name":"pequeno","description":"menor que 1 cm"} {"lang":"en","rank":1,"name":"big","description":"bigger than 10 cm"}, {"lang":"pt-br","rank":1,"name":"grande","description":"maior que 10 cm"}, ]
POST Vouchers
O endpoint
vouchersinterage com a tabela Vouchers. Note quevoucherspodem também ser importados diretamente através da importação de registros de indivíduos. O POST vouchers requer que informações de indivíduos já esteja cadastradas.
Os seguintes campos são permitidos na importação de dados:
individual = mixed- o id numérico ou organismID do indivíduo ao qual o voucher pertence obrigatório;biocollection = mixed- o id, nome ou acrônimo de uma BioColeção registrada a que o Voucher pertence obrigatório;biocollection_type = mixed- o nome ou código numérico de [tipo de nomenclatural](/docs/api/quick-reference /#nomenclature-types), se for o caso. O padrão é 0, ou seja, não é um tipo nomenclatural.biocollection_number = mixed- o código alfanumérico do voucher na BioColeção (número de tombo ou outro código de uso local);number = string- o número do coletor principal - somente se diferente do valor tag do indivíduo ao qual o voucher pertence;collector = mixed- ids ou abreviações de pessoas. Quando vários valores são informados, o primeiro é o coletor principal. Informe apenas se for diferente da lista de coletores do Indivíduo associado.date = AAAA-MM-DD ou array- necessário apenas se, com coletor e número, for diferente dos valores do Indivíduo associado. A data pode ser um Modelo Incompleto.dataset = number- herda o Conjunto de Dados ao qual o indivíduo pertence, mas você pode fornecer um Conjunto de Dados diferente se preferirnotes = string- qualquer anotação, um texto simples ou dados em JSON;
Campo Notes
O campo notes de qualquer modelo é para texto simples ou um texto formatado como objeto JSON contendo dados estruturados. A opção Json permite que você armazene dados estruturados personalizados em qualquer modelo que tenha o campo notes. Você pode, por exemplo, armazenar como notas alguns campos secundários de fontes originais ao importar dados, mas pode armazenar quaisquer dados adicionais que não sejam fornecidos pela estrutura do banco de dados do OpenDataBio. Esses dados não serão validados pelo OpenDataBio e a padronização de tags e valores depende de você. As notas Json serão importadas e exportadas como um texto JSON e serão apresentadas na interface como uma tabela formatada; URLs em seu Json serão apresentados como links nessa tabela.
3.4 - Atualizar dados - PUT
Atenção
Somente os endpoints listados abaixo podem ser atualizados usando a API e somente os campos PUT listados podem ser atualizados em cada endpoint. Os valores dos campos são os mesmos como explicados para a API POST, exceto que em todos os casos oID do registro a ser atualizado também deve ser fornecido.
| Endpoint | Description | PUT Fields |
|---|---|---|
| individuals | Atualizar Indivíduos | (id or individual_id),collector, tag, dataset, date, notes , taxon , identifier , identification_date , modifier, identification_notes , identification_based_on_biocollection, identification_based_on_biocollection_id , identification_individual |
| individual-locations | Atualizar Localidades de Indivíduos | (id or individual_location_id), individual, (location or latitude + longitude), altitude, location_notes, location_date_time, x, y, distance, angle |
| locations | Atualizar Localidades | (id or location_id), name, adm_level, (geom or lat+long) , parent, altitude, datum, x, y , startx, starty, notes, ismarine |
| measurements | Atualizar Medições | (id or measurement_id), dataset, date, object_type, object_type, person, trait_id, value, link_id, bibreference, notes, duplicated,parent_measurement |
| persons | Atualizar Pessoas | (id or person_id),full_name, abbreviation, email, institution, biocollection |
| vouchers | Atualizar Vouchers | (id or voucher_id),individual, biocollection, biocollection_type, biocollection_number, number, collector, date, dataset, notes |
4 - Modelo conceitual
Se você quiser ajudar no desenvolvimento, leia com atenção o modelo conceitual do OpenDataBio antes de começar a colaborar.
Para facilitar a compreensão dos conceitos, já que incluem muitas tabelas e relacionamentos complexos, o modelo de dados OpenDataBio está aqui dividido nas quatro categorias listadas abaixo.
4.1 - Objetos Centrais
Os objetos centrais são: Localidades, Vouchers, Indivíduos e Taxons. Essas entidades são consideradas “centrais” porque podem receber Medições, ou seja, você pode registrar valores para qualquer Variável.
-
O objeto Indivíduo refere-se a um organismo individual que foi observado uma vez (uma ocorrência) ou foi marcado para monitoramento, como uma árvore em uma parcela permanente, uma ave anilhada, um morcego rastreado por rádio. Os indivíduos podem ter um ou mais Vouchers em uma BioColeção e um ou vários Localidades e terão uma Identificação taxonômica. Qualquer atributo medido ou tomado para um individuo pode ser associado a este objeto por meio do modelo Medição.
-
O objeto Vouchers é para registros de espécimes coletados de Indivíduo e depositados em uma BioColeção. A identificação taxonômica e a localização de um voucher é aquela do próprio indivíduo a que pertence. Medições podem ser vinculadas a um Voucher quando você deseja registrar explicitamente os dados para aquela amostra específica (por exemplo, medições morfológicas; um marcador molecular de uma extração de uma amostra em um coleta de tecido). Caso contrário, você pode simplesmente registrar a medição para o indivíduo ao qual o voucher pertence. O modelo de voucher também está disponível como tipo especial de Variável, o
LinkType, tornando possível registrar contagens para o táxon do voucher em um determinado local. -
O objeto Localidades contém geometrias espaciais, como pontos e polígonos, e inclui
parcelasetransectoscomo casos especiais. Um Indivíduo pode ter uma localidade (por exemplo, uma planta) ou multiplas localidades (por exemplo, um animal monitorado). As localidades do tipo PARCELA e TRANSECTO podem ser registradas como geometria espacial ou apenas com geometria de ponto, e podem ter dimensões cartesianas (metros) registradas. Os indivíduos também podem ter posições cartesianas (X e Y ou angle e distance) em relação à sua Localidade, permitindo contabilizar o mapeamento tradicional de indivíduos em unidades de amostragem. Medições ecológicas relevantes, como dados de solo ou clima, são exemplos de Medições que podem ser vinculadas a localidades. -
O objeto Taxon, além de seu uso para a Identificação taxonômica de Indivíduos, pode receber Medições, permitindo a organização de dados secundários publicados ou qualquer tipo de informação ligada a um nome taxonômico. Uma Referência Bibliográfica pode ser incluída para indicar a fonte de dados. Além disso, o modelo Taxon está disponível como tipo especial de Variável, o
LinkType, tornando possível registrar contagens de Taxons em um determinado local.
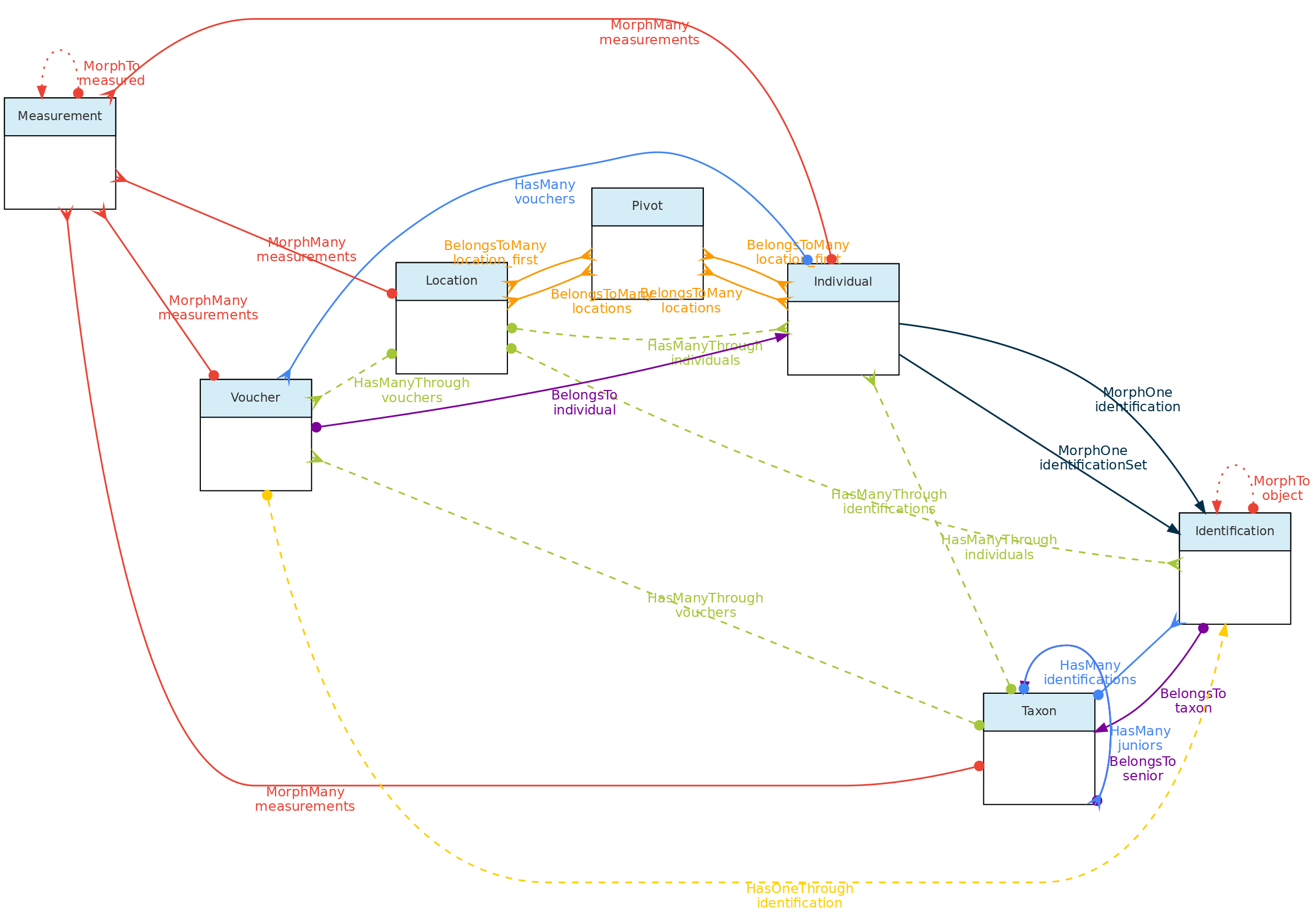
Esta figura mostra as relações entre os objetos centrais e com o modelo Medições. O Identificação também está incluído para maior clareza. Links sólidos são relacionamentos diretos, enquanto links tracejados são relacionamentos indiretos (por exemplo, os táxons têm muitos vouchers por meio de indivíduos e muitos indivíduos por meio de identificações). As linhas sólidas vermelhas vinculam os Objetos centrais com o modelo de Medição por meio de relações polimórficas. As linhas pontilhadas no modelo de Medição apenas permitem o acesso ao objeto central medido e aos modelos de variáveis do tipo de link.
Localidades
O modelo Localidades armazena dados que representam locais do mundo real. Eles podem ser países, cidades, unidades de conservação ou qualquer polígono espacial, ponto ou trilha na superfície da Terra. Esses objetos são hierárquicos e possuem um relacionamento pai-filho implementado pelo Nested Set Model para dados hierárquicos da biblioteca Laravel Baum que facilita tanto a validação quanto as consultas.
Tipos de Localidades especiais são parcelas e transectos, que juntamente com pontos permitem diferentes métodos de amostragem usados em estudos de biodiversidade. Esses tipos de Localidades também podem ser vinculados a uma localidade administrativa pai e, além disso, a três tipos adicionais de localidades pai como Unidades de Conservação, Territórios Indígenas e qualquer camada Ambiental representando classes de vegetação, classes de solo , etc … com geometrias espaciais definidas.
Indivíduo
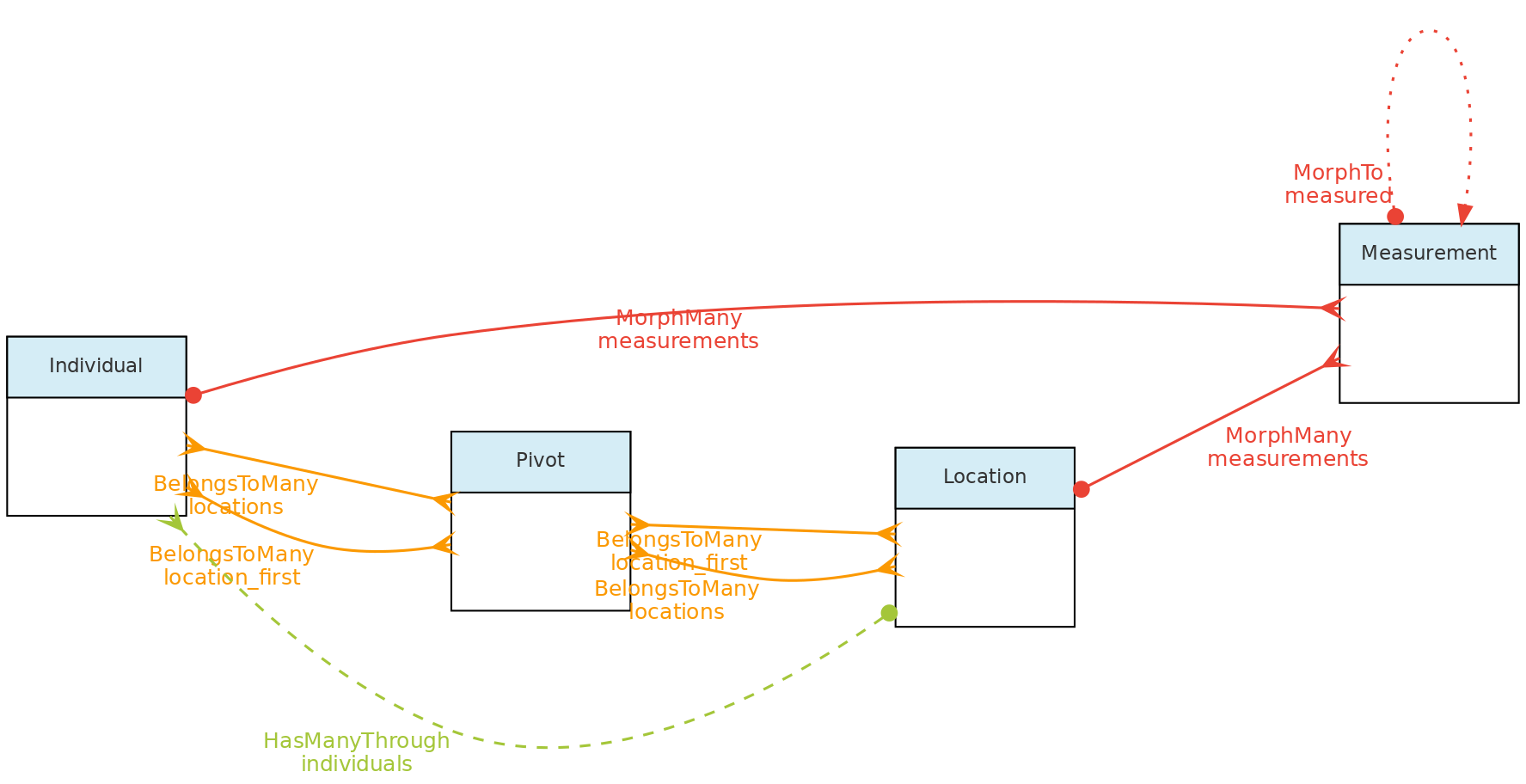 Esta figura mostra os relacionamentos do modelo
Esta figura mostra os relacionamentos do modelo Location através dos métodos implementados nas classes mostradas. A tabela dinâmica que vincula Localidade a Indivíduos permite que um indivíduo tenha vários locais e cada local para o indivíduo tenha atributos específicos como date_time, altitude, relative_position e notes.
 As mesmas tabelas relacionadas com o modelo
As mesmas tabelas relacionadas com o modelo Location com as relações diretas e não polimórficas indicadas.
Tabela locations
- As colunas
parent_idjunto comrgt,lftedephsão usadas para definir o modelo de conjunto aninhado para consultar ancestrais e descendentes de forma rápida. Apenasparent_idé especificado pelo usuário, as outras colunas são calculadas pela biblioteca Baum a partir dos valores id+parent_id que definem a hierarquia. O mesmo modelo hierárquico é usado para o Taxons, mas para locais há uma restrição espacial, ou seja, um filho deve estar dentro de uma geometria pai. - A coluna
adm_levelindica o nível administrativo, ou tipo, de localidade. Por padrão, os seguintesadm_levelsão configurados no OpenDataBio:2para o país,3para a primeira divisão dentro do país (província, estado),4para a segunda divisão (por exemplo, município), … atéadm_level = 10como áreas administrativas (o código do país é 2 para permitir a padronização com OpenStreeMaps, que é recomendado seguir se sua instalação incluir dados de diferentes países). Os níveis administrativos podem ser configurados em um OpenDataBio antes de importar quaisquer dados para o banco de dados, consulte o guia de instalação para obter detalhes sobre isso.99é o código para Unidades de Conservação - uma unidade de conservação é umalocationque pode estar vinculada a vários outros locais (qualquer local pode pertencer a uma única UC). Assim, uma localidade pode ter como pai um município e como uc a unidade de conservação a que pertence.98é o código para Territórios Indígenas - mesmas propriedades das Unidades de Conservação, mas tratadas separadamente apenas porque algumas UCs e TIs podem se sobrepor amplamente como é o caso da região amazônica97é o código para Camadas ambientais - mesmas propriedades das Unidades de Conservação e Territórios Indígenas, ou seja, podem ser vinculadas como localidade pai adicional a qualquer Ponto, Parcela ou Transecto e, portanto, seus indivíduos relacionados. Armazene polígonos e geometrias de multipolígonos representando classes ambientais, como unidades de vegetação, biomas, classes de solo, etc …100é o código paraparcelasesub-parcelas- as localidades de tipo parcelas podem ser registradas com geometria de ponto ou polígono, e também devem ter dimensões cartesianas associadas em metros. Se for uma localização de ponto, a geometria é definida a partir do ponto informado. As dimensões cartesianas de uma localidade de tipo parcela também podem ser combinadas com posições cartesianas de subparcelas (ou seja, uma localidade de parcela cujo pai também é uma localidade de parcela) e/ou de indivíduos dentro de tais parcelas, permitindo que indivíduos e subparcelas sejam mapeados dentro de uma subparcela sem especificações de geometria. Em outras palavras, se a geometria espacial da Parcela for desconhecida, ela pode ter como geometria um único ponto GPS ao invés de um polígono, mais suas dimensõesxey. Uma subparcela é uma parcela cuja localidade pai também é uma parcela, e deve consistir em um ponto marcando o início da subparcela mais suas dimensões cartesianas X e Y. Se a geometria do início da subplot for subparcela, ela pode ser armazenada como uma posição relativa à parcela pai usandostartxestarty.101para transectos - como parcelas, os transectos podem ser registrados tendo uma geometria LineString ou simplesmente uma única coordenada de Latitude e Longitude e uma dimensão. A dimensão cartesianaxpara transectos representa o comprimento em metros e é usada para criar linha (orientada para o Norte) quando apenas um ponto é informado. A dimensãoyé usada para validar os indivíduos como pertencentes à uma localidade do tipo transecto e representa a distância máxima da linha que um indivíduo deve cair para ser detectado naquele local.999para localidades ‘POINT’ como waypoints GPS - isto é para registro de qualquer ponto no espaço
- A coluna
datumpode registrar a propriedade do datum da geometria, se conhecida. Se deixado em branco, o local é considerado armazenado usando o datum WGS84. Porém, não há conversor embutido de outros tipos de dados. Portanto os mapas exibidos podem ficar incorretos se diferentes projeções forem usadas. Fortemente recomendado projetar dados como WSG84 para padronização. - A coluna
geomarmazena a geometria da localização no banco de dados, permitindo consultas espaciais em linguagem SQL, comodetecção das localidades pai. A geometria de um local pode serPOINT,POLYGON,MULTIPOLYGONouLINESTRINGe deve ser formatada usando Well-Known-Text representação geométrica da localidade. Quando um POLYGON é informado, o primeiro ponto dentro da string geométrica é privilegiado, ou seja, pode ser utilizado como referência para marcações relativas. Por exemplo, tal ponto será a referência para as colunasstartxestartyde uma subparcela. Portanto, para as geometriasplotetransect, importa qual ponto é listado primeiro na geometria WKT
Acesso a dados usuários completos podem registrar novas Localidades, editar detalhes de Localidades e remover registros de Localidades que não têm dados associados. As Localidades têm acesso público.
Indivíduos
O objeto Indivíduo representa um registro para um organismo individual. Pode ser uma única ocorrência no espaço-tempo de um animal, planta ou fungo, ou um indivíduo monitorado ao longo do tempo, como uma planta em parcela florestal permanente, ou um animal de captura-recaptura ou rádio-rastreamento.
Um Indivíduo pode ter um ou mais Vouchers representando amostras físicas do indivíduo armazenadas em uma ou mais BioColeção e pode ter um ou mais Localidades, representando o local ou locais onde o indivíduo foi registrado.
Indivíduos também podem ter uma Identificação taxonômica, que pode ser própria ou pode depender da identificação de outro indivíduo (identificação-dependente). A identificação é herdada por todos os Vouchers registrados para o Indivíduo. Portanto, os vouchers não têm sua identificação própria.
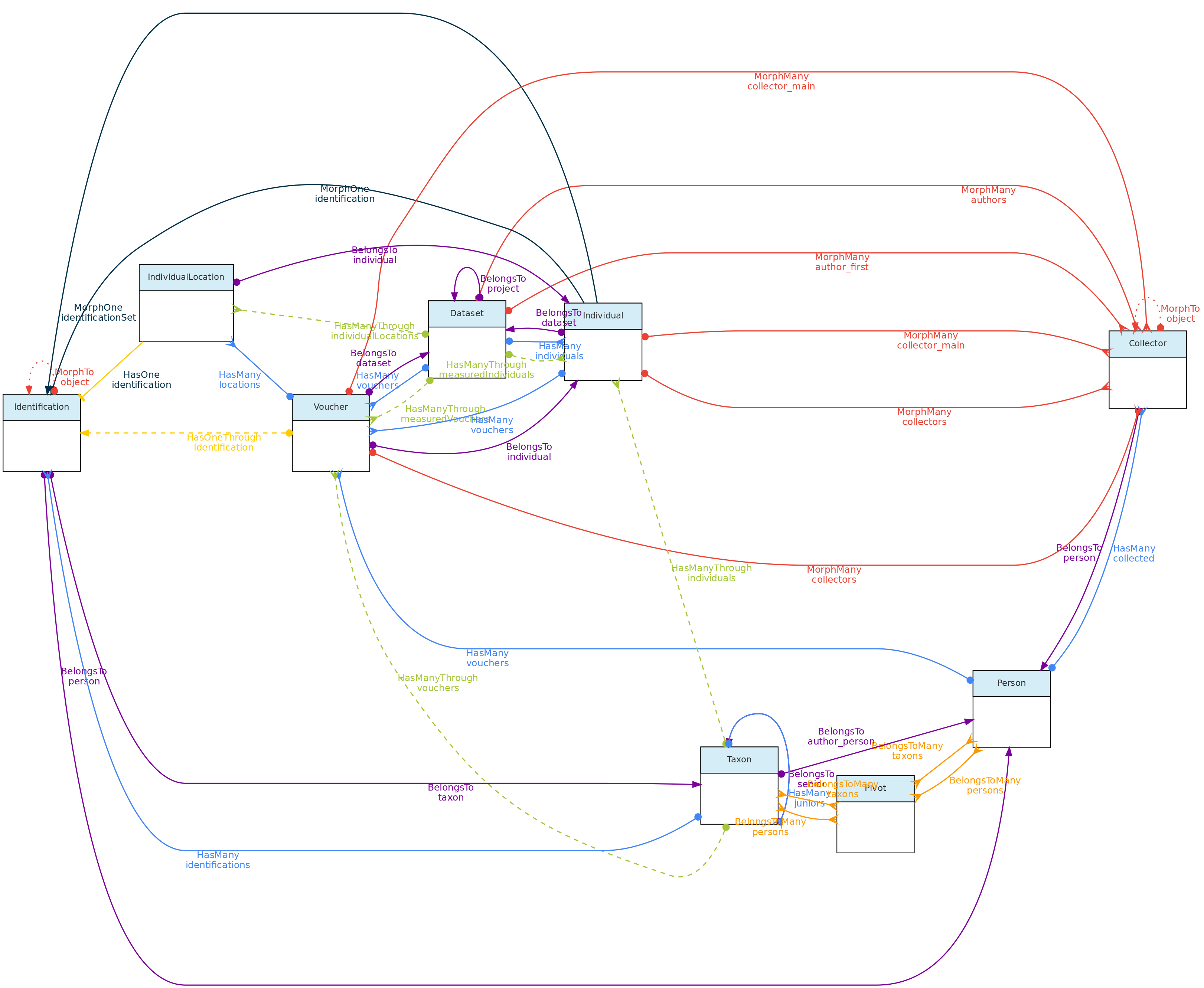 Esta figura mostra o Modelo Indivíduo e os modelos aos quais ele se relaciona, exceto os modelos de Medição e Localidades, já que seus relacionamentos com Indivíduos são mostrados em outra parte desta página. As linhas indicam os
Esta figura mostra o Modelo Indivíduo e os modelos aos quais ele se relaciona, exceto os modelos de Medição e Localidades, já que seus relacionamentos com Indivíduos são mostrados em outra parte desta página. As linhas indicam os métodos ou funções implementadas nas classes para acessar os relacionamentos. As linhas tracejadas indicam relações indiretas e as cores os diferentes tipos de métodos do Laravel Eloquent.
 As mesmas tabelas relacionadas com o modelo Indivíduo, suas colunas e as relações diretas e não polimóficas entre elas.
As mesmas tabelas relacionadas com o modelo Indivíduo, suas colunas e as relações diretas e não polimóficas entre elas.
Tabela individuals
- Um registro de um Indivíduo deve especificar pelo menos uma Localidade onde foi registrado, a
datedo registro, o identificador localtage ocollectorsdo registro e odatasetao qual o indivíduo pertence. - A localidade pode ser qualquer localidade cadastrado, independente do nível, permitindo armazenar registros históricos cujo georreferenciamento é apenas um local administrativo. Localidades são armazenadas na tabela
individual_location, tendo colunasdate_time,altitude,noteserelative_position. - A coluna
relative_positionarmazena as coordenadas cartesianas do Indivíduo em relação à sua localidade. Isso é apenas para indivíduos localizados em locais do tipoplot,transectoupoint. Por exemplo, uma parcela com dimensões de 100x100 metros (1ha) pode ter um indivíduo composição relativa = PONTO (50 50), que colocará o indivíduo no centro do local (isso é mostrado graficamente na interface da web conforme definido pelas coordenadasxeydo indivíduo). Se a localidade for uma subparcela, a posição dentro da parcela pai também pode ser calculada (isso foi projetado com as parcelas do ForestGeo em mente e é uma coluna na API individual GET. Se a localidade for um PONTO, a posição_relativa pode ser informada comoangle(= azimute) edistance, atributos frequentemente medidos em métodos de amostragem. Se a localidade for um TRANSECT, a posição_relativa posiciona o indivíduo em relação à linha, sendoxa distância ao longo do transecto a partir do primeiro ponto, e oya distância perpendicular onde o indivíduo está localizado, também levando em consideração alguns métodos de amostragem; - O campo
datenos modelos Indivíduo, Voucher, Medição e Identificação pode ser uma Data Incompleta, ou seja, apenas o ano ou ano + mês podem ser registrados. - A tabela Collector representa os coletores para um indivíduo ou voucher e está vinculada ao Modelo de pessoa. A tabela de coletores possui uma relação polimórfica com os objetos Voucher e Individual, definidos pelas colunas
object_ideobject_type, permitindo múltiplos coletores para cada registro individual ou voucher. O main_collector indicado é apenas o primeiro coletor listado para essas entidades. - O campo
tagé um código identificador para o indivíduo. Pode ser o número escrito na etiqueta de alumínio de uma árvore em uma parcela florestal, o número de uma anilha numa ave, ou onúmero de coletorde um espécime. A combinação demain_collector+tag+first_locationé restrita a ser única no OpenDataBio. A identificação taxonômica de um indivíduo pode ser definida de duas maneiras:- para identificação-própria um registro de Identificação taxonômica é criado na tabela de identifications e a coluna
identification_individual_idé preenchida com o próprioiddo indivíduo - para identificação-dependente, o id do Indivíduo que possui a Identificação é armazenado na coluna
identification_individual_id. - Conseqüentemente, o modelo Indivíduo contém dois métodos para se relacionar com o modelo de Identificação: um que define identificação-própria e outro que recupera as identificações taxonômicas usando a coluna
identification_individual_id.
- para identificação-própria um registro de Identificação taxonômica é criado na tabela de identifications e a coluna
- Os indivíduos podem ter um ou mais Vouchers depositados em uma BioColeção.
Acesso a dados Indivíduos pertencem a conjuntos de dados, então a política de acesso do conjuntos de dados se aplica aos indivíduos nele inseridos. Apenas os colaboradores e administradores do Conjunto de Dados podem inserir ou editar indivíduos, mesmo se o conjunto de dados for de acesso público.
Taxons
A ideia geral por trás do modelo Taxon é ferramentas para incorporar facilmente nomes taxonômicos válidos de repositórios Taxonômicos Online (atualmente Tropicos.org, GBIF e ZOOBANK estão implementados), mas permitindo a inclusão de nomes que também não são considerados válidos porque eles ainda são não publicados (por exemplo, um morfotipo), ou o usuário discorda da sinonímia publicada, ou o usuário deseja que todos os sinônimos sejam registrados como táxons inválidos no sistema. Além disso, permite definir um nível taxonômico de tipo
clado, permitindo armazenar, além das categorias de classificação taxonômica, qualquer nó da árvore da vida. Qualquer táxon registrado pode ser usado em identificações de Indivíduos e Medições podem ser vinculadas a nomes taxonômicos.
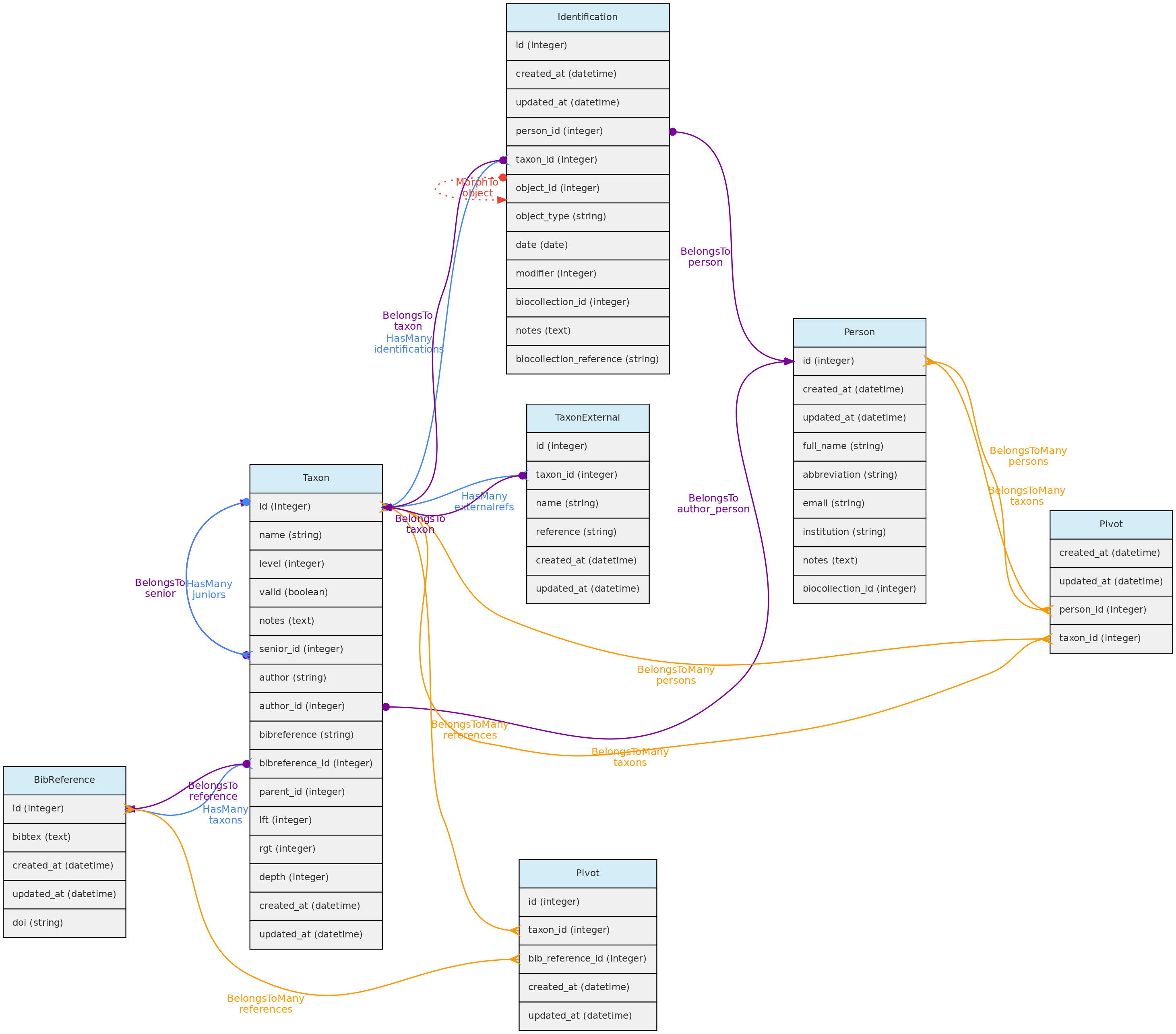 Modelo Taxon e suas relações. As linhas que ligam as tabelas indicam os
Modelo Taxon e suas relações. As linhas que ligam as tabelas indicam os métodos implementados nas classes mostradas, com cores indicando diferentes relacionamentos
Tabela Taxon
- Como, Localidades, o modelo Taxon tem um relacionamento pai-filho, implementado usando o modelo de conjunto aninhado para dados hierárquicos da biblioteca Laravel Baum que permite consultar ancestrais e descendentes. Consequentemente, as colunas
rgt,lftedephda tabela de taxons são preenchidas automaticamente por esta biblioteca na inserção ou atualização dos dados. - Para ambos, Taxon
authore Taxonbibreference, existem duas opções:- Para nomes publicados, a autoria da string recuperada pelas APIs será colocada na coluna
author = string. Para nomes não publicados, o autor é uma pessoa e será armazenado na colunaauthor_id. - Somente nomes publicados podem ter relação com BibReferences. O campo de string
bibreferenceda tabela Taxon armazena as strings recuperadas por meio de APIs externas, enquanto obibreference_idse vincula a um objeto Referência Bibliográfica. Eles são usados para armazenar a publicação onde o nome do táxon é descrito e pode ser inserido em ambos os formatos. - Além disso, um registro de táxon também pode ter muitas outras referências de Bib por meio de uma tabela dinâmica (
taxons_bibreference), permitindo vincular qualquer número de referências bibliográficas a um nome de táxon.
- Para nomes publicados, a autoria da string recuperada pelas APIs será colocada na coluna
- A coluna
levelrepresenta a classificação taxonômica (como ordem, gênero, etc.). Ele é numericamente codificado e padronizado de acordo com as regras gerais do IAPT, mas deve acomodar também categorias de nível de táxon relacionadas a animais. Consulte os códigos disponíveis na API Taxon para obter a lista de códigos. - A coluna
parent_idindica o pai do táxon, que pode estar vários níveis acima dele. O nível dos pais deve ser estritamente mais alto do que o nível do táxon, mas você não precisa seguir a hierarquia completa. É possível registrar um táxon sem os pais, por exemplo, um morfotipo não publicado para o qual o gênero e a família são desconhecidos pode ter umaordemcomo pai. - Os nomes das classificações taxonômicas são traduzidos de acordo com o
localedefinido pelo sistema, que também traduz a interface da web (atualmente implementados apenas em português e inglês). - O campo
nomeda tabela de táxons contém apenas a parte específica do nome (no caso de espécies, o epíteto específico), mas a inserção e exibição dos táxons por meio da API ou interface web deve ser feito com a combinação fullname. - É possível incluir sinônimos na tabela Taxon. Para isso, deve-se preencher o relacionamento
senior, que é o id do nome aceito (valid) para um táxoninvalid. Sesenior_idfor preenchido, o táxon é um sinônimojuniore deve ser marcado como invalid. - Ao inserir um novo táxon publicado, apenas o
nomeé necessário. O nome será validado e o autor, referência e sinônimos serão recuperados usando os seguintes serviços de API:- BackBone Taxonômico do GBIF - esta será a primeira verificação, da qual links para Tropicos e IPNI também podem ser recuperados se registrar um nome de planta.
- Tropicos - se não for encontrado no GBIF, o ODB pesquisará o nome no banco de dados de nomenclatura do Jardim Botânico do Missouri.
- IPNI - o Índice Internacional de Nomes Individuais é outro banco de dados usado para validar nomes individuais (temporariamente desativado)
- MycoBank - usado para validar um nome se não for encontrado pelos Tropicos nem IPNI apis, e usado para validar nomes para Fungi. Temporariamente desativado
- ZOOBANK - quando GBIF, Tropicos, IPNI e MycoBank não conseguem encontrar um nome, então o nome é testado contra a api ZOOBANK, que valida os nomes dos animais. Não fornece publicação de táxons, no entanto.
- Se um nome de táxon for encontrado nos bancos de dados Nomenclatural, o respectivo ID do repositório é armazenado nas tabelas
taxon_external, criando um link entre o registro do táxon OpenDataBio e o banco de dados nomenclatural externo. - Pessoas pode ser definidas como especialistas em táxons por meio de uma tabela dinâmica. Assim, um objeto Taxon pode ter vários especialistas taxonômicos registrados no OpenDataBio.
Acesso a dados: usuários plenos podem registrar um novo táxon e editar os registros existentes se eles não tiverem sido usados para identificação. Atualmente é impossível remover um táxon do banco de dados. A lista de táxons tem acesso público.
Vouchers
O modelo Voucher é usado para armazenar registros de espécimes ou amostras de indivíduos depositados em BioColeções. Portanto, as únicas informações obrigatórias exigidas para registrar um Voucher são individual, biocollection e se o espécime é um tipo de nomenclatural (o padrão é non-type se não informado).
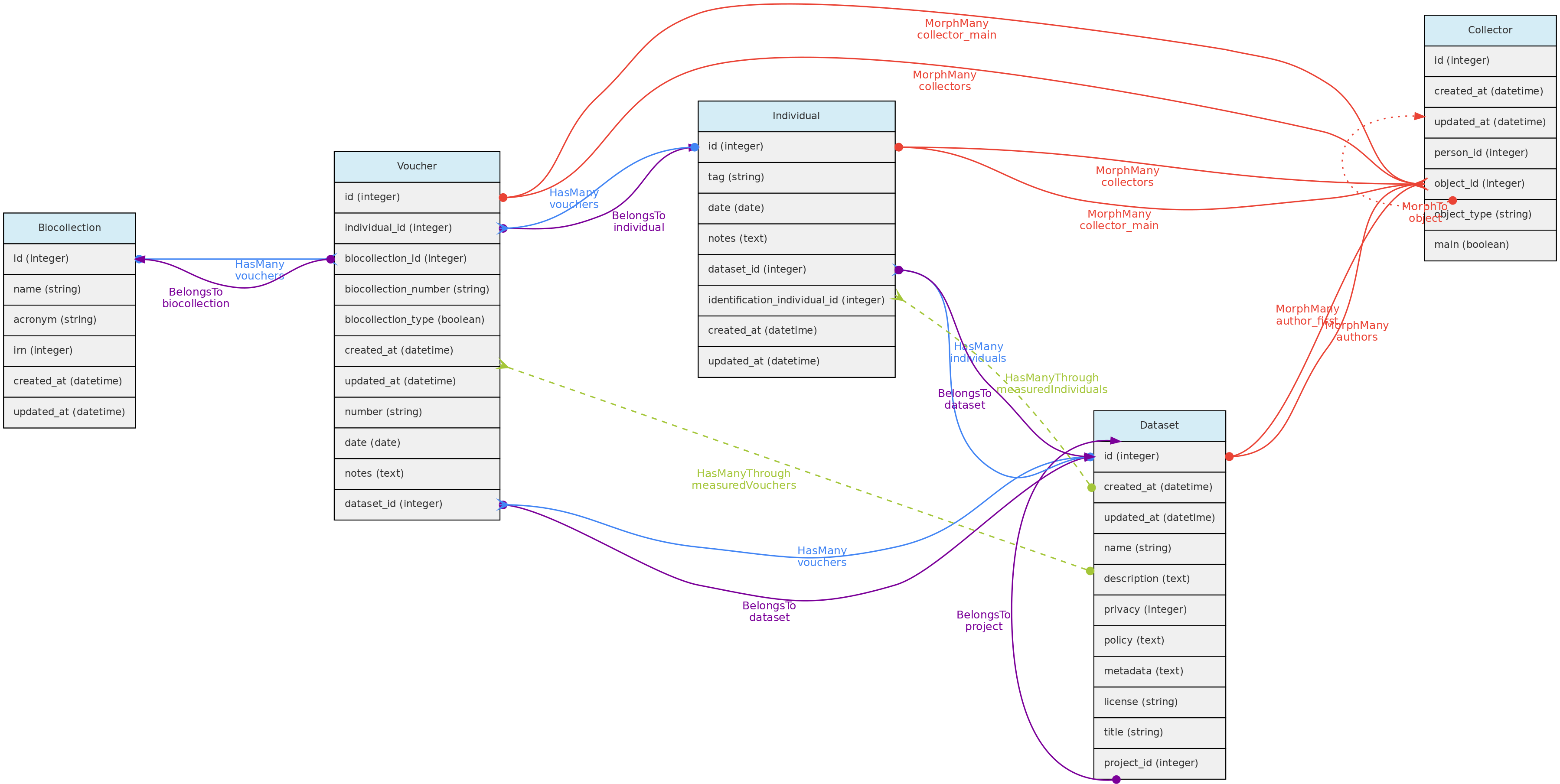 Modelo de voucher e suas relações. As linhas que ligam as tabelas indicam os
Modelo de voucher e suas relações. As linhas que ligam as tabelas indicam os métodos implementados nos modelos mostrados, com cores indicando diferentes relacionamentos. Note que a Identificação nem a Localidade são mostradas porque os Vouchers não têm seus próprios registros para esses dois modelos, eles são apenas herdados do Indivíduo ao qual o Voucher pertence
Vouchers table explained
- O Voucher pertence a um Indivíduo e a uma Biocoleção, portanto o
individual_ide obiocollection_idsão obrigatórios nesta tabela; biocollection_numberé o código alfanumérico do Voucher na Biocoleção, pode ser ’nulo’ para usuários que desejam apenas indicar que um Indivíduo registrado tem Vouchers em uma Bicoleção específica, ou para Vouchers registrados para biocoleções que não tem um código identificador;biocollection_type- é um código numérico que especifica se o Voucher na BioCollection é um tipo nomenclatural. O padrão é0(não é um tipo);1apenas para ‘Tipo’, uma forma genérica e outros números para outros tipos nomenclaturiscollectors, um ou múltiplos, são opcionais para Vouchers, exigidos apenas se forem diferentes dos Coletores do Indivíduo. Caso contrário, os coletores do Indivíduo são herdados pelo Voucher. Como para indivíduos, eles são implementados por meio de uma relações polimórfica com a tabela de collectors e o primeiro coletor é o coletor_principal para o voucher, ou seja, aquele que se relaciona comnumber.number, este é o número do coletor, mas como coletores, só deve ser preenchido se for diferente do valor datagdo indivíduo. Portanto,collectors,numberedatesão úteis para registrar Vouchers para Indivíduos que têm Vouchers coletados em momentos diferentes por pessoas diferentes.- O campo
datenos modelos indivíduos e Voucher pode ser uma data incompleta. Exigido apenas se for diferente do indivíduo a quem o voucher pertence. dataset_ido Voucher pertence a um Dataset, que controla a política de acesso;notesqualquer anotação de texto para o Voucher.- O modelo de Voucher interage com o modelo BibReference, permitindo vincular citações múltiplas a Vouchers. Isso é feito através da table
voucher_bibreference.
Acesso a dados Os vouchers pertencem a Conjuntos de Dados, portanto, a política de acesso a conjuntos de dados se aplica aos vouchers nele contidos. Os vouchers podem ter um conjunto de dados diferente de seus indivíduos. Se a política do conjunto de dados do Voucher for de acesso aberto e a Indivíduo não, o acesso aos dados do voucher será incompleto, portanto, o conjunto de dados do Voucher deve ter a mesma política de acesso ou uma política de acesso menos restrito do que o conjunto de dados do Indivíduo. Apenas os colaboradores e administradores do conjunto de dados podem inserir ou editar vouchers em um conjunto de dados, mesmo se o conjunto de dados for de acesso público.
4.2 - Objetos de Atributos
Medições
A tabela measurements armazena os valores para variáveis medidas para os objetos centrais. Seu relacionamento com os objetos centrais é definido por uma relações polimórficas usando as colunas measured_id e measured_type. Essas relações MorphTo são ilustradas e explicadas na página objetos centrais.
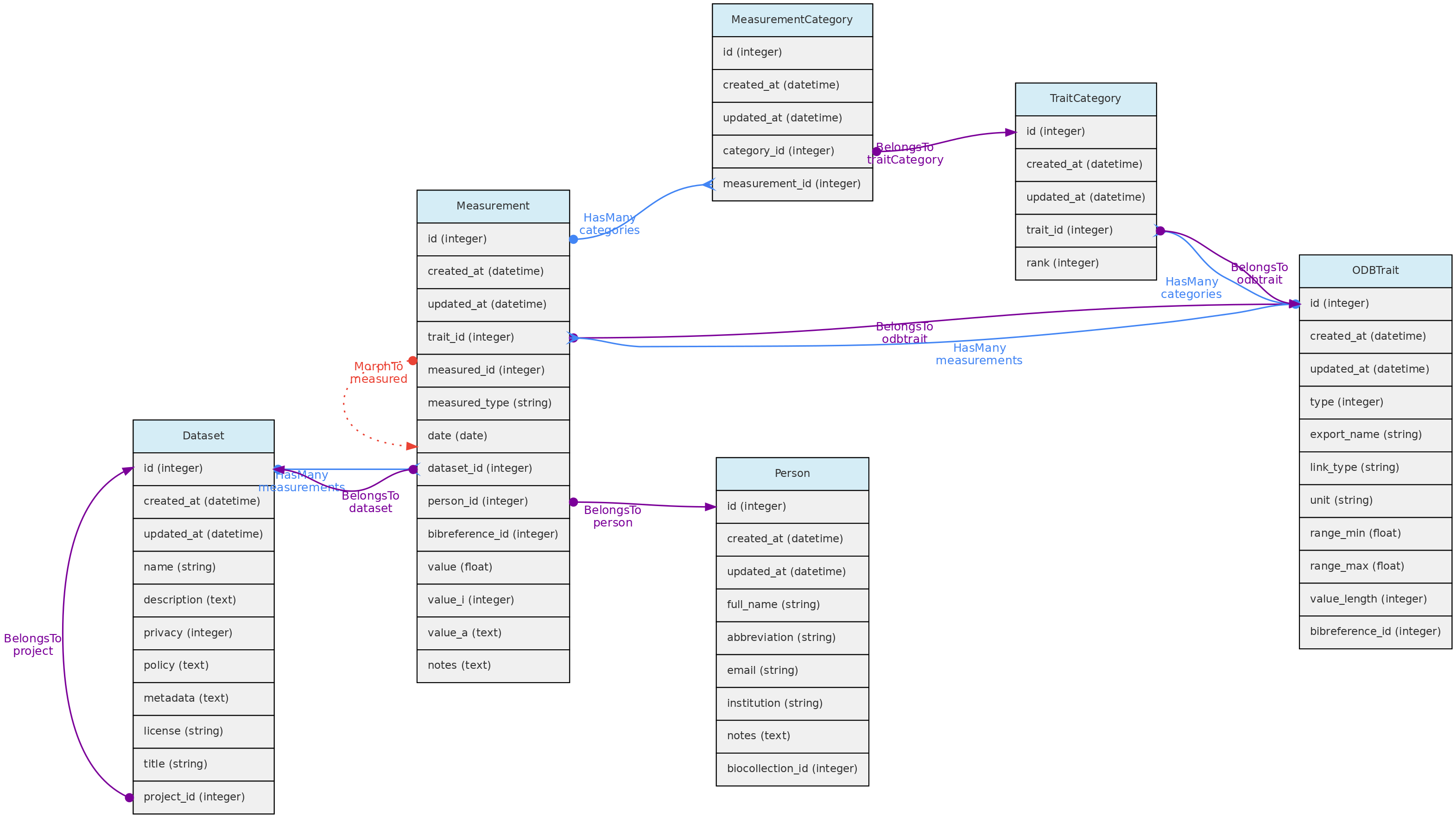
- medições devem pertencer a um Conjuntos de dados - coluna
dataset_id, que controla a política de acesso - Uma Pessoa deve ser indicada como o medidor (
person_id); - A coluna
bibreference_idpode ser usada para vincular medições extraídas de publicações à sua fonte fonte Bibliográfica - O valor para a variável medida (
trait_id) será armazenado em colunas diferentes, dependendo do tipo de variável:value- esta coluna flutuante armazenará valores para características reais quantitativas;value_i- esta coluna inteira armazenará valores para características Quantitative Integer; e é um campo opcional para características do tipo Link, permitindo, por exemplo, armazenar contagens para uma espécie (uma característica Link Taxon) em um local.value_a- esta coluna de texto armazenará valores para os tipos de traço Texto, Cor e Espectral.
- Valores para variáveis categóricas e ordinais são armazenados na tabela
measurement_category data- a data de medição é obrigatória em todos os casos
Acesso a dados As medições pertencem a Conjuntos de dados, portanto, a política de acesso a conjuntos de dados se aplica às medições nele. Apenas os colaboradores e administradores do conjunto de dados podem inserir ou editar medições em um conjunto de dados, mesmo se o conjunto de dados for de acesso público.
Variáveis
A tabela traits representa as variáveis definidas pelo usuário para coletar Mediçõespara um dos objetos centrais.
Essas características personalizadas fornecem enorme flexibilidade aos usuários para registrar suas variáveis de interesse. Obviamente, tal flexibilidade tem um custo na padronização dos dados, pois uma mesma variável pode ser registrada de forma diferentes em qualquer instalação do OpenDataBio. Para minimizar a redundância na ontologia de características, os usuários que criam características são avisados sobre esse problema e uma lista de características semelhantes é apresentada no caso de ser encontrada por comparação de nomes de características.
As variáveis têm restrições de edição para evitar perda de dados ou alteração não intencional do significado dos dados. Portanto, embora a lista de variáveis esteja disponível para todos os usuários, as definições de variáveis não podem ser alteradas se outra pessoa também usou a variável para armazenar medições.
Variáveis são entidades traduzíveis, então seus valores de name e description podem ser armazenados em vários idiomas (veja traduções de usuário. Isso é colocado na tabela user_translations através de uma relação polimórfica.
A definição da variável deve ser tão específica quanto necessário. A medição da altura das árvores usando medição direta ou um clinômetro, por exemplo, pode não ser facilmente convertida de uma para outra e deve ser armazenada em variáveis diferentes. Portanto, é altamente recomendável que o campo de definição de variável inclua informações como instrumento de medição e outros metadados que permitam que outros usuários entendam se podem usar sua variável ou criar uma nova.
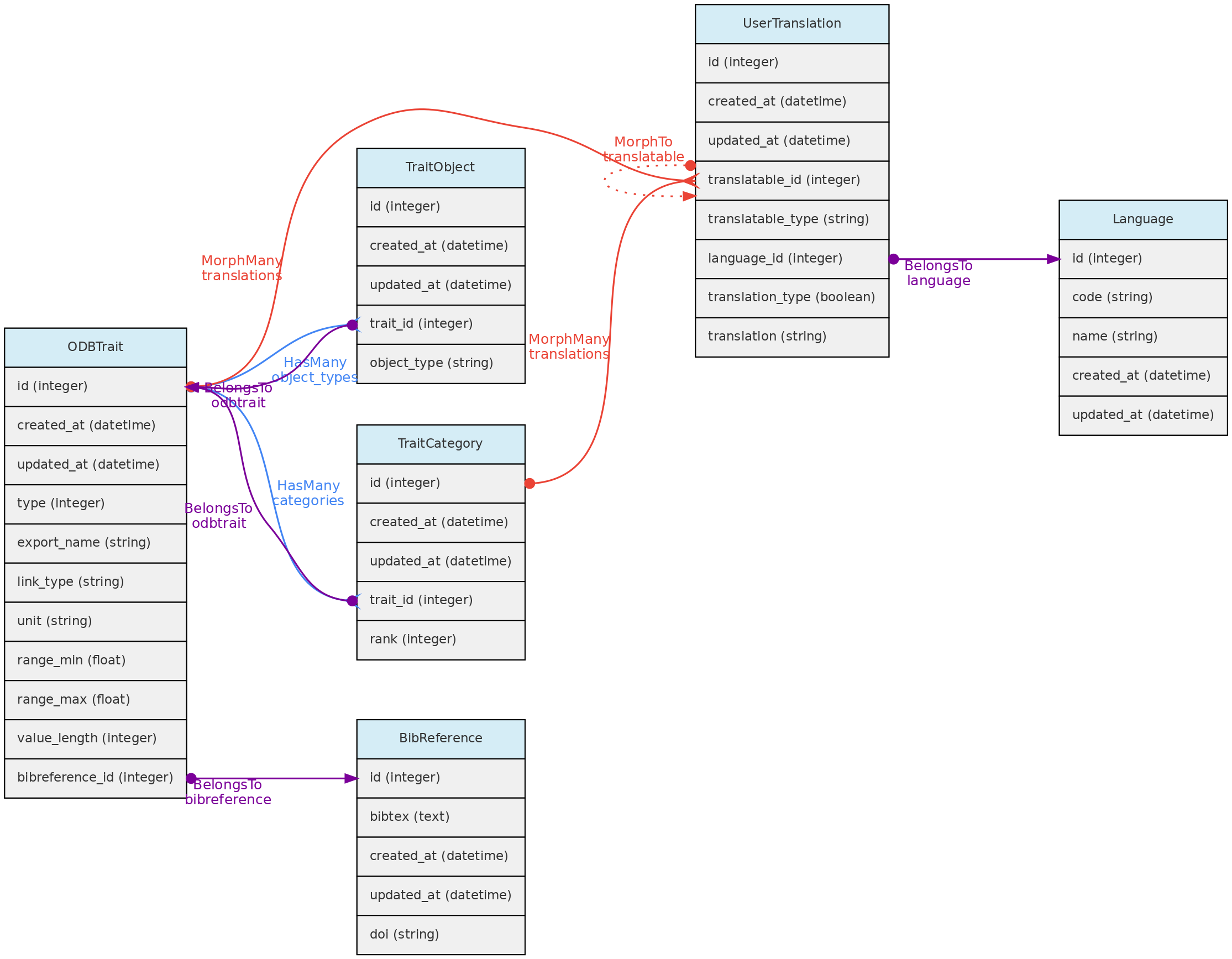
- A definição da variável deve incluir um
export_name, que será usado durante as exportações de dados nos formulários de entrada da interface. Os nomes de exportação devem ser únicos e não devem ter tradução. São recomendados nomes de exportação curtos e [camelCase](https://en.wikipedia.org/wiki/Camel_case) ou [PascalCase](https://en.wikipedia.org/wiki/pascal_case). - Os seguintes tipos de características estão disponíveis:
- Quantitativo real - para números reais;
- Número inteiro quantitativo - para contagens;
- Categórico - para varáveis categóricas de seleção única;
- Múltiplo categórico - para muitas categorias selecionáveis;
- Ordinal categórico - para uma categoria ordenada selecionável (dados semiquantitativos);
- Texto - para qualquer valor de texto;
- Cor - para qualquer valor de cor, especificado pelo código de cor hexadecimal (paleta de cores)
- Link - este é um tipo de variável especial no OpenDataBio. Apenas links para Taxons e Vouchers estão implementados. Exemplo de uso: se você deseja armazenar contagens de espécies conduzidas em uma Localidade, você pode criar uma variável do tipo de link_taxon ou link_voucher. A medição para tal variável terá um campo opcional
valuepara armazenar as contagens. Este tipo de característica também pode ser usado para especificar o hospedeiro de um parasita ou o número de insetos predadores. - Espectral - projetado para acomodar dados espectrais, compostos de vários valores de absorbância ou refletância para diferentes números de onda.
- GenBank - armazena os números de acesso do GenBank que permitem recuperar dados moleculares vinculados a indivíduos ou vouchers armazenados no banco de dados através do GenBank Serviço API.
- A tabela Traits contém campos que permitem a validação do valor da medição, dependendo do tipo de variável:
range_maxerange_min- se definido para variáveis quantitativas, as medições terão que se ajustar ao intervalo especificado;value_length- obrigatório apenas para variáveis espectrais, valida o comprimento (número de valores) de uma medição espectral;link_type- se a variável for do tipo Link, a medição emvalue_ideve ser um id do objeto do tipo de link;- Para variáveis do tipo cor, os valores são validados no processo de criação da medição e devem estar em conformidade com um código hexadecimal de cores. Um seletor de cores é apresentado na interface web para inserção e edição de medições de cores;
- Variáveis categóricas e ordinais serão validadas para as categorias registradas ao importar medições por meio da API;
- A coluna
unitdefine a unidade de medida para a variável. - A coluna
bibreference_idé a chave de uma única Referência Bibliográfica que pode ser vinculad à definição da variável. - A tabela
trait_objectsarmazena o tipo de objetos centrais para o qual a variável pode ter uma medição;
Acesso aos dados O nome, definição, unidade e categorias de uma variável não podem ser atualizados ou removidos se houver alguma medição registrada no banco de dados. As únicas exceções são: (a) é permitido adicionar novas categorias para variáveis categóricas (não ordinais); (b) o usuário atualizando a variavel é a única pessoa que possui medições para a característica; (c) o usuário que atualiza a variável é um administrador de todos os conjuntos de dados com medições usando a variável.
Formulários
Um Formulário é um grupo organizado de variáveis definido por um usuário para criar um formulário personalizado para inserir medições através da interface web. Um Formulário consiste em um grupo de variáveis ordenadas, que podem ser marcadas como “obrigatórias”. As entidades relacionadas são o Relatório e o Filtro.
Isso ainda é experimental e em desenvolvimento
4.3 - Objetos de Acesso
Os objetos centrais são: Localidades, Vouchers, Individual e Taxons. Essas entidades são consideradas “centrais” porque podem receber Medições, ou seja, você pode registrar valores para qualquer Variável.
Conjuntos de dados controlam acesso aos dados e representam um publicação de dados dinâmica, cuja versão é definida pela data da última edição do registros incluídos. Conjuntos de dados contém Medições, Indivíduos, Vouchers e Arquivos de Mídia.
Projetos são apenas grupos de Conjuntos de dados e de Usuários, representando grupos de usuários com acessibilidade comum a conjuntos de dados cuja privacidade é definida para ser controlada por um projeto.
BioColeções - este modelo serve para criar uma lista reutilizável de acrônimos de Coleções Biológicas no registro de Vouchers. Mas tem opcionalmente a possibilidade de gerir uma coleção de registros de Vouchers e seus Indivíduos, paralelamente ao controle provido por Conjuntos de dados. O controle é apenas na edição e na entrada de dados. Neste caso a BioColeção é administrada pelo sistema.
Projetos e BioColeções devem ter pelo menos um Usuário definido como administrador, que tem controle total sobre o conjunto de dados ou projeto, incluindo a atribuição das seguintes funções a outros usuários: administrador, colaborador ouvisualizador:
- Colaboradores podem inserir e editar objetos, mas não podem excluir registros, nem alterar o conjunto de dados ou a configuração do projeto.
- Visualizadores têm acesso somente de leitura aos dados que não são de acesso aberto.
- Apenas usuários plenos e super-admins podem ser designados como administradores ou colaboradores. Assim, se um usuário que era administrador ou colaborador de um conjunto de dados for rebaixado a “Usuário registrado”, ele se tornará um visualizador.
- Super-admins apenas podem habilitar uma BioColeção para ser administrada pelo sistema.
BioColeções
O modelo Biocollection tem duas funções: (1) prover uma lista de acrônimos para registrar Vouchers de qualquer Coleção Biológica ; (2) gerir os dados de uma Coleção Biológica, facilitando o registro de novos dados (qualquer usuário entra seus dados usando as validações realizadas pelo software e solicita pela interface aos curadores da Coleção o registro dos dados que é feito pelos usuários autorizados na BioColeção, que a partir da transferência passa a controlar a edição dos dados dos Indivíduos. A opção (2) precisa ser implementada por um usuário SuperAdministrador do sistema, que ao habilitar a BioColeção para ser administrada pelo sistema, implementa o modelo de solicitações ODBRequest para que usuários possam pedir dados, amostras, ou registros e mudanças nos dados.
O objeto Biocollection pode ser uma Coleção Biológica formal, como as registradas no Index Herbariorum, ou qualquer outra Coleção Biológica.
O objeto Biocollection também interage com o modelo Person. Quando uma Pessoa está vinculada a uma Biocoleção, ela será listada como especialista taxonômico e pode ter também um vínculo com Taxons.
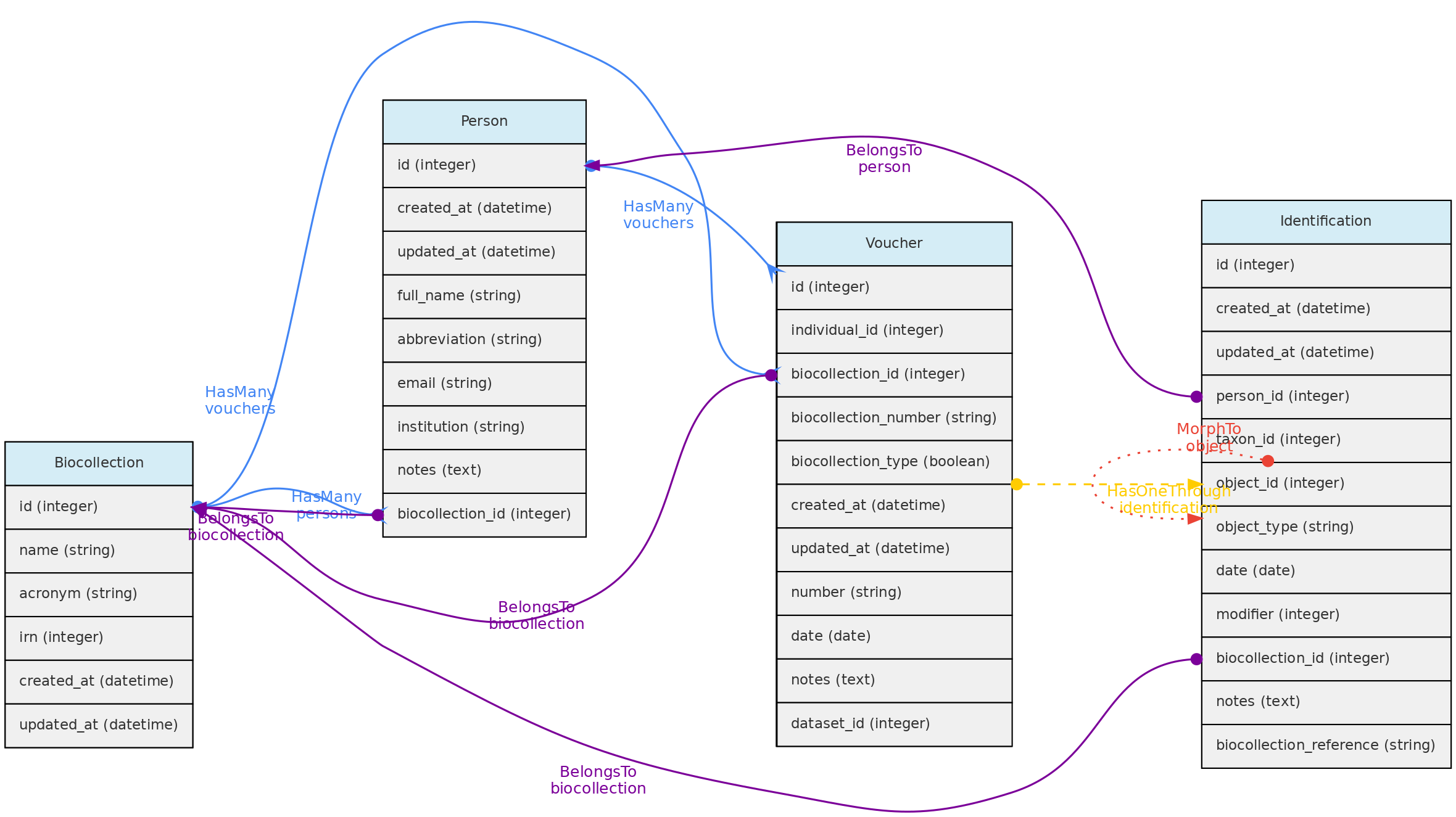
Acesso a dados - Usuários plenos podem registrar BioColeções, mas apenas usuários super-administradores podem tornar uma BioColeção adminstrável pelo sistema. Remover BioColeções pode ser feito se não há Vouchers ligados e se não é administrada pelo sistema. Se administrável, o modelo interage com Usuários, que podem ser administradores (curadores, pode tudo) ou colaboradores (podem entrar e editar dados, mas não podem apagar registros). Dados de outros Conjuntos de Dados podem fazer parte da BioColeção, permitindo que usuários tenham seus dados completo, mas o controle da edição dos registros é da BioColeção.
Conjuntos de Dados
Conjuntos de Dados são grupos de Medições, Indivíduos, Vouchers Arquivos de Mídia, ae podem ter um ou mais Usuários administrators, collaborators or viewers. Administradores podem definir o nivel de acesso para acesso público, restrito à usuários cadastrados or restrito à usuários autorizados or restrito à usuários do projeto. Este controle de acesso aos dados dentro de um conjunto de dados, conforme exemplificado no diagrama abaixo:
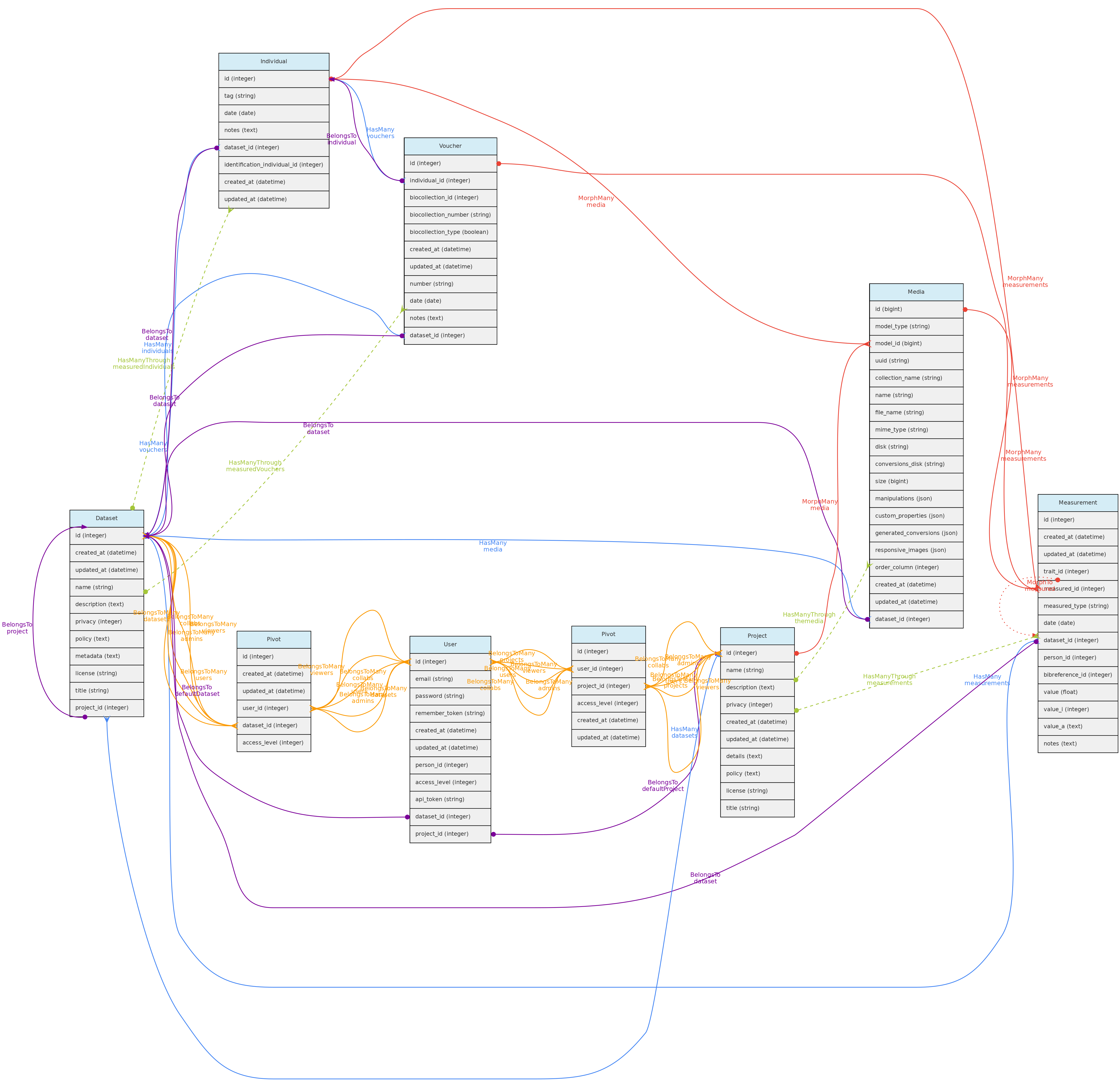
Conjuntos de dados podem ter muitas Referências bibliográficas, que junto com os campos policy, metadata permitem anotar o conjunto de dados com informações relevantes para o compartilhamento de dados:
* Vincule qualquer publicação que tenha usado o conjunto de dados e, opcionalmente, indique se são de citação obrigatória ao usar os dados;
* Defina uma política de dados específica ao usar os dados além de uma licença pública [CreativeCommons.org]((https://creativecommons.org/licenses/)
* Detalhe quaisquer metadados relevantes, além daqueles que são automaticamente recuperados do banco de dados, como as definições dos Variáveis medidas.
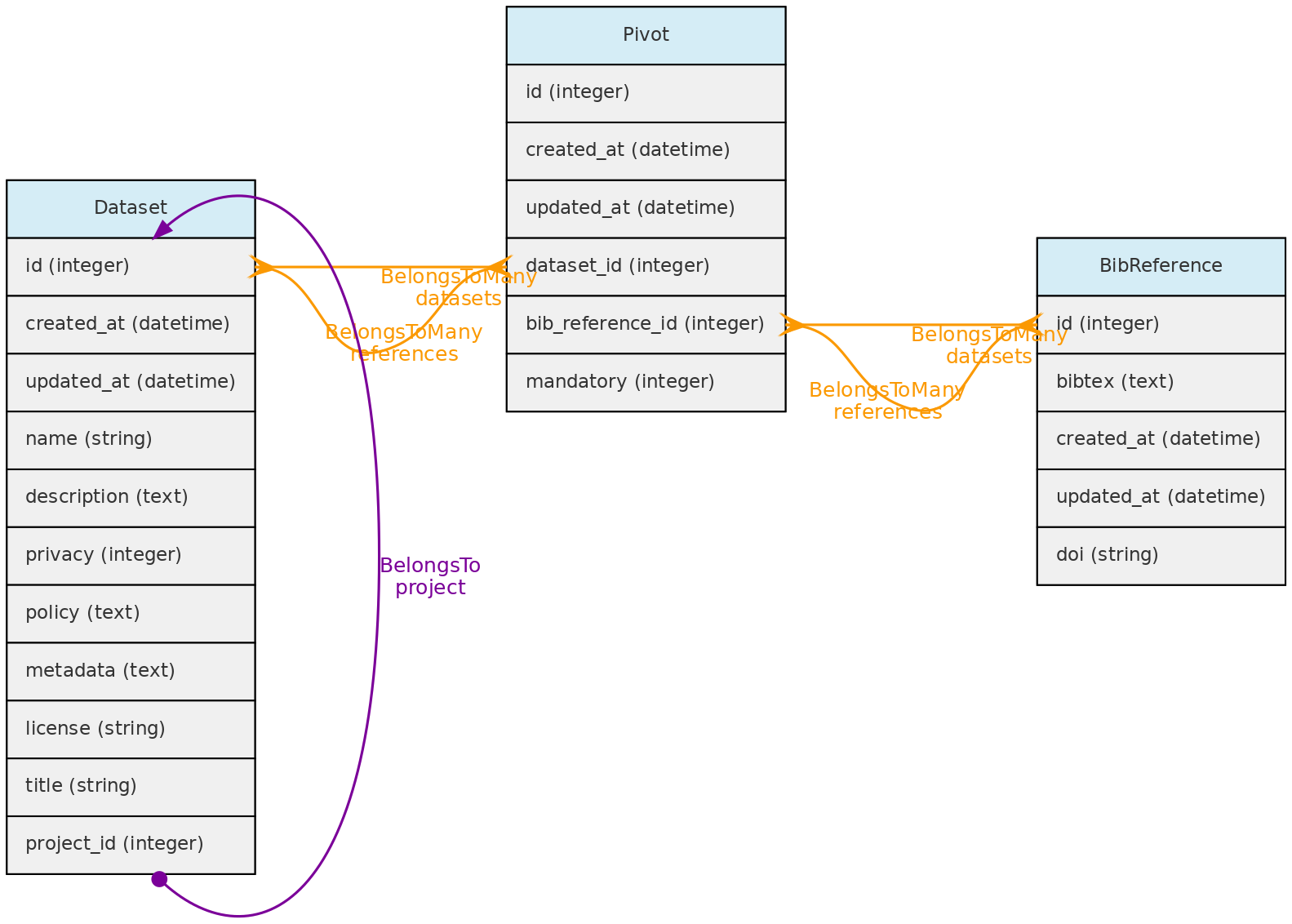
Projetos
Projetos são apenas grupos de Conjuntos de dados e interagem com Usuários, tendo administradores,colaboradores ou visualizadores. Esses usuários podem controlar todos os conjuntos de dados dentro do Projeto que tenham como política de acesso restrita aos usuários do projeto.
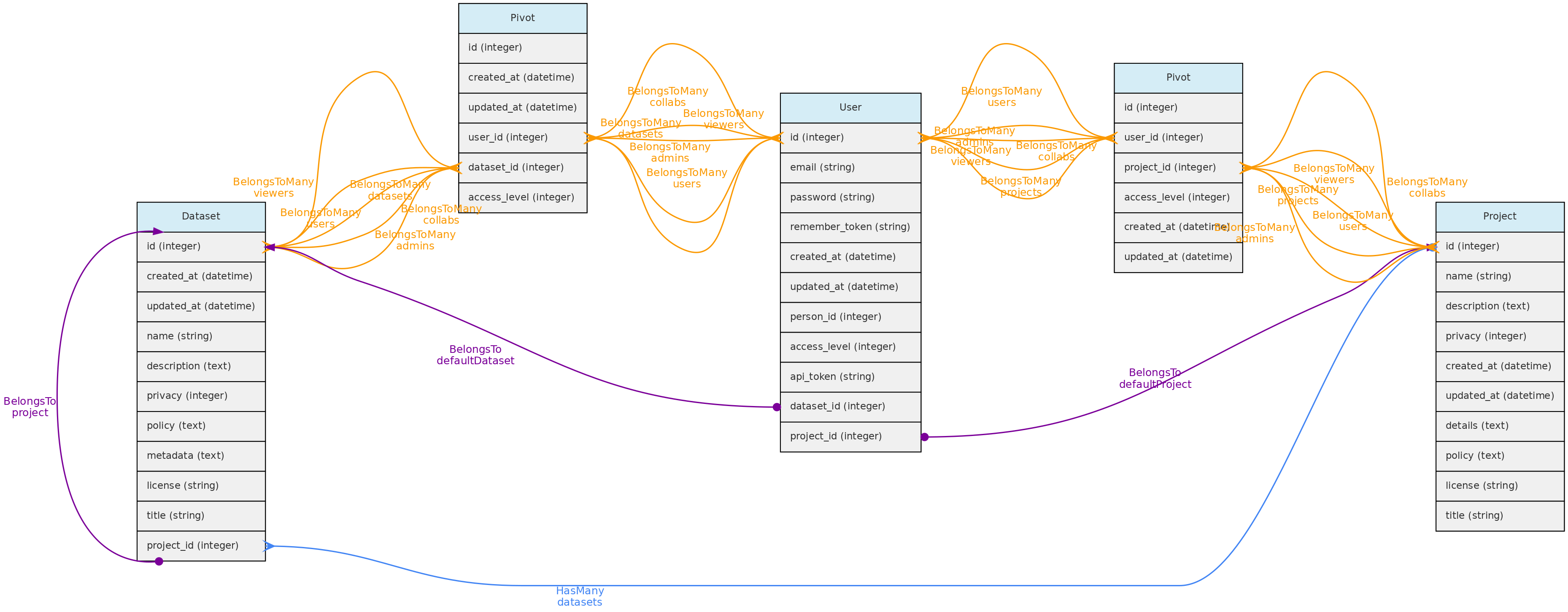
Usuários
O tabela users armazena informações sobre os usuários e administradores do banco de dados. Cada Usuário pode ser associado a uma Pessoa. Quando esse usuário insere novos dados, essa pessoa é usada como a pessoa padrão nos formulários. A pessoa só pode estar associada a um único usuário.
Existem três níveis de acesso possíveis para um usuário:
* Usuário registrado (o nível mais baixo) - tem muito poucas permissões
* Usuário pleno ou completo - podem ser atribuídos como administradores ou colaboradores de Projetos e Conjuntos de Dados;
* SuperAdmin (o nível mais alto). - os superadministradores têm acesso a todos os objetos, independentemente da configuração do conjunto de dados. Categoria para os administradores da instalação.
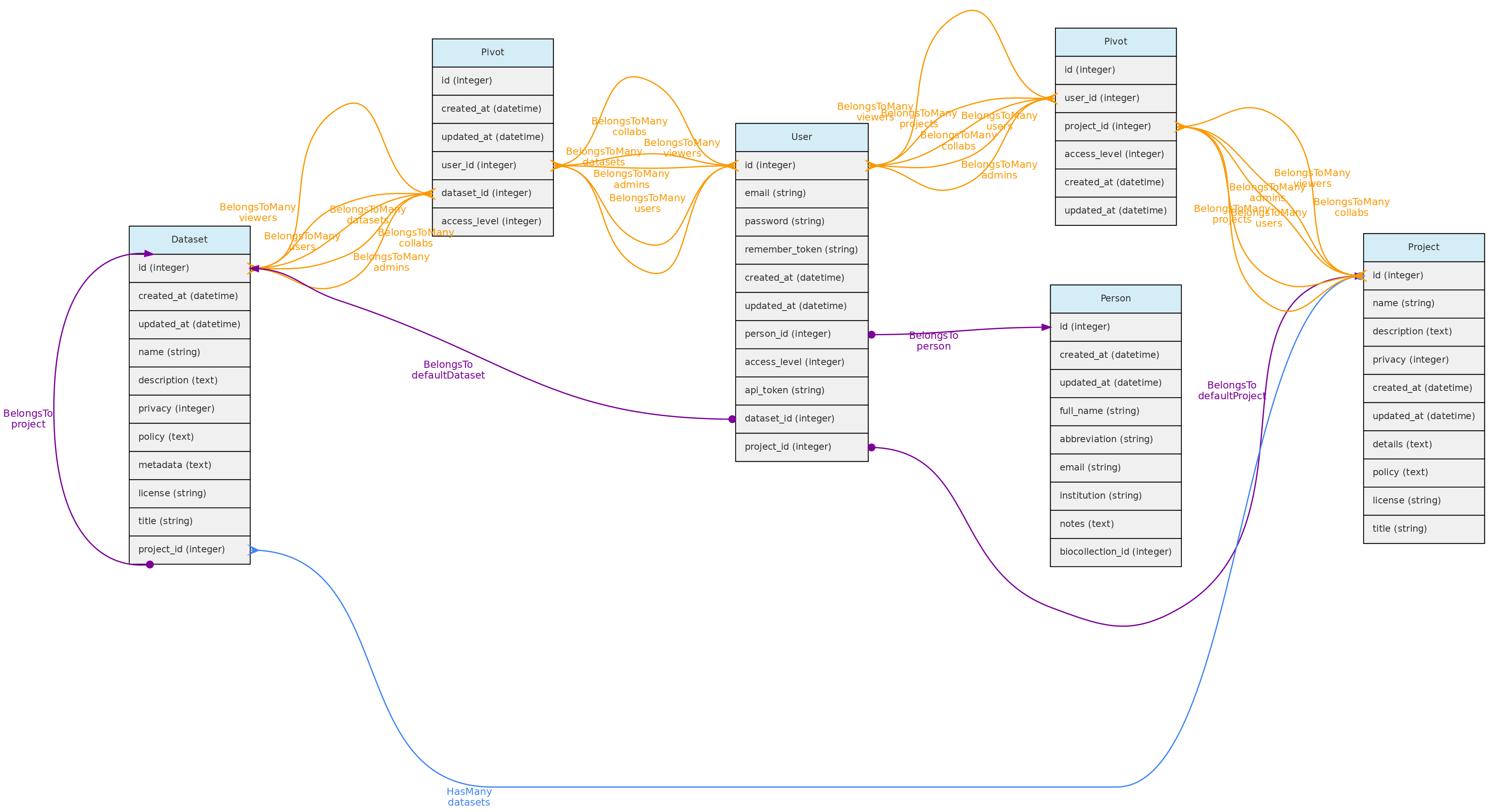
Cada usuário é atribuído ao nível usuário registrado quando ele ou ela se registra em um sistema OpenDataBio. Depois disso, alguém que seja SuperAdmin pode promovê-lo a Usuário Pleno ou SuperAdmin. Os SuperAdmins também podem editar outros usuários e removê-los do banco de dados.
Para cada usuário pleno é criado um projeto e um conjunto de dados de uso pessoal. Esses workspaces permitem que os usuários importem dados antes de incorporá-los a um projeto maior ou mais definitivo.
Acesso a dados: os usuários são criados no momento do registro. Apenas os administradores podem atualizar e excluir registros do usuário.
Jobs do usuário
A tabela user_jobs é usada para armazenar temporariamente tarefas que são executadas em segundo plano, como a importação e exportação de dados. Qualquer usuário tem permissão para criar um Job; cancelar seus próprios Jobs; listar os Jobs que não foram excluídos.
A tabela jobs contém os dados usados pelo framework Laravel para interagir com a Queue. Os dados desta tabela são excluídos quando o trabalho é executado com êxito. A tabela user_jobs é usada para manter essas informações, além de permitir logs do processo, repetir tarefas com erros e cancelar tarefas que ainda não foram concluídas.
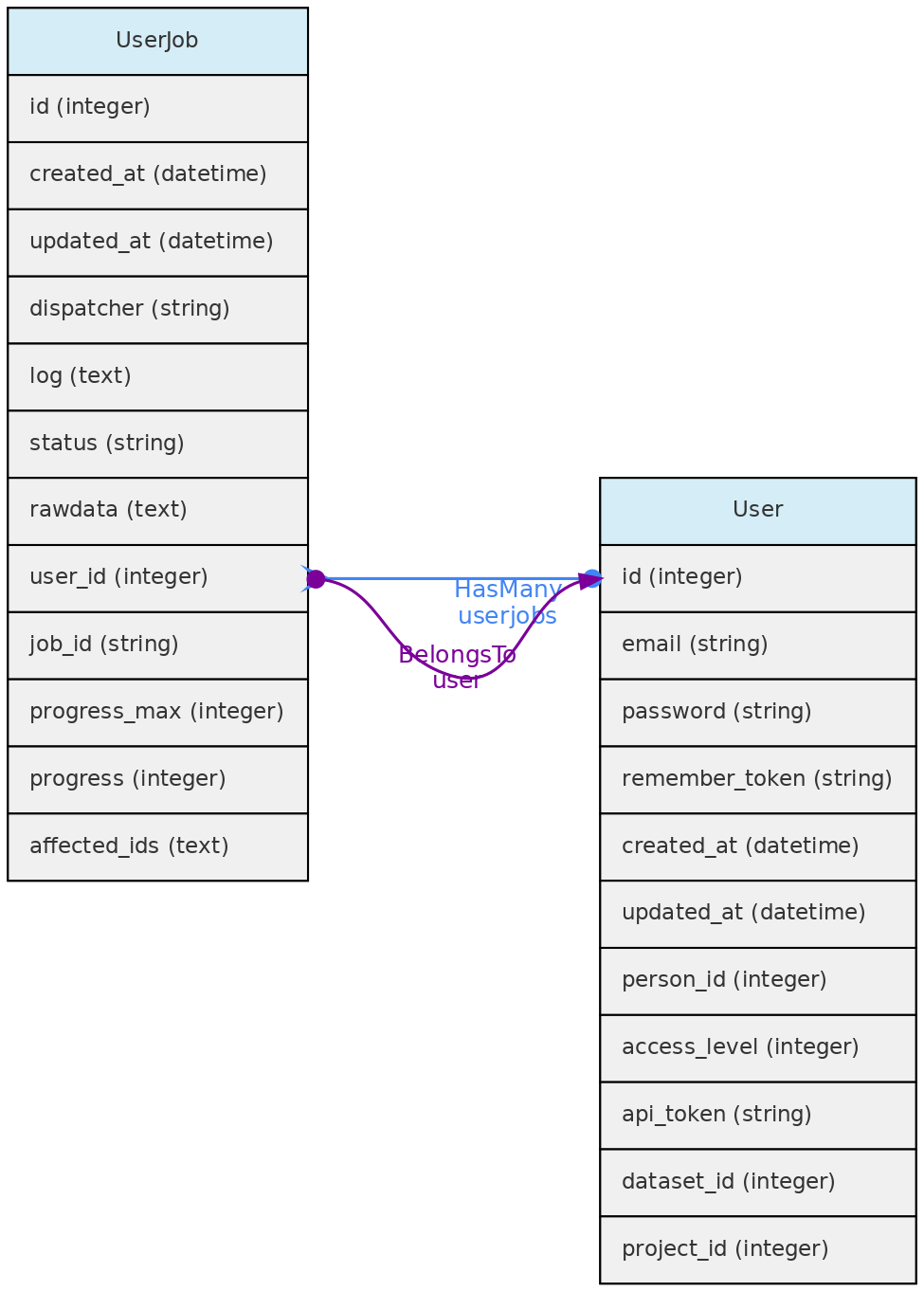
Acesso a dados: Cada usuário registrado pode ver, editar e remover seus próprios Jobs.
4.4 - Objetos Auxiliares
Referências Bibliográficas
A tabela bib_references contém basicamente referências formatadas em BibTex armazenadas na coluna bibtex. Você pode facilmente importar referências para o OpenDataBio através da interface, apenas especificando o doi ou simplesmente enviando um registro bibtex ou um arquivo .bib. Essas referências bibliográficas podem ser usadas para:
- Conjuntos de dados - com a opção de definir referências para as quais a citação é obrigatória quando o Conjunto de Dados for usado em publicações; mas todas as referências que usaram o conjunto de dados podem ser vinculadas ao conjunto de dados; os links são feitos com uma tabela dinâmica chamada
dataset_bibreference; - Taxons:
- para especificar a referência na qual o nome do táxon foi descrito, atualmente obrigatório em algumas revistas taxonômicas como PhytoTaxa. Esta referência de descrição é armazenada no
bibreference_idda tabela Taxons. - para registrar qualquer referência a um nome de táxon, que são então vinculados por meio de uma tabela dinâmica chamada
taxons_bibreference.
- para especificar a referência na qual o nome do táxon foi descrito, atualmente obrigatório em algumas revistas taxonômicas como PhytoTaxa. Esta referência de descrição é armazenada no
- Vincule uma Medição a uma fonte publicada;
- Indique a origem de uma definição de Variável.
- Indique citações obrigatórias para um conjunto de dados ou vincule referências usando os dados para um conjunto de dados
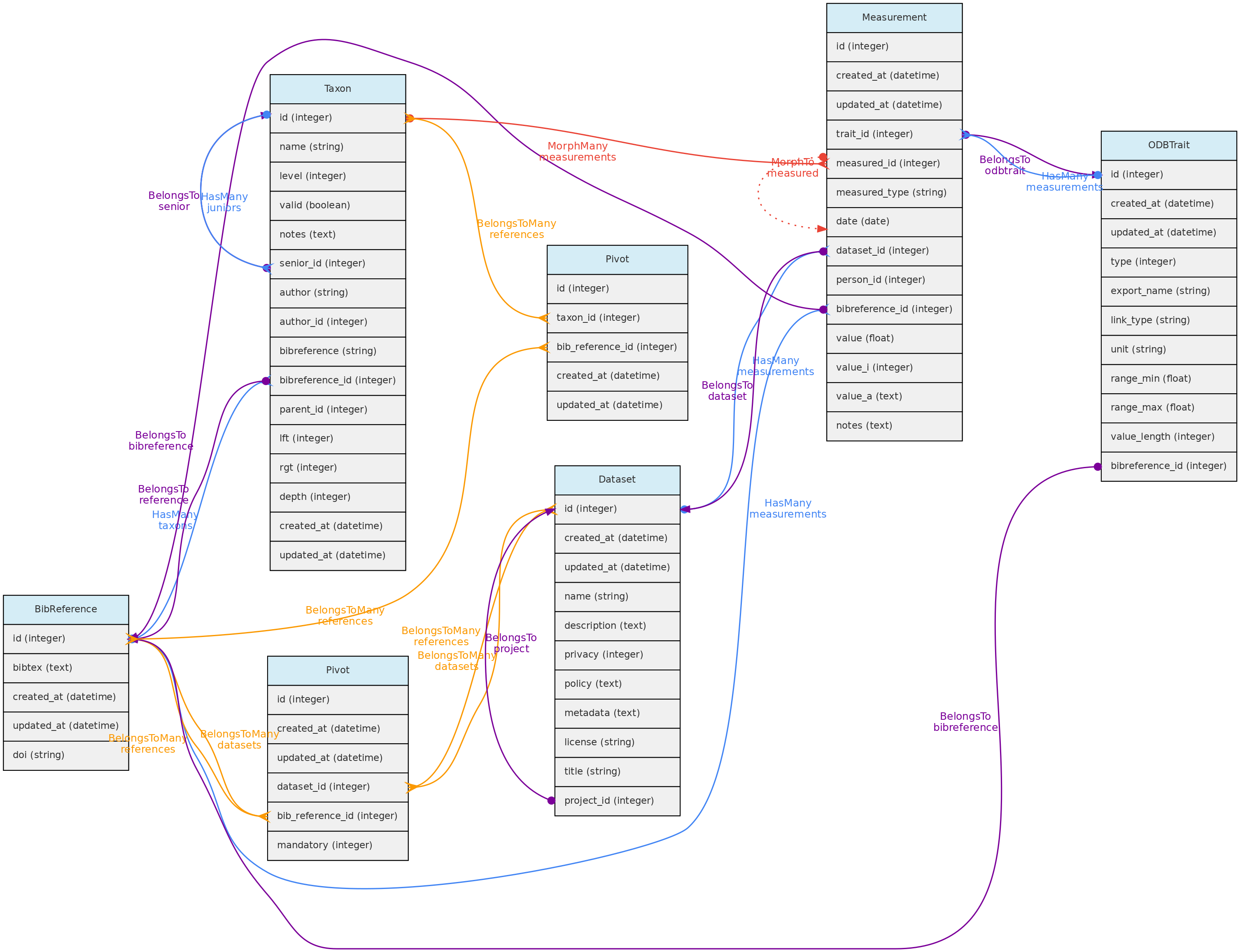 O modelo BibReference e seus relacionamentos. As linhas que ligam as tabelas indicam os
O modelo BibReference e seus relacionamentos. As linhas que ligam as tabelas indicam os métodos implementados, com cores indicando diferentes relacionamentos
Tabela Bibreferences
- A
bibtexKey, autores e outros campos relevantes são extraídos do registro em formato bibtex armazenado na colunabibtex. - A ** bibtexkey deve ser única** no banco de dados e uma função auxiliar é fornecida para padronizá-la com o formato
<von? sobrenome> <ano> <primeira palavra do título>. A “parte von” do nome é “von”, “di”, “de la”, que fazem parte do sobrenome de alguns autores. A primeira palavra do título ignora palavras de interrupção comuns, como “a”, “o” ou “em”. - DOI para uma referencia pode ser especificado no campo BibTex relevante ou em uma entrada de texto separada, e são armazenados no campo
doiquando presente. Uma API externa encontra o registro bibliográfico quando um usuário informa odoina interface.
**Acesso a dados** [usuários completos](/docs/concepts/data-access/#user) podem registrar novas referências, editar detalhes de referências e remover registros de referência que não têm dados associados. BibReferences tem acesso público!
Identificação Taxonômica
O modelo Identificação representa a identificação taxonômica de Indivíduos.
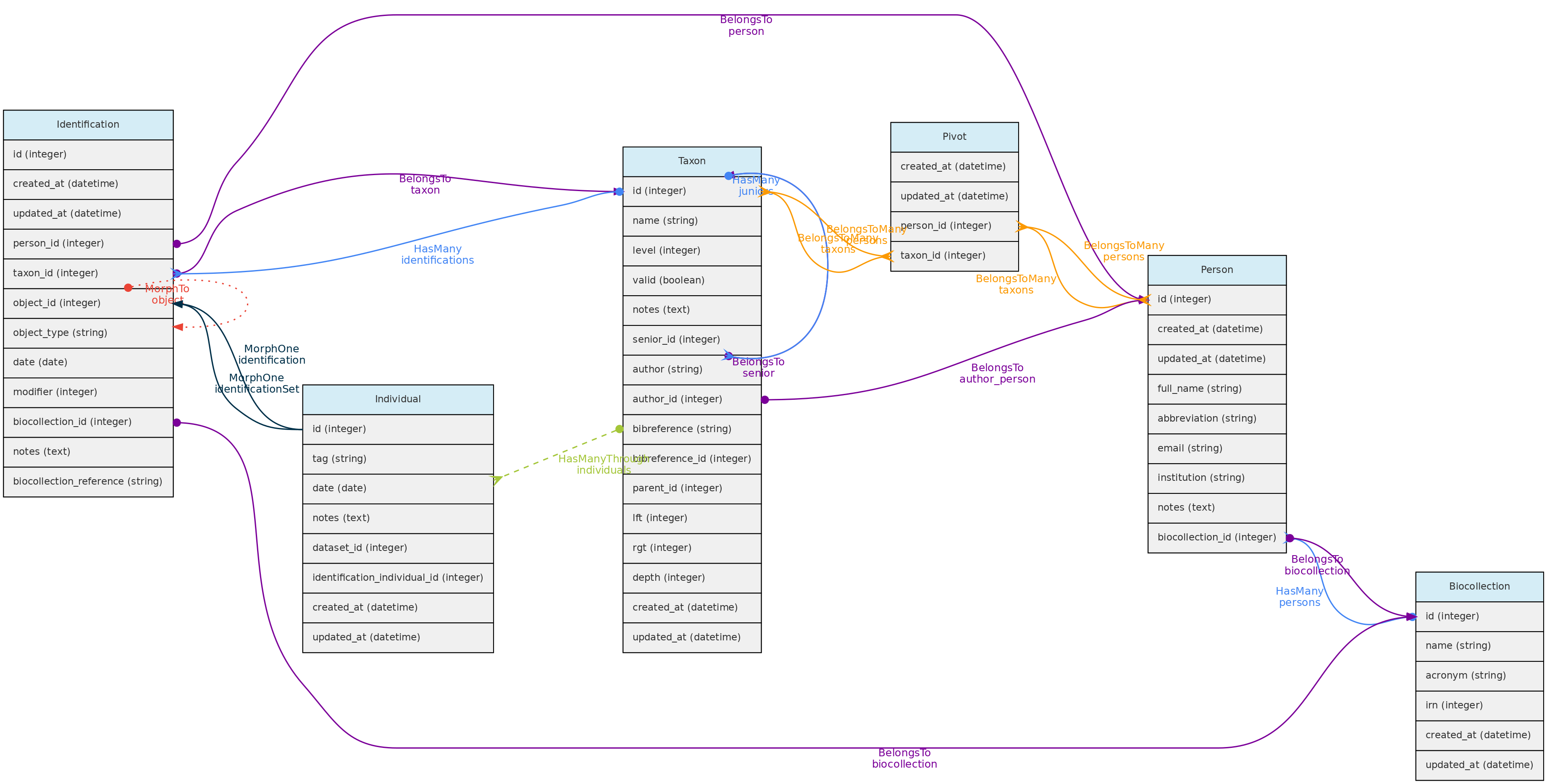 Modelo de identificação e suas relações. Linhas ligando tabelas indicam os
Modelo de identificação e suas relações. Linhas ligando tabelas indicam os métodos implementados, com cores indicando diferentes relacionamentos do Laravel Eloquent
Tabela identifications
- O modelo de Identificação inclui vários campos opcionais, mas além de
taxon_id,person_id, a Pessoaresponsável pela identificação e adatede identificação são obrigatórios.
- O valor de
datepode ser uma Data Incompleta, por exemplo apenas o ano ou ano + mês podem ser registrados.
- Os seguintes campos são opcionais:
modifier- é um código numérico que anexa um modificador taxonômico ao nome. Valores possíveis ’s.s.’ = 1, ’s.l.’ = 2, ‘cf.’ = 3, ‘aff.’ = 4, ‘vel aff.’ = 5, o padrão é 0 (nenhum).notes- um texto de escolha, útil para adicionar comentários à identificação.biocollection_idebiocollection_reference- esses campos devem ser usados para indicar que a identificação é baseada na comparação com um voucher depositado em uma coleção biológica e cria um link entre o indivíduo identificado e o espécime da BioColeção no qual a identificação foi baseada.biocollection_idarmazena o id de BioColeção ebiocollection_referenceo identificador único do espécime comparado, ou seja, seria o equivalente aobiocollection_numberdo modelo Voucher, mas esta referência não precisa ser de um voucher registrado no banco de dados.
- O relacionamento com o modelo individual é definido por relações polimórficas usando os campos
object_typeeobject_id. [Na estrutura atual, isso pode ser substituído por um ‘individual_id’ na tabela de identificação. A relação polimórfica foi herdada de uma versão anterior de desenvolvimento e foi mantida porque o modelo de Identificação poderá ser necessário para vincular outros objetos]. - Mudanças na Identificação de Indivíduos são auditadas para rastrear o histórico de mudanças
Acesso aos dados: as identificações são atributos dos Indivíduos e não possuem acesso independente!
Pessoas
O modelo Persons armazena nomes de pessoas que podem ou não ser um Usuário diretamente envolvido com a base de dados. Pessoas podem ser:
* coletores de Vouchers, Indivíduos e Arquivos de Mídia
* identificadores taxonômicos de Indivíduos;
* medidores de Medições;
* autores para nomes não publicados de Taxons;
* especialistas taxonômicos - ligados ao modelo Taxon pela tabela person_taxon;
* autores de Conjuntos de Dados
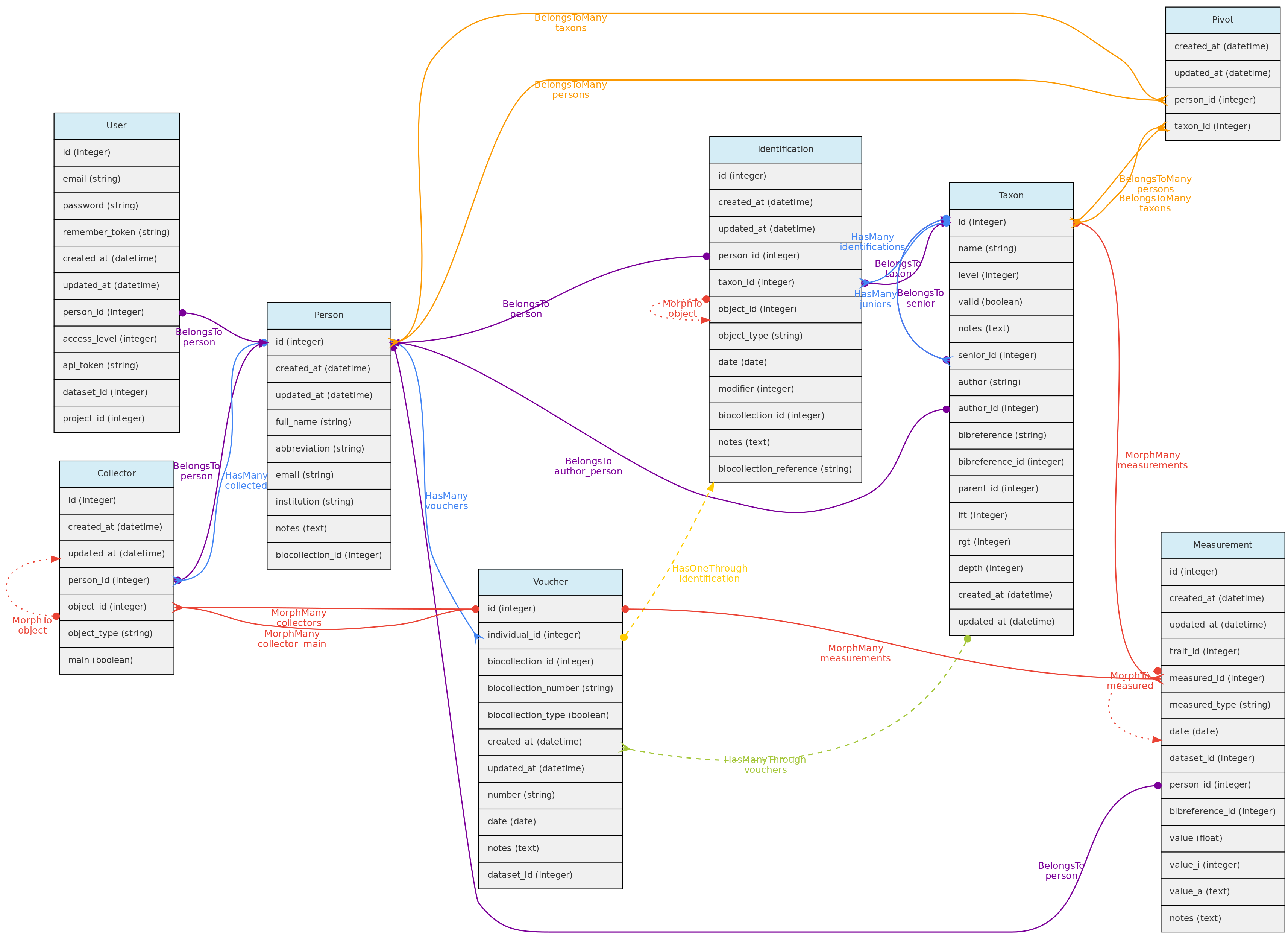 Modelo Persons e seus relacionamentos. As linhas que ligam as tabelas indicam os
Modelo Persons e seus relacionamentos. As linhas que ligam as tabelas indicam os métodos implementados, com cores indicando diferentes tipos de métodos do Laravel Eloquent, linhas sólidas as relações diretas e tracejadas as indiretas
Tabela persons
- as colunas obrigatórias são a pessoa
full_nameeabbreviation; - ao cadastrar uma nova pessoa, o sistema sugere o nome
abbreviation, mas o usuário é livre para alterá-lo para melhor adaptá-lo à abreviatura usual de cada pessoa. A ** abreviatura deve ser única ** no banco de dados, duplicatas não são permitidas na tabela Pessoas. Portanto, duas pessoas com exatamente o mesmo nome devem ser diferenciadas de alguma forma na colunaabbreviation. - A coluna
biocollection_idda tabela Pessoas é usada para listar a qual BioColeção uma pessoa está associada, que pode ser usada quando a Pessoa também é um especialista taxonômico. - Adicionalmente, também podem ser informados o
e-maile ainstitutiona que pertence a pessoa. - Cada usuário pode ser vinculado a uma pessoa pelo
person_idna tabela Usuário. Essa pessoa é então usada como a pessoa ‘padrão’ quando o usuário está logado no sistema.
**Acesso a dados** [usuários completos](/docs/concepts/data-access/#user) podem registrar novas pessoas e editar as pessoas inseridas e remover pessoas que não possuem dados associados. Os administradores podem editar qualquer pessoa. A lista de pessoas tem acesso público.
Arquivos de Mídia
Arquivos de mídia são semelhantes a Medições no sentido de que podem estar associados a qualquer objeto central. Os arquivos de mídia podem ser imagens (jpeg, png, gif, tif), arquivos de vídeo ou áudio e podem ser disponibilizados gratuitamente ou colocados em um Conjuntos de dados com uma política de acesso definida. Uma licença CreativeCommons.org pode ser atribuída a eles. Os arquivos de mídia podem ser anotados, ou seja, você pode definir palavras-chave para eles, permitindo consultá-los por Tags. Por exemplo, uma imagem individual pode ser marcada com ‘flores’ ou ‘frutos’ para indicar o que está na imagem, ou uma tag que informa sobre a qualidade da imagem.
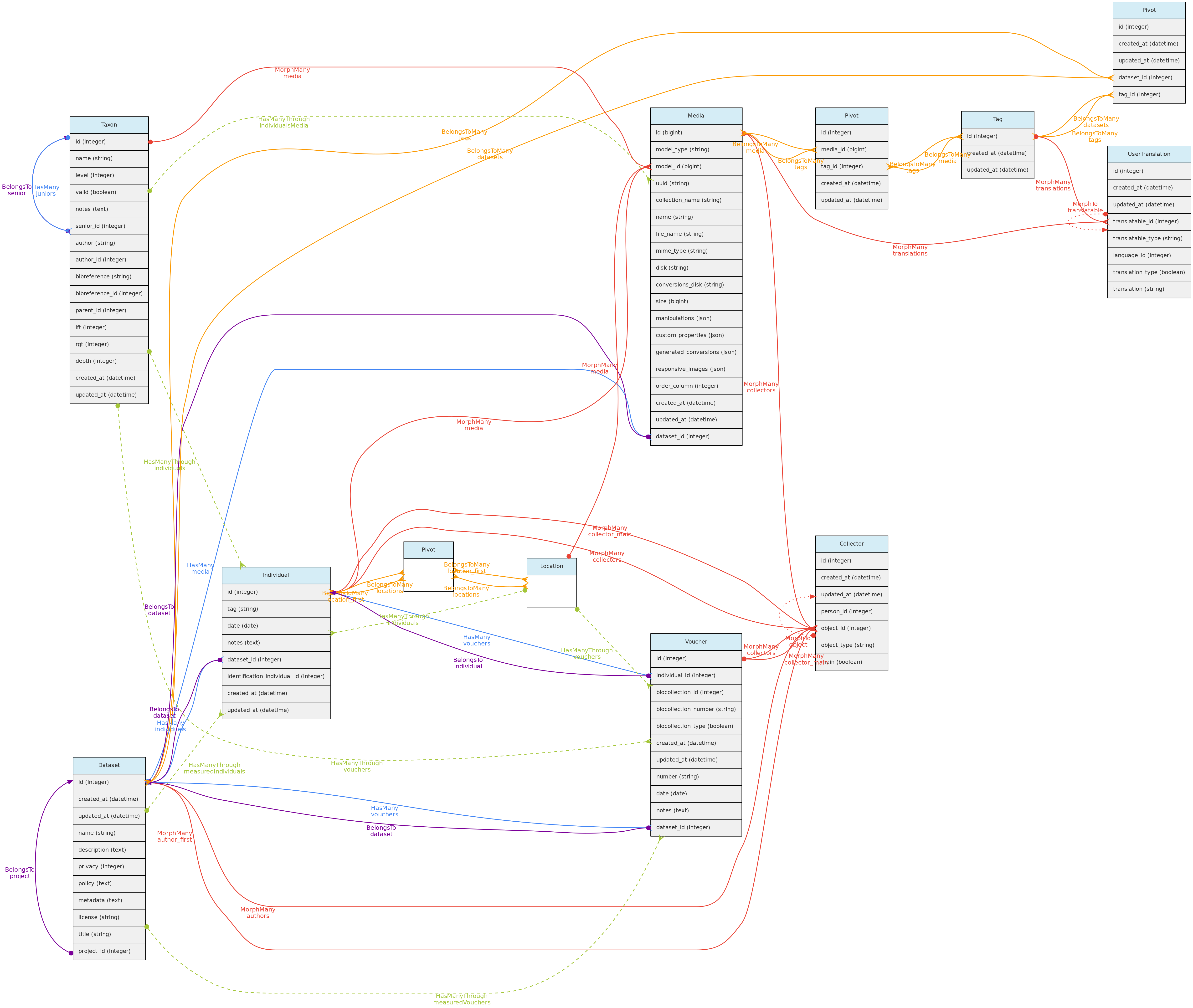
- Os arquivos de mídia (imagem, vídeo, áudio) são vinculados aos objetos centrais por meio de uma relações polimórfica definida pelas colunas
model_idemodel_type. - Múltiplas Pessoas podem ser associadas à Mídia para créditos, eles estão vinculados à tabela collectors, também através de relações polimóficas.
- Uma Mídia pode ter uma
descriptionem cada idioma configurado na tabela languages, que será armazenada na tabelauser_translations. As entradas para cada idioma são mostradas nos formulários da interface. - Os arquivos de mídia não são armazenados no banco de dados, mas na pasta de armazenamento do servidor.
- É possível fazer upload em lote de arquivos de mídia por meio da interface web, exigindo também um arquivo informando os objetos aos quais vincular a mídia e os metadados.
Acesso a dados usuários completos podem registrar arquivos de mídia e excluir aqueles que eles inseriram. Se a mídia estiver em um conjunto de dados, os administradores do conjunto de dados podem excluir a mídia além do usuário. Os arquivos de mídia têm acesso público, exceto quando vinculados a um conjunto de dados com restrições de acesso.
Tag Model
O modelo Tag permite que os usuários definam palavras-chave traduzíveis que podem ser usadas para sinalizar Conjuntos de dados, Projetos ou Arquivos de Mídia. O modelo Tag está vinculado a esses objetos por meio de uma tabela para cada um, denominada dataset_tag, project_tag e media_tag, respectivamente.
Um Tag pode ter name e description em cada idioma configurado na tabela de Idiomas, que serão armazenados na tabela user_translations. As entradas para cada idioma são mostradas nos formulários da interface.
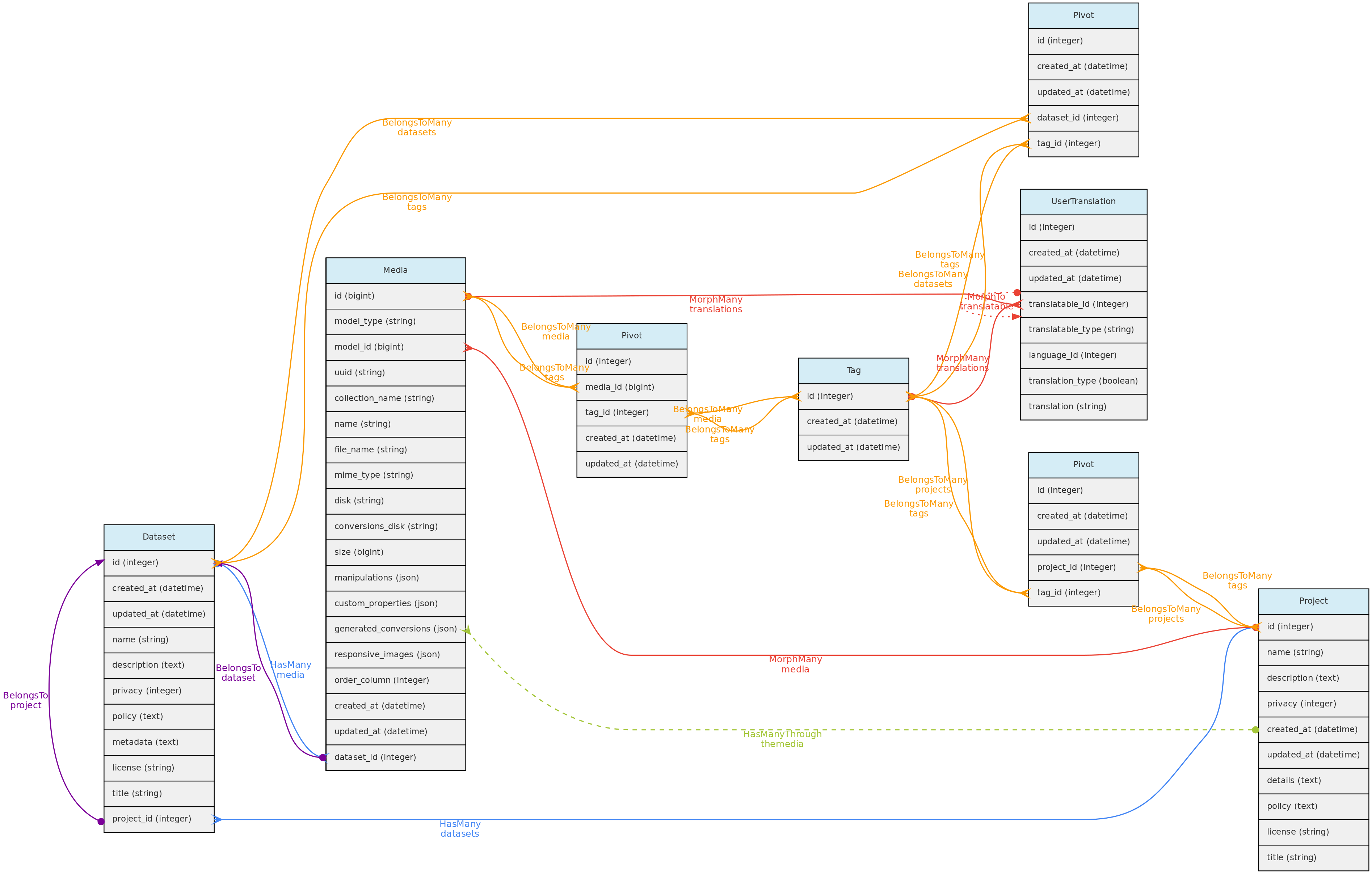
Acesso a dados usuários completos podem registrar tags, editar as inseridas e excluir as que não foram usadas. As tags têm acesso público, pois são apenas palavras-chave para facilitar a navegação.
Traduções do usuário
O modelo UserTranslation armazena as traduções de dados do usuário para: descrições e nomes de Variáveis e de categorias para variáveis categóricas; descrições de Arquivos de Mídia e para Tags. As relações com esses modelos são estabelecidas por relações polimórfica usando os campos translatable_type e translatable_id. Este modelo permite traduções para qualquer idioma listado na tabela languages, atualmente acessível apenas para inserção e edição diretamente no banco de dados SQL. Os formulários de entrada na interface web serão listados para os idiomas registrados.
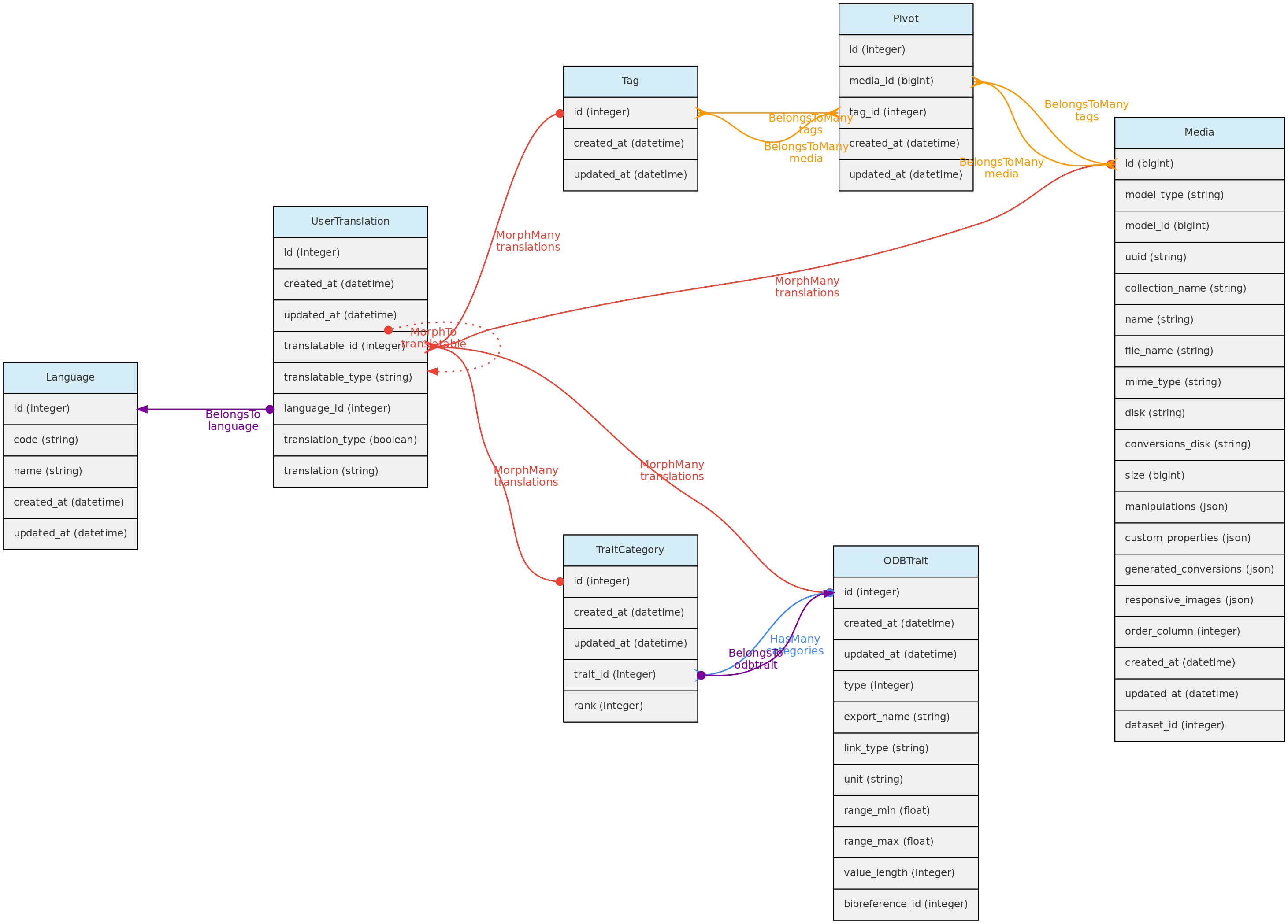
Datas incompletas
Datas para Vouchers, Indivíduos, Medições e Identificações podem ser incompletas, mas pelo menos ano é obrigatório em todos os casos. As colunas date nas tabelas são do tipo ‘date’ e as datas incompletas são armazenadas com 00 na parte ausente: ‘2005-00-00’ quando apenas o ano é conhecido; ‘1988-08-00’ quando apenas o mês é conhecido.
Auditando mudanças
As modificações nos registros do banco de dados são registradas na tabela activity_log. Esta tabela é gerada pelo pacote ActivityLog. As atividades são mostradas em um link ‘Histórico’ fornecido no show.view dos modelos.
- O pacote armazena as alterações como json no campo
properties, que contém dois elementos:attributeeold, que são basicamente os valores novos vs antigos que foram alterados. Essa estrutura deve ser respeitada. - A classe ActivityFunctions contém funções personalizadas para ler as propriedades do registro Json armazenado na tabela
activity_loge encontra os valores para mostrar na tabela de dados History; - A maioria das mudanças são registradas pelo pacote como um ’trait’ chamada dentro da classe. Estes permitem registrar automaticamente a maioria das atualizações e são configurados para registrar apenas os campos que foram alterados, não registros inteiros (opção
dirty). Além disso, a criação de registros não é anotada como atividade, apenas as alterações. - Algumas alterações, como de coletores e de identificações indivíduos são registradas separadamente, pois envolvem tabelas relacionadas e o registro é especificado nos arquivos do Controlador;
- O registro contém um campo
log_nameque agrupa os tipos de registro e é usado para distinguir os tipos de atividade e é útil para pesquisar a tabela de dados do histórico; - Dois registros especiais também são feitos para Conjuntos de Dados:
- Qualquer download de um Conjunto de Dados pela interface é registrado, então os administradores podem rastrear quem e quando o conjunto de dados foi baixado;
- Qualquer solicitação de conjunto de dados também é registrada pelo mesmo motivo
O clean-command do pacote NÃO DEVE ser usado durante uma instalação em produção, caso contrário, apagará todas as alterações registradas. Se executado, apagará os logs anteriores ao tempo especificado no arquivo /config/activitylog.php.
A tabela ActivityLog tem a seguinte estrutura:
causer_typeecauser_idserão o usuário que fez a alteraçãosubject_typeesubject_idserão o modelo e registro alteradoslog_name- para agrupar logs e permite consultasdescription- um tanto redundante com log_name em um contexto OpenDataBio.properties- armazena as mudanças, por exemplo, e a mudança de identificação terá um log como:
{
"attributes":
{
"person_id":"2",
"taxon_id":"1424",
"modifier":"2",
"biocollection_id":"1",
"biocollection_reference":"1234",
"notes":"A new fake note has been inserted",
"date":"2020-02-08"},
"old":{
"person_id":674,
"taxon_id":1413,
"date":"1995-00-00",
"modifier":0,
"biocollection_id":null,
"notes":null,
"biocollection_reference":null
}
}
5 - Como contribuir
Reportar bugs e sugerir melhorias
Criar um issue em um dos repositórios GitLab abaixo, dependendo do problema.
Antes de postar, verifique se o que você quer relatar, perguntar ou propor já não está num issue aberto.
Identifique seu problema com uma ou mais etiquetas.
Issues para software
Issues para o pacote do R
Issues para este site de documentação
Colabore com o desenvolvimento, traduções de idiomas e documentos
Esperamos que este projeto cresça de forma colaborativa, necessário para o seu desenvolvimento e utilização no longo prazo. Portanto, colaboradores são bem-vindos para ajudar a corrigir e melhorar o OpenDataBio. A lista de problemas ou melhorias é um lugar para começar a saber o que é necessário fazer. Você pode também contribuir com Tutorias, melhorando a documentação.
As seguintes diretrizes são recomendadas se você deseja colaborar:
- Comunique-se com o administrador do repositório OpenDataBio indicando em quais questões deseja trabalhar e junte-se à equipe de desenvolvimento.
- Faça um
Forkdo repositório - Boa prática criar um
branchpara guardar suas modificações ou adições - Quando estiver satisfeito com os resultados, faça uma solicitação de
pull-requestao mantenedor do projeto para revisar sua contribuição e mesclá-la com o código do repositório. Consulte a Ajuda do GitLab para obter mais informações sobre pull requests.
Diretivas de programação
- Use a instalação do docker para desenvolvimento, que compartilhada entre todos os desenvolvedores facilita a depuração. A biblioteca Laravel-Datatables é incompatível com
php artisan serve, então este comando não deve ser usado. - Este software deve aderir ao Controle de Versão Semântico, a partir da versão 0.1.0-alpha1. O pacote R complementar e a Documentação (este site) devem seguir um esquema de controle de versão semelhante. Ao alterar a versão, uma tag de lançamento (release) deve ser criada com a versão antiga.
- Todas as variáveis e funções devem ser nomeadas em Inglês, com as entidades e campos relacionados ao banco de dados sendo nomeados no singular. Todas as tabelas (quando apropriado) devem ter uma coluna “id” e as chaves estrangeiras devem fazer referência à tabela base com o sufixo “_id”, exceto em casos de autojunções (como “taxon.parent_id”) ou chaves estrangeiras polimórficas. O id de cada tabela tem tipo INT e deve ser autoincrementado.
- Use a classe laravel migration para adicionar qualquer modificação à estrutura do banco de dados. A migração deve incluir, se aplicável, a manipulação de dados existentes, permitindo upgrades.
- Use camelCase para métodos (ou seja, relacionamentos) e snake_case para funções.
- Documente o código com comentários e crie páginas de documentação neste site, se necessário.
- Deve haver uma estrutura para armazenar quais plugins estão instalados em uma determinada versão do banco de dados quais são as versões de sistema compatíveis.
- Este sistema usa o Laravel Mix para compilar o código SASS e JavaScript usado. Se você adicionar ou modificar esses arquivos, utilize
npm run proddepois de fazer qualquer alteração nesses arquivos.
Colabore com a documentação
Tutoriais para lidar com tarefas específicas são bem vindos!
Para criar um tutorial:
Forko repositório de documentação. Ao clonar este repositório ou do seu fork inclua a opção de submódulo para obter também o repositório de tema Docsy incluído. Você precisará de Hugo para executar este site em seu localhost.- Crie um
branchpara confirmar suas modificações ou adições - Adicione seu tutorial:
- Crie uma pasta dentro de
contents/{lang}/docs/Tutorialsusando kebab-case para o nome da pasta. Ex.primeiro-tutorial - Você pode criar um tutorial em um único idioma ou em vários idiomas. Basta colocá-lo na pasta correta
- Dentro da pasta criada, crie um arquivo chamado
_index.mde crie o conteúdo de markdown com seu tutorial. - Você pode começar copiando o conteúdo de um tutorial já incluído ou veja a documentação do Docsy
- Quando estiver satisfeito com os resultados, faça uma solicitação de pull para pedir ao mantenedor do projeto para revisar sua contribuição e mesclá-la com o repositório. Consulte a Ajuda do GitLab para obter mais informações sobre o uso de solicitações pull.
Colabore com traduções
Você pode ajudar com as traduções da interface do aplicativo ou deste site com a documentação. Se quiser ter um novo idioma para sua instalação, compartilhe sua tradução, criando um pull request com os novos arquivos.
Novo idioma para a interface da web:
- faça um fork e crie um branch para o repositório principal
- crie uma pasta para o novo idioma usando o Código ISO 639-1 dentro da pasta
resources/lang
cd opendatabio
cd resources/lang
cp en es
- traduza todos os valores para todas as variáveis dentro de todos os arquivos na nova pasta (pode usar a tradução do google para começar, apenas certifique-se de que os nomes das variáveis não sejam traduzidos, caso contrário, não funcionará).
- adicione o idioma ao array em
config/languages.php - adicionar o idioma à tabela de languages do banco de dados criando uma migração laravel
- solicite um pull request
Novo idioma para o site de documentação
- faça um fork e crie um branch para o repositório de documentação
- crie uma pasta para o novo idioma usando o Código ISO 639-1 dentro da pasta
content
bash
cd opendatabio.gitlab.io
cd content
cp pt es
- verifique todos os arquivos dentro da pasta e traduza onde necessário (pode usar a tradução do google, apenas certifique-se de traduzir apenas o que pode ser traduzido)
- Veja se funciona bem na sua máquina local (precisa installar Hugo e servir digitando
hugo servena pasta do site, que ficará visível pelo navegador no endereço http://localhost:1313/. - empurre para o seu branch e faça um pull request
Relações polimóficas
Algumas das relações dentro da OpenDataBio são mapeadas usando Relações polimórficas. Elas são indicadas em um modelo por ter um campo terminando em _id e um campo terminando em _type. Por exemplo, todos os Objetos Centrais podem ter Medições, e essas relações são estabelecidas na tabela measurements pelas colunas measured_id e measured_type, o primeiro armazenando a id do modelo relacionado, o segundo é a classe do modelo medido em strings como ‘App\Models\Individual’, ‘App\Models\Voucher’, ‘App\Models\Taxon’, ‘App\Models\Location’.
Imagens do modelo conceitual
A maioria das figuras para explicar o modelo de dados foram geradas usando Laravel ER Diagram Generator, que permite mostrar todos os métodos implementados em cada modelo e não apenas os links diretos da tabela:
Para gerar essas figuras, um comando personalizado php artisan foi gerado. Esse commando está definido no arquivo app/Console/Commands/GenerateOdbErds.php.
Para atualizar as figuras siga os seguintes passos:
- As figuras são configuradas no arquivo
config/erd-generator-odb.php. Existem muitas opções adicionais para personalizar as figuras alterando ou adicionando variáveis graphviz ao arquivoconfig/erd-generator-base.php. - O comando personalizado é
php artisan odb: erd {$ model}, onde model é a chave dos arrays emconfig / erd-generator-odb.php, ou a palavra" all “, para regenerar todas as figuras doc.`bash cd opendatabio fazer ssh php artisan odb: erd all` - As figuras serão salvas em
storage / app / public / dev-imgs - Copie as novas imagens para a pasta do site de documentação. Eles precisam ser colocados em
contents / {lang} / concepts / {subfolder}para todos os idiomas e nas respectivas subpastas.
6 - Tutoriais
Este espaço do site OpenDataBio é para estudos de caso de uso do OpenDataBio pela interface, mas principalmente usando o OpenDataBio R package. Como contribuir com um tutorial.
6.1 - Obter dados via R
O pacote Opendatabio-R foi criado para permitir aos usuários interagir com um servidor OpenDataBio, para obter (GET) dados ou importar (POST) dados para a base de dados. Este tutorial é um exemplo básico de como obter dados.
Configure a conexão
- Configure a conexão com o servidor OpenDataBio usando a função
odb_config()do pacote. Os parâmetros mais importantes para esta função sãobase_url, que deve apontar para a URL da API do seu servidor OpenDataBio etoken, que é o token de acesso usado para autenticar seu usuário. - O
tokensó é necessário para obter dados de conjuntos de dados que possuem uma das políticas de acesso restrito. Os dados dos conjuntos de dados de acesso público podem ser extraídos sem a especificação do token. - Seu token está disponível em seu perfil na interface web
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
A configuração mais avançada envolve a definição de uma versão de API específica, um agente de usuário personalizado ou outros cabeçalhos HTTP, mas isso não é coberto aqui.
Teste sua conexão
A função odb_test() pode ser usada para verificar se a conexão foi bem sucedida e se
seu usuário foi identificado corretamente:
odb_test(cfg)
#will output
Host: https://opendb.inpa.gov.br/api/v0
Versions: server 0.9.1-alpha1 api v0
$message
[1] "Success!"
$user
[1] "admin@example.org"
Como alternativa, você pode especificar esses parâmetros como variáveis de sistema. Antes de iniciar o R, configure isso em seu shell (ou adicione ao final de seu arquivo .bashrc):
export ODB_TOKEN="YourToken"
export ODB_BASE_URL="https://opendb.inpa.gov.br/api"
export ODB_API_VERSION="v0"
Obter dados
Verifique a [Referência rápida da API GET]/docs/api/quick-reference) para obter uma lista completa de endpoints e parâmetros de solicitação.
Para dados de acesso público o token é opcional. Abaixo dois exemplos para Localidades e Taxons que são de acesso aberto. Siga um raciocínio semelhante para usar os demais endpoints. Veja a ajuda do pacote em R para todas as funções odb_get_{endpoint} disponíveis.
Obtendo nomes de táxons
Consulte GET API Taxon Endpoint para uma lista dos parâmetros de solicitação e uma lista de campos de resposta.
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
#get id for a taxon
mag.id = odb_get_taxons(params=list(name='Magnoliidae',fields='id,name'),odb_cfg = cfg)
#use this id to get all descendants of this taxon
odb_taxons = odb_get_taxons(params=list(root=mag.id$id,fields='id,scientificName,taxonRank,parent_id,parentName'),odb_cfg = cfg)
head(odb_taxons)
Algo como, dependo da sua base:
id scientificName taxonRank parent_id parentName
1 25 Magnoliidae Clado 20 Angiosperms
2 43 Canellales Ordem 25 Magnoliidae
3 62 Laurales Ordem 25 Magnoliidae
4 65 Magnoliales Ordem 25 Magnoliidae
5 74 Piperales Ordem 25 Magnoliidae
6 93 Chloranthales Ordem 25 Magnoliidae
Obtendo locais
Consulte GET API Location Endpoint para os parâmetros de solicitação e uma lista de campos de resposta.
Obtenha alguns campos listando todas as Unidades de Conservação (adm_level=99) registradas no servidor:
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
odblocais = odb_get_locations(params = list(fields='id,name,parent_id,parentName',adm_level=99),odb_cfg = cfg)
head(odblocais)
Se o servidor usar os dados de seed fornecidos o resultado será:
id name
1 5628 Estação Ecológica Mico-Leão-Preto
2 5698 Área de Relevante Interesse Ecológico Ilha do Ameixal
3 5700 Área de Relevante Interesse Ecológico da Mata de Santa Genebra
4 5703 Área de Relevante Interesse Ecológico Buriti de Vassununga
5 5707 Reserva Extrativista do Mandira
6 5728 Floresta Nacional de Ipanema
parent_id parentName
1 6 São Paulo
2 6 São Paulo
3 6 São Paulo
4 6 São Paulo
5 6 São Paulo
6 6 São Paulo
Pegar as parcelas importadas no tutorial de localidades.
Para obter um objeto espacial no R, use a função readWKT do pacote rgeos.
library(rgeos)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
locais = odb_get_locations(params=list(adm_level=100),odb_cfg = cfg)
locais[,c('id','locationName','parentName')]
colnames(locais)
for(i in 1:nrow(locais)) {
geom = readWKT(locais$footprintWKT[i])
if (i==1) {
plot(geom,main=locais$locationName[i],cex.main=0.8)
axis(side=1,cex.axis=0.5)
axis(side=2,cex.axis=0.5,las=2)
} else {
plot(geom,main=locais$locationName[i],add=T,col='red')
}
}
Figura gerada:

6.2 - Importar dados via R
O pacote Opendatabio-R foi criado para permitir aos usuários interagir com um servidor OpenDataBio, tanto para obter (GET) dados ou para importar (POST) dados para o banco de dados. Este tutorial é um exemplo básico de como importar dados.
Configure a conexão
- Configure a conexão com o servidor OpenDataBio usando a função
odb_config()do pacote. Os parâmetros mais importantes para esta função sãobase_url, que deve apontar para a URL da API do seu servidor OpenDataBio etoken, que é o token de acesso usado para autenticar seu usuário. - O
tokensó é necessário para obter dados de conjuntos de dados que possuem uma das políticas de acesso restrito. Os dados dos conjuntos de dados de acesso público podem ser extraídos sem a especificação do token. - Seu token está disponível em seu perfil na interface web
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
#create a config object
cfg = odb_config(base_url=base_url, token = token)
#test connection
odb_test(cfg)
Importar Dados (POST API)
Verifique a [Referência rápida da API]/docs/api/quick-reference) para obter uma lista completa dos POST endpoints e os campos necessários para importação de dados.
Funções de importação OpenDataBio-R
Todas as funções de importação têm a mesma assinatura: o primeiro argumento é um data.frame com os dados a serem importados, e o segundo parâmetro é um objeto de configuração gerado por odb_config.
Ao escrever uma solicitação de importação, verifique os documentos da API POST para entender quais colunas podem ser declaradas no data.frame.
Todas as funções de importação retornam um id do job, que pode ser usado para verificar se o job ainda está em execução, se terminou com sucesso ou se encontrou um erro. Este id de trabalho pode ser usado nas funções odb_get_jobs(), odb_get_affected_ids() e odb_get_log(), para encontrar detalhes sobre a submissão de importação (job). Você também pode ver o log em sua lista de trabalhos do usuário na interface da web.
Atenção
Ordem é importante - a importação correta de dados pode depender de registros já registrados. Por exemplo, importar um indivíduo com uma identidade de táxon requer o nome do táxon registrado no banco de dados, então primeiro valide sua lista de táxons usando a API GET e, em seguida, importe os registros para o Indivíduos.Trabalhando com datas e datas incompletas
Para Indivíduos, Vouchers e identificações, você pode usar datas incompletas.
O formato de data usado no OpenDataBio é AAA-MM-DD (ano - mês - dia), portanto, uma entrada válida seria 2018-05-28.
Particularmente em dados históricos, o dia (ou mês) exato pode não ser conhecido, então você pode substituir esses campos por NA: ‘1979-05-NA’ significa “um dia desconhecido, em maio de 1979” e ‘1979-NA- NA ‘significa “dia e mês desconhecidos, 1979”. Você não pode adicionar uma data para a qual tenha apenas o dia, mas pode, se tiver apenas o mês, se for realmente significativo de alguma forma.
6.2.1 - Importar Localidades
OpenDataBio é distribuído com um conjunto de dados de localidades para o Brasil, que inclui estados, municípios, unidades de conservação federais, terras indígenas e os biomas.
Trabalhar com dados espaciais é uma área delicada, por isso tentamos tornar o fluxo de trabalho para inserir Localidades o mais fácil possível.
Se você deseja fazer upload dos limites administrativos de um país, você também pode baixar um arquivo geojson em OSM-Boundaries e carregue-o diretamente através da interface da web. Ou use o repositório GADM exemplificado abaixo.
A importação é direta, mas os principais problemas a serem considerados:
- OpenDataBio armazena as geometrias de localidades usando representação de texto conhecido (WKT).
- As localidades são hierárquicas, portanto, uma localidade DEVE estar completamente dentro de sua localidade pai. O método de importação tentará detectar as localidades pai com base em sua geometria. Portanto, você não precisa informar um pai. No entanto, às vezes a localidade pai e a localidade filho compartilham uma borda ou têm pequenas erros que evitam a detecção. Portanto, se a importação não colocar o local onde você esperava, pode-se atualizar ou importar informando o pai correto. Quando você informar a localidade pai, uma segunda verificação será realizada adicionando um buffer à localidade pai e deverá resolver o problema.
- Os polígonos de países podem ser importados sem detecção ou definição dos pais, e registros marítimos podem ser vinculados a um pai, mesmo que não estejam contidos no polígono pai. Isso requer informar um campo específico (
ismarine) e deve ser usado nestes casos. - Padronizar a geometria para uma projeção comum de uso no sistema. Fortemente recomendado o uso de EPSG:4326 WGS84. Padronize antes de importar.
- Considere enviar seus polígonos político-administrativos antes de adicionar PONTOS, PLOTS ou TRANSECTOS específicos;
- Unidades de Conservação, Territórios Indígenas e Camadas Ambientais podem ser adicionados como locais e serão tratados como casos especiais, pois alguns desses locais abrangem diferentes unidades administrativas. Portanto, uma localidade de tipo POINT, PLOT ou TRANSECTpode pertencer a umA UC, umA TI e muitas camadas ambientais se estas estiverem armazenadas no banco de dados. Essas localidades relacionadas são detectadas automaticamente a partir da geometria da localidade.
Verifique a POST API de localidades para entender quais colunas podem ser declaradas ao importar localidades.
Adm_level define o tipo de localidade
O nível administrativo (adm_level) de um local é um número:
2para país;3à10como outras como ‘áreas administrativas’, seguindo a convenção OpenStreeMap para facilitar a importação de dados externos e traduções locais (À SER IMPLEMENTADO) . Portanto, para o Brasil, os códigos são (Estados = 4, Municípios = 8);999para locais de ‘POINT’ como waypoints GPS;101para transectos100é o código para PARCELAS e SUBPARCELAS;99é o código para Unidades de Conservação98para Territórios Indígenas97para polígonos ambientais (por exemplo, Floresta Ombrofila Densa ou Bioma Amazônia)
Importando polígonos espaciais
Limites administrativos do GADM
Limites administrativos também podem ser importados sem sair de R, obtendo dados de GDAM e usando as funções odb_import*
library(raster)
library(opendatabio)
#download áreas administrativas do GADM para um país
#get country codes
crtcodes = getData('ISO3')
bra = crtcodes[crtcodes$NAME%in%"Brazil",]
#define a path where to save the downloaded spatial data
path = "GADMS"
dir.create(path,showWarnings = F)
#o número de admin_levels em cada país varia
#obter todos os níveis existentes em seu computador
runit =T
level = 0
while(runit) {
ocrt <- try(getData('GADM', country=bra, level=level,path=path),silent=T)
if (class(ocrt)=="try-error") {
runit = FALSE
}
level = level+1
}
#read downloaded data and format to odb
files = list.files(path, full.name=T)
locations.to.odb = NULL
for(f in 1:length(files)) {
ocrt <- readRDS(files[f])
#class(ocrt)
#convert the SpatialPolygonsDataFrame to OpenDataBio format
ocrt.odb = opendatabio:::sp_to_df(ocrt) #only for GADM data
locations.to.odb = rbind(locations.to.odb,ocrt.odb)
}
#see without geometry
head(locations.to.odb[,-ncol(locations.to.odb)])
#you may add a note to location
locations.to.odb$notes = paste("Source gdam.org via raster::get_data()",Sys.Date())
#adjust the adm_level to fit the OpenStreeMap categories
ff = as.factor(locations.to.odb$adm_level)
(lv = levels(ff))
levels(ff) = c(2,4,8,9)
locations.to.odb$adm_level = as.vector(ff)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=locations.to.odb,odb_cfg=cfg)
Atenção: você pode querer verificar se há exclusividade de nome + pai em vez de apenas nome, já que nome + pai é uma combinação única. Você não pode salvar dois locais com o mesmo nome dentro do mesmo pai.
Example usando um shapefile
library(rgdal)
#read your shape file
path = 'mymaps'
file = 'myshapefile.shp'
layer = gsub(".shp","",file,ignore.case=TRUE)
data = readOGR(dsn=path, layer= layer)
#you may reproject the geometry to standard of your system if needed
data = spTransform(data,CRS=CRS("+proj=longlat +datum=WGS84"))
#convert polygons to WKT geometry representation
library(rgeos)
geom = rgeos::writeWKT(data,byid=TRUE)
#prep import
names = data@data$name #or the column name of the data
shape.to.odb = data.frame(name=names,geom=geom,stringsAsFactors = F)
#need to add the admin_level of these locations
shape.to.odb$admin_level = 2
#and may add parent and note if your want
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=shape.to.odb,odb_cfg=cfg)
Example importando de um KML
#read file as SpatialPolygonDataFrame
file = "myfile.kml"
file.exists(file)
mykml = readOGR(file)
geom = rgeos::writeWKT(mykml,byid=TRUE)
#prep import
names = mykml@data$name #or the column name of the data
to.odb = data.frame(name=names,geom=geom,stringsAsFactors = F)
#need to add the admin_level of these locations
to.odb$admin_level = 2
#and may add parent or any other valid field
#import
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=to.odb,odb_cfg=cfg)
Importar Parcelas e SubParcelas
Parcelas e transectos são casos especiais no OpenDataBio:
- Eles podem ser definidas com uma geometria do tipo Polygon ou LineString, respectivamente;
- Ou eles podem ser registrados apenas como localidaes de tipo POINT. Nesse caso, o OpenDataBio criará o polígono ou linestring para você;
- Dimensões (x e y) são armazenadas em metros para PARCELAS. Portanto elas devem ser quadradas ou retangulares.
- SubParcelas são localidades do tipo PARCELA tendo outra localidade do tipo PARCELA como pai e também devem ter posições cartesianas (startX, startY) dentro da localidade pai além das dimensões. A posição cartesiana refere-se às posições X e Y dentro da PARCELA pai e, portanto, DEVE ser menor do que o pai X e Y.
- SubParcela é o único tipo de localidade que pode ser registrado sem uma coordenada geográfica ou geometria, que será calculada a partir da geometria da PARCELA pai usando os valores startx e starty.
Parcela e SubParcela - exemplo 01
Você precisa de pelo menos uma coordenada geográfica para registrar uma localidade do tipo PLOT. A geometria (ou latitude e longitude) não pode estar vazia.
Este exemplo registra uma parcela em Manaus de 100x100m, informando sua geometria e depois importa algumas subparcelas sem especificação de geometria.
#geometry of a plot in Manaus
southWestCorner = c(-59.987747, -3.095764)
northWestCorner = c(-59.987747, -3.094822)
northEastCorner = c(-59.986835,-3.094822)
southEastCorner = c(-59.986835,-3.095764)
geom = rbind(southWestCorner,northWestCorner,northEastCorner,southEastCorner)
library(sp)
geom = Polygon(geom)
geom = Polygons(list(geom), ID = 1)
geom = SpatialPolygons(list(geom))
library(rgeos)
geom = writeWKT(geom)
to.odb = data.frame(name='A 1ha example plot',x=100,y=100,notes='a fake plot',geom=geom, adm_level = 100,stringsAsFactors=F)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=to.odb,odb_cfg=cfg)
Aguarde alguns segundos e, em seguida, importe subtramas para esta plotagem.
#importar subparcelas de 20x20m para a PARCELA acima sem indicar uma geometria.
(parent = odb_get_locations(params = list(name='A 1ha example plot',fields='id,name',adm_level=100),odb_cfg = cfg))
sub1 = data.frame(name='sub plot 40x40',parent=parent$id,x=20,y=20,adm_level=100,startx=40,starty=40,stringsAsFactors=F)
sub2 = data.frame(name='sub plot 0x0',parent=parent$id,x=20,y=20,adm_level=100,startx=0,starty=0,stringsAsFactors=F)
sub3 = data.frame(name='sub plot 80x80',parent=parent$id,x=20,y=20,adm_level=100,startx=80,starty=80,stringsAsFactors=F)
dt = rbind(sub1,sub2,sub3)
#import
odb_import_locations(data=dt,odb_cfg=cfg)
Capturas de tela das parcelas importadas
Abaixo capturas de tela para as parcelas importadas com o código acima
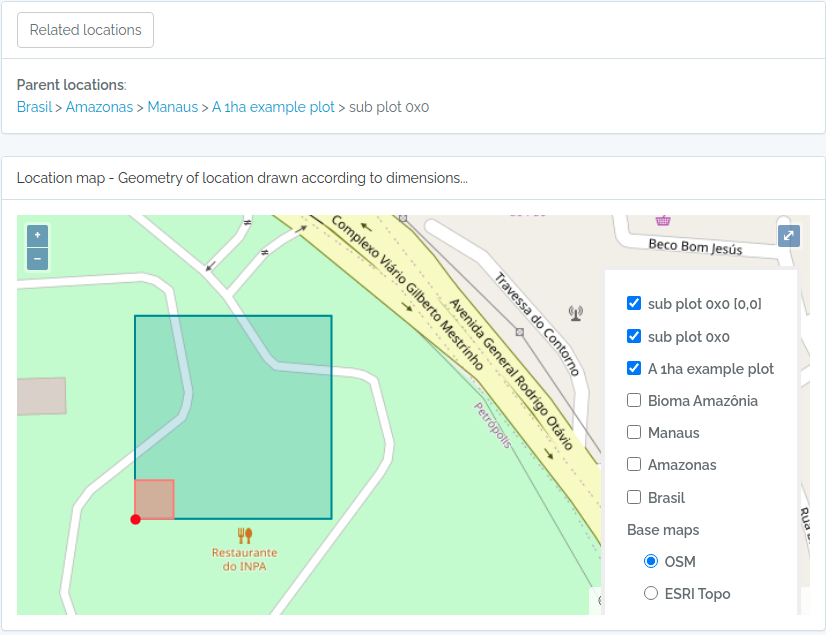
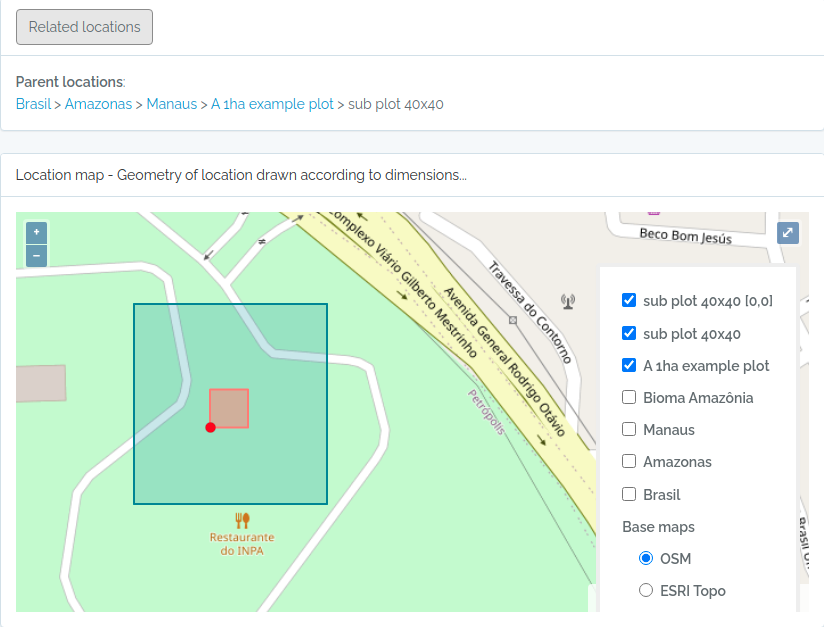
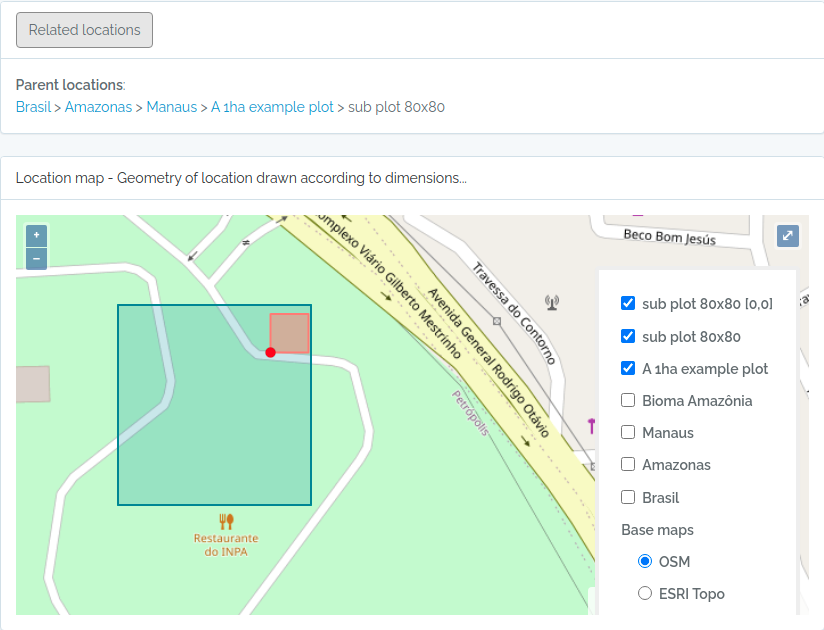
Parcela e SubParcela - exemplo 02
Importe uma parcela e suas subparcelas tendo apenas:
- a coordenada geográfica de um único ponto, representando a coordenada [0,0] da parcela.
- um azimute ou ângulo da direção da parcela (se não informar, Norte será usado
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
#the plot
geom = "POINT(-59.973841 -2.929822)"
to.odb = data.frame(name='Example Point PLOT',x=100, y=100, azimuth=45,notes='OpenDataBio point plot example',geom=geom, adm_level = 100,stringsAsFactors=F)
odb_import_locations(data=to.odb,odb_cfg=cfg)
#define 20x20 subplots cartesian coordinates
x = seq(0,80,by=20)
xx = rep(x,length(x))
yy = rep(x,each=length(x))
names = paste(xx,yy,sep="x")
#importar esses subplots sem ter uma geometria, mas especificando a localidade pai
parent = odb_get_locations(params = list(name='Example Point PLOT',adm_level=100),odb_cfg = cfg)
to.odb = data.frame(name=names,startx=xx,starty=yy,x=20,y=20,notes="OpenDataBio 20x20 subplots example",adm_level=100,parent=parent$id)
odb_import_locations(data=to.odb,odb_cfg=cfg)
#obter os locais importados usando o parâmetro root
locais = odb_get_locations(params=list(root=parent$id),odb_cfg = cfg)
locais[,c('id','locationName','parentName')]
colnames(locais)
for(i in 1:nrow(locais)) {
geom = readWKT(locais$footprintWKT[i])
if (i==1) {
plot(geom,main=locais$locationName[i],cex.main=0.8,col='yellow')
axis(side=1,cex.axis=0.7)
axis(side=2,cex.axis=0.7,las=2)
} else {
plot(geom,add=T,border='red')
}
}
A figura gerada acima:
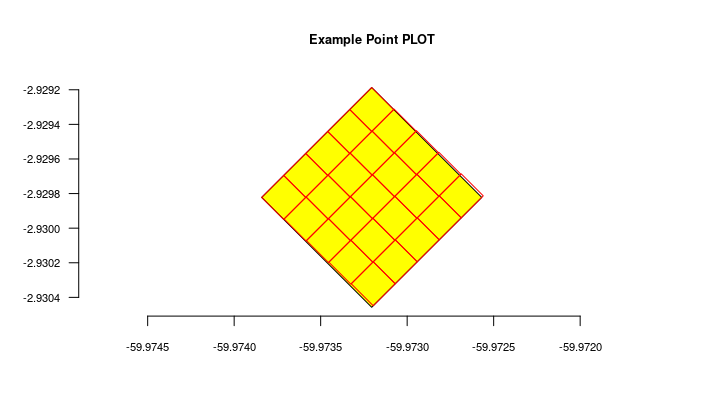
Importar transectos
Este código importará dois transectos, um definido por uma geometria (LINESTRING), e o outro apenas por uma única coordenada geográfica (POINT). Veja as figuras abaixo para o resultado importado.
#geometry of transect in Manaus
#read trail from a kml file
#library(rgdal)
#file = "acariquara.kml"
#file.exists(file)
#mykml = readOGR(file)
#library(rgeos)
#geom = rgeos::writeWKT(mykml,byid=TRUE)
#above will output:
geom = "LINESTRING (-59.9616459699999993 -3.0803612500000002, -59.9617394400000023 -3.0805952900000002, -59.9618530300000003 -3.0807376099999999, -59.9621049400000032 -3.0808563200000001, -59.9621949100000009 -3.0809758500000002, -59.9621587999999974 -3.0812666800000001, -59.9621092399999966 -3.0815010400000000, -59.9620656999999966 -3.0816403499999998, -59.9620170600000009 -3.0818584699999998, -59.9620740699999999 -3.0819864099999998)";
#prep data frame
#o valor y refere-se a um buffer em metros aplicado à trilha
#y é usado para validar a inserção de indivíduos relacionados
to.odb = data.frame(name='A trail-transect example',y=20, notes='OpenDataBio transect example',geom=geom, adm_level = 101,stringsAsFactors=F)
#import
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
odb_import_locations(data=to.odb,odb_cfg=cfg)
#IMPORTA UM SEGUNDO TRANSECTO SEM GEOMETRIA
# então você precisa informar o valor x, que é o comprimento do transecto
#ODB irá mapear este transecto orientado pelo parâmetro azimute (sul no exemplo abaixo)
#point geometry = ponto inicial
geom = "POINT(-59.973841 -2.929822)"
to.odb = data.frame(name='A transect point geometry',x=300, y=20, azimuth=180,notes='OpenDataBio point transect example',geom=geom, adm_level = 101,stringsAsFactors=F)
odb_import_locations(data=to.odb,odb_cfg=cfg)
locais = odb_get_locations(params=list(adm_level=101),odb_cfg = cfg)
locais[,c('id','locationName','parentName','levelName')]
O código acima resultará nas duas localidades a seguir:
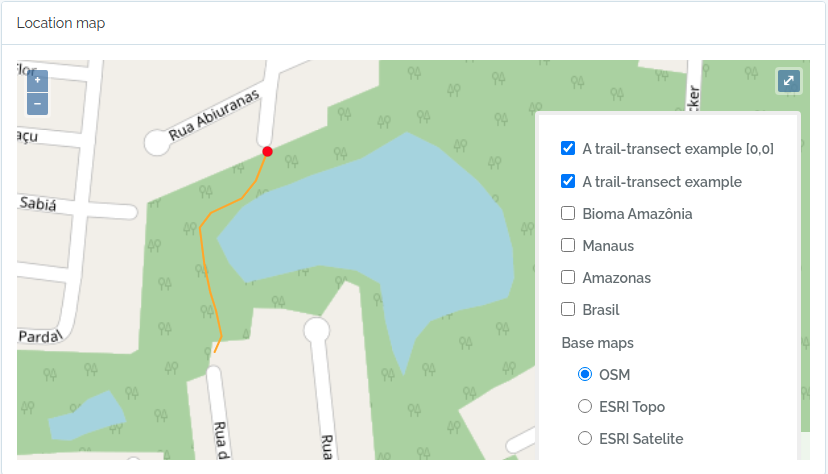
6.2.2 - Importar Taxons
Atenção
- Para uma importação bem-sucedida de nomes taxomicos publicados você só precisa fornecer um
name, sendo o nome completo para espécies e infra-espécies (por exemplo, Licaria canella tenuicarpa, Ocotea delicata), da mesma forma quecanonicalNamedo GBIF, ou seja, não inclui os ranks infra-específicos; - Não há necessidade de importar táxons pais para nomes publicados, OpenDataBio faz isso para você. O processo de importação usará o
nameque você informou para recuperar informações de táxons de um repositório de nomenclatura: atualmente Tropicos, GBIF BackBone Taxonomy e ZOOBANK. Se o nome do táxon for encontrado, ele obterá automaticamente as informações necessárias e também todos os táxons pais (ou nomes aceitos se estiver marcado como sinônimo) conforme necessário, e armazenará no banco de dados para você. Se onamefor inválido, ou seja, é sinônimo de outro nome, ele será importado, mas marcado como inválido. - Se informar
parente for diferente do detectado pela API o pai informado tem preferência. - Para espécies não publicadas
level,parent, e umapersonouauthor_idtambém devem ser fornecidos; - A API externa usará uma consulta difusa durante a pesquisa de GBIF se não encontrar uma correspondência exata, permitindo detectar até mesmo quando está com erros ortográficos. No entanto, erros de grafia nos nomes podem impedir que um táxon seja armazenado.
- A taxonomia é hierárquica e a estrutura dentro do OpenDataBio pode ser semelhante a uma árvore. Portanto, você pode adicionar, se quiser, qualquer nó da árvore da vida na tabela Taxon. Para isso, você precisa usar o nível do táxon
Clade. Mas observe que isso pode não ser relevante se você não vincular dados a esses nomes de clados. - Como a verificação da nomenclatura é demorada, seja paciente.
OpenDataBio é distribuído com uma tabela semente de Taxons, que inclui a raiz de todos os reinos e alguns nós da filogenia das plantas, ou seja, o os nós da àrvore no nível de ordem para Angiospermas - APWeb Tree.
Um exemplo simples de nome publicado
Os scripts abaixo foram testados, estando a base já populada com até o nível de Ordem para Angiospermas.
Na tabela de táxons, as famílias Moraceae, Lauraceae e Solanaceae ainda não estavam registradas:
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
exists = odb_get_taxons(params=list(root="Moraceae,Lauraceae,Solanaceae"),odb_cfg=cfg)
Retornou:
data frame with 0 columns and 0 rows
Agora importe algumas espécies e uma infraespécie para as famílias acima, especificando seu nome completo (canonicalName):
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
spp = c("Ficus schultesii", "Ocotea guianensis","Duckeodendron cestroides","Licaria canella tenuicarpa")
splist = data.frame(name=spp)
odb_import_taxons(splist, odb_cfg=cfg)
Agora verifique se foi importado (note o argumento root):
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
exists = odb_get_taxons(params=list(root="Moraceae,Lauraceae,Chrysobalanaceae"),odb_cfg=cfg)
head(exists[,c('id','scientificName', 'taxonRank','taxonomicStatus','parentNameUsage')])
Retorno:
id scientificName taxonRank taxonomicStatus parentName
1 252 Moraceae Family accepted Rosales
2 253 Ficus Genus accepted Moraceae
3 254 Ficus schultesii Species accepted Ficus
4 258 Solanaceae Family accepted Solanales
5 259 Duckeodendron Genus accepted Solanaceae
6 260 Duckeodendron cestroides Species accepted Duckeodendron
7 255 Lauraceae Family accepted Laurales
8 256 Ocotea Genus accepted Lauraceae
9 257 Ocotea guianensis Species accepted Ocotea
10 261 Licaria Genus accepted Lauraceae
11 262 Licaria canella Species accepted Licaria
12 263 Licaria canella subsp. tenuicarpa Subspecies accepted Licaria canella
Observe que embora tenhamos especificado apenas os nomes das espécies e infra-espécies, a API importou também toda a hierarquia parental necessária até a família, porque as ordens já estavam registradas.
Um exemplo de nome publicado inválido
O nome Licania octandra pallida (Chrysobalanaceae) foi recentemente movido com sinônimo de Leptobalanus octandrus pallidus.
O roteiro a seguir exemplifica o que acontece nesses casos.
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
#lets check
exists = odb_get_taxons(params=list(root="Chrysobalanaceae"),odb_cfg=cfg)
exists
#in this test returns an empty data frame
#data frame with 0 columns and 0 rows
#now import
spp = c("Licania octandra pallida")
splist = data.frame(name=spp)
odb_import_taxons(splist, odb_cfg=cfg)
#see the results
exists = odb_get_taxons(params=list(root="Chrysobalanaceae"),odb_cfg=cfg)
exists[,c('id','scientificName', 'taxonRank','taxonomicStatus','parentName')]
Retorno:
id scientificName taxonRank taxonomicStatus parentName
1 264 Chrysobalanaceae Family accepted Malpighiales
2 265 Leptobalanus Genus accepted Chrysobalanaceae
3 267 Leptobalanus octandrus Species accepted Leptobalanus
4 269 Leptobalanus octandrus subsp. pallidus Subspecies accepted Leptobalanus octandrus
5 266 Licania Genus accepted Chrysobalanaceae
6 268 Licania octandra Species invalid Licania
7 270 Licania octandra subsp. pallida Subspecies invalid Licania octandra
Observe que, embora tenhamos especificado apenas um nome de infra-espécie, a API importou também toda a hierarquia pai necessária até a família e, como o nome é inválido, também importou o nome aceito para esta infra-espécie e seus pais.
Uma espécie ou morfotipo não publicado
É comum ter nomes de espécies locais não publicados (morfotipos) para plantas em parcelas, ou ainda trabalhos taxonômicos ainda não publicados. As designações não publicadas são específicas do projeto e, portanto, DEVEM também fornecer um autor, pois diferentes projetos podem usar o mesmo código ‘sp.1’ ou ‘sp.A’ para seus táxons não publicados.
Você pode vincular um nome não publicado como qualquer nível de táxon e não precisa usar a lógica de gênero + espécie para atribuir um morfotipo para o qual o gênero ou taxonomia de nível superior é indefinida. Por exemplo, você pode armazenar um nível de ’espécie’ com o nome ‘Indet sp.1’ e parent_name ‘Laurales’, se a determinação formal de nível mais baixo que você tem é o nível de ordem. Neste exemplo, não há necessidade de armazenar um gênero Indet e táxons da família Indet apenas para contabilizar este morfotipo não identificado.
##assign an unpublished name for which you only know belongs to the Angiosperms and you have this node in the Taxon table already
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
#check that angiosperms exist
odb_get_taxons(params=list(name='Angiosperms'),odb_cfg = cfg)
#if it is there, start creating a data.frame to import
to.odb = data.frame(name='Morphotype sp.1', parent='Angiosperms', stringsAsFactors=F)
#get species level numeric code
to.odb$level=odb_taxonLevelCodes('species')
#you must provide an author that is a Person in the Person table. Get from server
odb.persons = odb_get_persons(params=list(search='João Batista da Silva'),odb_cfg=cfg)
#found
head(odb.persons)
#add the author_id to the data.frame
#NOTE it is not author, but author_id or person)
#this makes odb understand it is an unpublished name
to.odb$author_id = odb.persons$id
#import
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
odb_import_taxons(to.odb,odb_cfg = cfg)
Verifique o registro importado:
exists = odb_get_taxons(params=list(name='Morphotype sp.1'),odb_cfg = cfg)
exists[,c('id','scientificName', 'taxonRank','taxonomicStatus','parentName','scientificNameAuthorship')]
Algumas colunas para o registro importado:
id scientificName taxonRank taxonomicStatus parentName scientificNameAuthorship
1 276 Morphotype sp.1 Species unpublished Angiosperms João Batista da Silva - Silva, J.B.D.
Importar um clado publicado
Você pode adicionar um clado Taxon e pode referenciar uma publicação usando a entrada bibkey. Portanto, é possível armazenar de fato todos os nós relevantes de qualquer filogenia na hierarquia do Taxon.
#parent já deve estar armazenado
odb_get_taxons(params=list(name='Pagamea'),odb_cfg = cfg)
#define o clado a ser armazenado
to.odb = data.frame(name='Guianensis core', parent_name='Pagamea', stringsAsFactors=F)
to.odb$level = odb_taxonLevelCodes('clade')
#adicione uma referência à publicação onde está publicado
#importe a referência do bib para o banco de dados de antemão
odb_get_bibreferences(params(bibkey='prataetal2018'),odb_cfg=cfg)
to.odb$bibkey = 'prataetal2018'
#então adicione nomes de espécies válidos como filhos deste clado em vez do nível de gênero
children = data.frame(name = c('Pagamea guianensis','Pagamea angustifolia','Pagamea puberula'),stringsAsFactors=F)
children$parent_name = 'Guianensis core'
children$level = odb_taxonLevelCodes('species')
children$bibkey = NA
#merge
to.odb = rbind(to.odb,children)
#import
odb_import_taxons(to.odb,odb_cfg = cfg)
6.2.3 - Importar Pessoas
Ver o POST Persons API docs para entender quais colunas podem ser declaradas ao importar Pessoas.
A API verificará por abreviações idênticas, que é a única restrição da classe Pessoa. Abreviações são exclusivas e duplicações não são permitidas. Isso não impede que os dados baixados de repositórios tenham abreviações ou nomes completos diferentes para a mesma pessoa. Portanto, você deve padronizar os dados secundários antes de importá-los para o servidor para minimizar esses erros comuns.
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ"
cfg = odb_config(base_url=base_url, token = token)
one = data.frame(full_name='Adolpho Ducke',abbreviation='DUCKE, A.',notes='Grande botânico da Amazônia',stringsAsFactors = F)
two = data.frame(full_name='Michael John Gilbert Hopkins',abbreviation='HOPKINKS, M.J.G.',notes='Curador herbário INPA',stringsAsFactors = F)
to.odb= rbind(one,two)
odb_import_persons(to.odb,odb_cfg=cfg)
#pode adicionar um email
Pegar os dados
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
cfg = odb_config(base_url=base_url)
persons = odb_get_persons(odb_cfg=cfg)
persons = persons[order(persons$id,decreasing = T),]
head(persons,2)
resultado:
id full_name abbreviation email institution notes
613 1582 Michael John Gilbert Hopkins HOPKINKS, M.J.G. <NA> NA Curador herbário INPA
373 1581 Adolpho Ducke DUCKE, A. <NA> NA Grande botânico da Amazônia
6.2.4 - Importar Variáveis
Atenção
- Recomendamos fortemente que você use a interface da web para inserir variáveis uma por uma. Use importações em lote somente se você tiver muitas variáveis.
- Antes de importar qualquer variável certifique-se de que ela já não esteja cadastrada no sistema com um nome diferente. Prevenir a duplicação é importante porque a classe Trait é compartilhada entre todos os usuários de uma instalação OpenDataBio. Isso também significa que, uma vez que uma variável é usada para Medições, você só poderá alterar a definição da variável, por exemplo, um nome de uma categoria, se você for o único usuário que inseriu medições para a variável. Caso contrário, outra pessoa usou essa definição e você não poderá alterá-la.
- export_name deve ser exclusivo em uma única instalação ODB. Este nome é usado ao exportar dados e em formulários, e você deve considerar torná-lo o mais curto e informativo (de definição) possível. Além disso, a API validará o nome da exportação - deve ser camelCase, PascalCase ou snake_case, e você não pode adicionar espaços ou qualquer caractere especial (acentos). Ex. ‘dbh’, ‘dbhPom’, ’leafLength’, ‘LeafLength’ ou ’leaf_length’, respectivamente.
As características podem ser importadas usando odb_import_traits().
Leia atentamente o Traits POST API.
Tipos de variáveis
Veja odb_traitTypeCodes() para os códigos numéricos possíveis tipos de variáveis
Traduções Nome da Variável e de Categorias
Os campos name e description podem ter um dos seguintes conteúdos:
- usando o código do idioma como chaves:
list("en" = "Diameter at Breast Height","pt-br" ="Diâmetro a Altura do Peito") - ou usando os nomes dos idiomas como chaves:
list("English" ="Diameter at Breast Height","Portuguese" ="Diâmetro a Altura do Peito").
O campo categories deve incluir para cada categoria + classificação + idioma os seguintes campos: lang= misto - obrigatório, o id, código ou nome do idioma da traduçãoname= string - obrigatório, o nome da categoria traduzido obrigatório (name + rank + lang deve ser único)rank= número - obrigatório, a classificação é importante para indicar a mesma categoria entre os idiomas e define variáveis ordinais;description= string - opcional para categorias, uma definição da categoria.
Isso pode ser formatado como um data.frame e colocado na coluna categories de outro data.frame:
data.frame(
rbind(
c("lang"="en","rank"=1,"name"="small","description"="smaller than 1 cm"),
c("lang"="pt-br","rank"=1,"name"="pequeno","description"="menor que 1 cm"),
c("lang"="en","rank"=2,"name"="big","description"="bigger than 10 cm"),
c("lang"="pt-br","rank"=2,"name"="grande","description"="maior que 10 cm")
),
stringsAsFactors=FALSE
)
Variável quantitativa
Para variáveis quantitativas para valores integers ou real (type 0 ou 1).
odb_traitTypeCodes()
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#do this first to build a correct data.frame as it will include translations list
to.odb = data.frame(type=1,export_name = "dbh", unit='centimeters',stringsAsFactors = F)
#add translations (note double list)
#format is language_id = translation (and the column be a list with the translation lists)
to.odb$name[[1]]= list('1' = 'Diameter at breast height', '2' = 'Diâmetro à altura do peito')
to.odb$description[[1]]= list('1' = 'Stem diameter measured at 1.3m height','2' = 'Diâmetro do tronco medido à 1.3m de altura')
#measurement validations
to.odb$range_min = 10 #this will restrict the minimum measurement value allowed in the trait
to.odb$range_max = 400 #this will restrict the maximum value
#measurements can be linked to (classes concatenated by , or a list)
to.odb$objects = "Individual,Voucher,Taxon" #makes no sense link such measurements to Locations
to.odb$notes = 'this is quantitative trait example'
#import
odb_import_traits(to.odb,odb_cfg=cfg)
Variável categórica
- Deve incluir categorias. A única diferença entre as características ordinais e categóricas é que as categorias ordinais terão um rank, ou ordem. Observe que as variáveis ordinais são semiquantitativas e, portanto, se você tiver categorias, pergunte-se se elas não são realmente ordinais e registre de acordo.
- Assim como o
namee adescriptionda variável, as categorias também podem ter traduções em diferentes idiomas, e você DEVE inserir as traduções para os idiomas disponíveis (odb_get_languages ()) para que a variável esteja acessível em todos os idiomas. O inglês é obrigatório, portanto, pelo menos o nome em inglês deve ser informado. As categorias podem ter uma descrição associada, mas às vezes o nome da categoria é autoexplicativo, portanto, as descrições das categorias não são obrigatórias.
odb_traitTypeCodes()
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#do this first to build a correct data.frame as it will include translations list
#do this first to build a correct data.frame as it will include translations list
to.odb = data.frame(type=3,export_name = "specimenFertility", stringsAsFactors = F)
#trait name and description
to.odb$name = data.frame("en"="Specimen Fertility","pt-br"="Fertilidade do especímene",stringsAsFactors=F)
to.odb$description = data.frame("en"="Kind of reproductive stage of a collected plant","pt-br"="Estágio reprodutivo de uma amostra de planta coletada",stringsAsFactors=F)
#categories (if your trait is ORDINAL, the add categories in the wanted order here)
categories = data.frame(
rbind(
c('en',1,"Sterile"),
c('pt-br',1,"Estéril"),
c('en',2,"Flowers"),
c('pt-br',2,"Flores"),
c('en',3,"Fruits"),
c('pt-br',3,"Frutos"),
c('en',4,"Flower buds"),
c('pt-br',4,"Botões florais")
),
stringsAsFactors =FALSE
)
colnames(categories) = c("lang","rank","name")
#descriptions not included for categories as they are obvious,
# but you may add a 'description' column to the categories data.frame
#objects for which the trait may be used for
to.odb$objects = "Individual,Voucher"
to.odb$notes = 'a fake note for a multiselection categorical trait'
to.odb$categories = list(categories)
#import
odb_import_traits(to.odb,odb_cfg=cfg)
Variável tipo LINK
Um variável de tipo LINK permite vincular um Táxon ou Voucher como uma medição de outro objeto. Por exemplo, você pode conduzir um inventário de plantas para o qual possui apenas contagens para o táxon associado a uma localidade. Portanto, você pode criar uma variável do tipo LINK, que permitirá que você armazene os valores de contagem para qualquer Táxon como medições para um local específico (PONTO, POLÍGONO).
Use a interface ou siga o exemplo para as demais variáveis.
Variáveis do tipo texto ou cor
Variáveis de texto permitem o armazenamento de observações textuais. A cor permitirá apenas códigos de cores.
odb_traitTypeCodes()
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
to.odb = data.frame(type=5,export_name = "taxonDescription", stringsAsFactors = F)
#trait name and description
to.odb$name = data.frame("en"="Taxonomic descriptions","pt-br"="Descrições taxonômicas",stringsAsFactors=F)
to.odb$description = data.frame("en"="Taxonomic descriptions from the literature","pt-br"="Descrições taxonômicas da literatura",stringsAsFactors=F)
#will only be able to use this trait for a measurment associated with a Taxon
to.odb$objects = "Taxon"
#import
odb_import_traits(to.odb,odb_cfg=cfg)
Variáveis espectrais
Variáveis espectrais são específicos para dados espectrais. Você deve especificar a amplitude dos números de ondas para os quais você pode ter dados de absorbância ou refletância e o comprimento dos espectros a serem armazenados como medições para permitir a validação durante a entrada. Portanto, para cada intervalo e espaçamento dos valores espectrais que você tem, uma variável ESPECTRAL diferente deve ser criada.
Use a interface ou siga o exemplo para as demais variáveis.
6.2.5 - Importar Indivíduos & Vouchers
Attention
- Você pode importar Vouchers juntamente com o registro do Indivíduo. Use o endpoint POST-Voucher apenas para indivíduos já registrados, caso contrário, siga abaixo para importar indivíduos e seus vouchers de uma só vez.
- Os indivíduos podem ter uma identificação taxonômica própria ou uma identificação taxonômica dependente
- Os indivíduos podem ter vários locais, mas ao registrar o indivíduo, um único local é necessário. Você pode então importar locais adicionais usando o Individual-location POST API.
Indivíduos podem ser importados usando odb_import_individuals() e vouchers com odb_import_vouchers().
Leia atentamente o Individual POST API e o Voucher POST API.
Exemplo simples
Inventando dados para 1 registro de um indivíduo:
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#the number in the aluminium tag in the forest
to.odb = data.frame(tag='3405.L1', stringsAsFactors=F)
#the collectors (get ids from the server)
(joao = odb_get_persons(params=list(search='joao batista da silva'),odb_cfg=cfg)$id)
(ana = odb_get_persons(params=list(search='ana cristina sega'),odb_cfg=cfg)$id)
#ids concatenated by | pipe
to.odb$collector = paste(joao,ana,sep='|')
#tagged date (lets use an incomplete).
to.odb$date = '2018-07-NA'
#lets place in a Plot location imported with the Location post tutorial
plots = odb_get_locations(params=list(name='A 1ha example plot'),odb_cfg=cfg)
head(plots)
to.odb$location = plots$id
#relative position within parent plot
to.odb$x = 10.4
to.odb$y = 32.5
#or could be
#to.odb$relative_position = paste(x,y,sep=',')
#taxonomic identification
taxon = 'Ocotea guianensis'
#check that exists
(odb_get_taxons(params=list(name='Ocotea guianensis'),odb_cfg=cfg)$id)
#person that identified the individual
to.odb$identifier = odb_get_persons(params=list(search='paulo apostolo'),odb_cfg=cfg)$id
#or you also do to.odb$identifier = "Assunção, P.A.C.L."
#the used form only guarantees the persons is there.
#may add modifers as well [may need to use numeric code instead]
to.odb$modifier = 'cf.'
#or check with to see you spelling is correct
odb_detModifiers()
#and submit the numeric code instaed
to.odb$modifier = 3
#an incomplete identification date
to.odb$identification_date = list(year=2005)
#or to.odb$identification_date = "2005-NA-NA"
Importando esse indivíduo
odb_import_individuals(to.odb,odb_cfg = cfg)
#lets import this individual
odb_import_individuals(to.odb,odb_cfg = cfg)
#check the job status
odb_get_jobs(params=list(id=130),odb_cfg = cfg)
Ops, esqueci de informar um dataset e meu usuário não tem um dataset default definido.
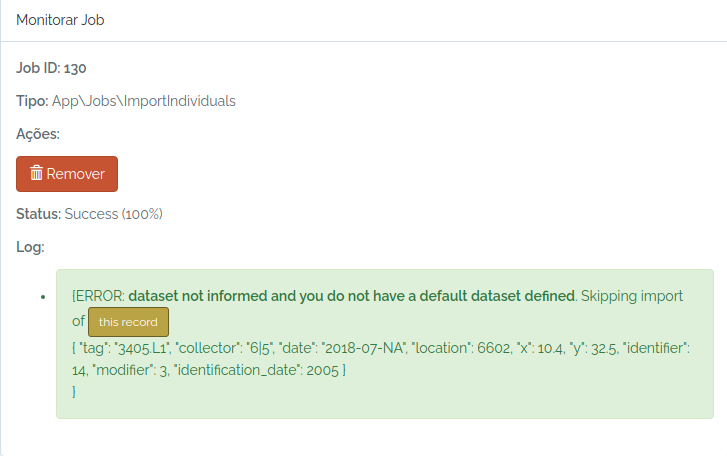
Então, eu apenas informo um conjunto de dados existente e tento novamente:
dataset = odb_get_datasets(params=list(name="Dataset test"),odb_cfg=cfg)
dataset
to.odb$dataset = dataset$id
odb_import_individuals(to.odb,odb_cfg = cfg)
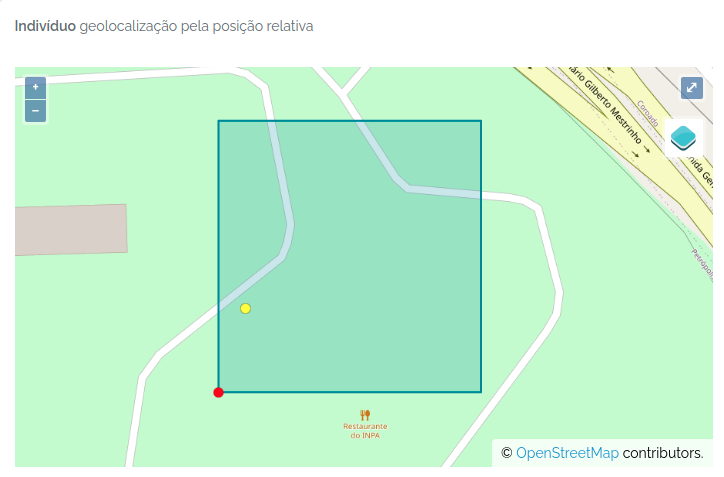
Importando Indivíduos e Vouchers de uma vez
Indivíduos são o objeto real que possui a maior parte das informações relacionadas aos Vouchers, que são amostras em uma Biocoleção. Portanto, você pode importar um registro de um indivíduo com a especificação de um ou mais vouchers.
#a fake plant record somewhere in the Amazon
aplant = data.frame(taxon="Duckeodendron cestroides", date="2021-09-09", latitude=-2.34, longitude=-59.845,angle=NA,distance=NA, collector="Oliveira, A.A. de|João Batista da Silva", tag="3456-A",dataset=1)
#a fake set of vouchers for this individual
herb = data.frame(biocollection=c("INPA","NY","MO"),biocollection_number=c("12345A","574635","ANOTHER FAKE CODE"),biocollection_type=c(2,3,3))
#add this dataframe to the object
aplant$biocollection = NA
aplant$biocollection = list(herb)
#another fake plant
asecondplant = data.frame(taxon="Ocotea guianensis", date="2021-09-09", latitude=-2.34, longitude=-59.89,angle=240,distance=50, collector="Oliveira, A.A. de|João Batista da Silva", tag="3456",dataset=1)
asecondplant$biocollection = NA
#merge the fake data
to.odb = rbind(aplant,asecondplant)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
odb_import_individuals(to.odb, odb_cfg=cfg)
Verifique os dados importados
O script acima criou registros para o modelo Indivíduo e Voucher:
#get the imported individuals using a wildcard
inds = odb_get_individuals(params = list(tag='3456*'),odb_cfg = cfg)
inds[,c("basisOfRecord","scientificName","organismID","decimalLatitude","decimalLongitude","higherGeography") ]
Retorna:
basisOfRecord scientificName organismID decimalLatitude decimalLongitude higherGeography
1 Organism Ocotea guianensis 3456 - Oliveira - UnnamedPoint_5989234 -2.3402 -59.8904 Brasil | Amazonas | Rio Preto da Eva
2 Organism Duckeodendron cestroides 3456-A - Oliveira - UnnamedPoint_5989234 -2.3400 -59.8900 Brasil | Amazonas | Rio Preto da Eva
E Vouchers
#get the vouchers imported with the first plant data
vouchers = odb_get_vouchers(params = list(individual=inds$id),odb_cfg = cfg)
vouchers[,c("basisOfRecord","scientificName","organismID","collectionCode","catalogNumber") ]
Retorna:
basisOfRecord scientificName occurrenceID collectionCode catalogNumber
1 PreservedSpecimens Duckeodendron cestroides 3456-A - Oliveira -INPA.12345A INPA 12345A
2 PreservedSpecimens Duckeodendron cestroides 3456-A - Oliveira -MO.ANOTHER FAKE CODE MO ANOTHER FAKE CODE
3 PreservedSpecimens Duckeodendron cestroides 3456-A - Oliveira -NY.574635 NY 574635
Importar Vouchers para Indivíduos registrados
Atenção
- Vouchers são amostras de Indivíduos depositados em BioColeções. Se a Biocoleção for um recurso genético, o Voucher pode representar apenas uma amostra de DNA ou tecido.
- Portanto, os Vouchers devem pertencer a um Indivíduo e à uma Biocoleção, que são as informações obrigatórias para o cadastro de Vouchers. Coletores, data de coleta, localização e identidade taxonômica podem simplesmente ser extraídos do cadastro do Indivíduo.
- O Voucher pode ter coletor, número de coletor e data de coleta próprios, distintos do Indivíduo a que pertence, quando necessário.
Os campos obrigatórios são:
individual= id do indivíduo ou nome completo (organismID);biocollection= sigla ou id da Biocoleção - useodb_get_biocollections()para verificar se está registrado, caso contrário, primeiro registre a Biocoleção no banco de dados;
Para campos adicionais veja Voucher POST API.
Uma importação de voucher simples
#a holotype voucher with same collector and date as individual
onevoucher = data.frame(individual=1,biocollection="INPA",biocollection_number=1234,biocollection_type=1,dataset=1)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
odb_import_vouchers(onevoucher, odb_cfg=cfg)
#get the imported voucher
voucher = odb_get_vouchers(params=list(individual=1),cfg)
vouchers[,c("basisOfRecord","scientificName","occurrenceID","collectionCode","catalogNumber") ]
Voucher diferente para um indivíduo
Dois vouchers para o mesmo Indivíduo, um com o mesmo coletor e data de coleta do Indivíduo, outro em data diferente e por outros cobradores.
#one with same date and collector as individual
one = data.frame(individual=2,biocollection="INPA",biocollection_number=1234,dataset=1,collector=NA,number=NA,date=NA)
#this one with different collector and date
two= data.frame(individual=2,biocollection="INPA",biocollection_number=4435,dataset=1,collector="Oliveira, A.A. de|João Batista da Silva",number=3456,date="1991-08-01")
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
to.odb = rbind(one,two)
odb_import_vouchers(to.odb, odb_cfg=cfg)
#get the imported voucher
voucher = odb_get_vouchers(params=list(individual=2),cfg)
voucher[,c("basisOfRecord","scientificName","occurrenceID","collectionCode","catalogNumber") ]
Registros importados:
basisOfRecord scientificName occurrenceID collectionCode catalogNumber recordedByMain
1 PreservedSpecimens Unidentified plot tree - Vicentini -INPA.1234 INPA 1234 Vicentini, A.
2 PreservedSpecimens Unidentified 3456 - Oliveira -INPA.4435 INPA 4435 Oliveira, A.A. de
6.2.6 - Importar Medições
Atenção
- A medição tem uma restrição: valores ou categorias de medição duplicados para o mesmo object_id e mesma data não serão importados, a menos que você especifique
duplicated = 1para o registro. - Para importar medições você precisa coletar algumas informações de outros modelos. Você precisa fornecer um
datasetpara o qual você tem permissões,trait_id(export_name ou id),value(o conteúdo varia dependendo do tipo de variável),date(uma data completa ou incompleta),person(pessoa que mediu),object_type(uma de taxon, individual, voucher ou location) eobject_id(o id do object_type). Para o tipo de variável LINK você deve fornecer uma colunalink_id, e ‘value’ torna-se opcional para este caso específico. - DICA: Para relacionar medições de diferentes variáveis, você pode adicionar palavaras chave campo de medição
notes. Por exemplo, você mede LeafLength e LeafWidth para as mesmas folhas, portanto, em ambas as medições, adicione “leaf1”, “leaf2” ao campo de nota para vinculá-los. Além disso, se você medir o DAP e o DAP.POM em um inventário de parcela florestal e se tiver vários troncos para uma planta, você pode adicionar “caule1”, “caule2” para permitir a correspondência de um DAP do caule com sua altura específica de medição. Esse tipo de link pode ser útil em análises. - Várias validações acontecerão durante a importação da medição. Considere verificar os dados localmente antes de enviar um trabalho (job) para facilitar a solução de problemas e garantir que os valores sejam consistentes com suas expectativas. Você não poderá ATUALIZAR nem EXCLUIR medidas com a API, portanto, evite importar medições que precisarão ser corrigidas posteriormente (uma por uma).
- Para características quantitativas, OpenDataBio não pode validar se suas medições estão na mesma unidade que a definição de variável. Portanto, verifique qual unidade a variável possui e converta seus dados de acordo antes de enviar.
As medições podem ser importadas usando odb_import_measurements(). Leia atentamente o Measurements POST API.
Variáveis Quantitativas
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#obter o id do trait do servidor (verifique se o trait existe)
#gerar alguns dados falsos para 10 medições
dbhs = sample(seq(10,100,by=0.1),10)
object_ids = sample(1:3,length(dbhs),replace=T)
dates = sample(as.Date("2000-01-01"):as.Date("2000-03-31"),length(dbhs))
dates = lapply(dates,as.Date,origin="1970-01-01")
dates = lapply(dates,as.character)
dates = unlist(dates)
to.odb = data.frame(
trait_id = 'dbh',
value = dbhs,
date = dates,
object_type = 'Individual',
object_id=object_ids,
person="Oliveira, A.A. de",
dataset = 1,
notes = "some fake measurements",
stringsAsFactors=F)
#isso só funcionará se a pessoa existir, os ids individuais existirem
#e se a característica com export_name = dbh existe
odb_import_measurements(to.odb,odb_cfg=cfg)
Baixar os dados importados:
dad = odb_get_measurements(params = list(dataset=1),odb_cfg=cfg)
dad[,c("id","basisOfRecord", "measured_type", "measured_id", "measurementType",
"measurementValue", "measurementUnit", "measurementDeterminedDate",
"datasetName", "license")]
id basisOfRecord measured_type measured_id measurementType measurementValue measurementUnit measurementDeterminedDate
1 1 MeasurementsOrFact App\\Models\\Individual 3 dbh 86.8 centimeters 2000-02-19
2 2 MeasurementsOrFact App\\Models\\Individual 2 dbh 84.8 centimeters 2000-03-25
3 3 MeasurementsOrFact App\\Models\\Individual 2 dbh 65.7 centimeters 2000-03-15
4 4 MeasurementsOrFact App\\Models\\Individual 3 dbh 88.0 centimeters 2000-03-05
5 5 MeasurementsOrFact App\\Models\\Individual 3 dbh 35.3 centimeters 2000-01-04
6 6 MeasurementsOrFact App\\Models\\Individual 2 dbh 36.0 centimeters 2000-03-23
7 7 MeasurementsOrFact App\\Models\\Individual 2 dbh 78.6 centimeters 2000-03-22
8 8 MeasurementsOrFact App\\Models\\Individual 2 dbh 69.7 centimeters 2000-03-09
9 9 MeasurementsOrFact App\\Models\\Individual 3 dbh 12.3 centimeters 2000-01-30
10 10 MeasurementsOrFact App\\Models\\Individual 3 dbh 14.7 centimeters 2000-01-18
datasetName license
1 Dataset test CC-BY 4.0
2 Dataset test CC-BY 4.0
3 Dataset test CC-BY 4.0
4 Dataset test CC-BY 4.0
5 Dataset test CC-BY 4.0
6 Dataset test CC-BY 4.0
7 Dataset test CC-BY 4.0
8 Dataset test CC-BY 4.0
9 Dataset test CC-BY 4.0
10 Dataset test CC-BY 4.0
Medidas categóricas
As categorias DEVEM ser informadas por seus ids ou nome no campo value. Para características CATEGORICAL ou ORDINAL, value deve ser um valor único. Para CATEGORICAL_MULTIPLE, value pode ser um ou vários ids de categorias ou nomes separados por um de | ou ; ou,.
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#a categorical trait
(odbtraits = odb_get_traits(params=list(name="specimenFertility"),odb_cfg = cfg))
#base line
to.odb = data.frame(trait_id = odbtraits$id, date = '2021-07-31', stringsAsFactors=F)
#the plant was collected with both flowers and fruits, so the value are the two categories
value = c("Flowers","Fruits")
#get categories for this trait if found
(cats = odbtraits$categories[[1]])
#check that your categories are registered for the trait and get their ids
value = cats[match(value,cats$name),'id']
#make multiple categories ids a string value
value = paste(value,collapse=",")
to.odb$value = value
#this links to a voucher
to.odb$object_type = "Voucher"
#get voucher id from API (must be ID).
#Search for collection number 1234
odbspecs = odb_get_vouchers(params=list(number="3456-A"),odb_cfg=cfg)
to.odb$object_id = odbspecs$id[1]
#get dataset id
odbdatasets = odb_get_datasets(params=list(name='Dataset test'),odb_cfg=cfg)
head(odbdatasets)
to.odb$dataset = odbdatasets$id
#person that measured
odbperson = odb_get_persons(params=list(search='ana cristina sega'),odb_cfg=cfg)
to.odb$person = odbperson$id
#import'
odb_import_measurements(to.odb,odb_cfg=cfg)
#get imported
dad = odb_get_measurements(params = list(voucher=odbspecs$id[1]),odb_cfg=cfg)
dad[,c("id","basisOfRecord", "measured_type", "measured_id", "measurementType",
"measurementValue", "measurementUnit", "measurementDeterminedDate",
"datasetName", "license")]
id basisOfRecord measured_type measured_id measurementType measurementValue measurementUnit
1 11 MeasurementsOrFact App\\Models\\Voucher 1 specimenFertility Flowers, Fruits NA
measurementDeterminedDate datasetName license
1 2021-07-31 Dataset test CC-BY 4.0
Cores
Para valores de cor, você deve inserir cor como seus códigos de strings RGB hexadecimal, para que possam ser renderizados graficamente e na interface da web. Portanto, qualquer valor de cor é permitido e seria mais fácil usar as cores da paleta na interface da web para inserir tais medidas. O pacote gplots permite que você converta nomes de cores em códigos RGB hexadecimais se você quiser fazer isso por meio da API.
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#get the trait id from the server (check that trait exists)
odbtraits = odb_get_traits(odb_cfg=cfg)
(m = match(c("fruitColor"),odbtraits$export_name))
#base line
to.odb = data.frame(trait_id = odbtraits$id[m], date = '2014-01-13', stringsAsFactors=F)
#get color value
#install.packages("gplots",dependencies = T)
library(gplots)
(value = col2hex("red"))
to.odb$value = value
#this links to a specimen
to.odb$object_type = "Individual"
#get voucher id from API (must be ID). Search for collection number 1234
odbind = odb_get_individuals(params=list(tag='3456'),odb_cfg=cfg)
odbind$scientificName
to.odb$object_id = odbind$id[1]
#get dataset id
odbdatasets = odb_get_datasets(params=list(name='Dataset test'),odb_cfg=cfg)
head(odbdatasets)
to.odb$dataset = odbdatasets$id
#person that measured
odbperson = odb_get_persons(params=list(search='ana cristina sega'),odb_cfg=cfg)
to.odb$person = odbperson$id
odb_import_measurements(to.odb,odb_cfg=cfg)

Medições de tipo de LINK de banco de dados
O tipo de variável LINK permite registrar dados de contagem, como por exemplo o número de indivíduos de uma espécie em um determinado local. Você deve fornecer o objeto vinculado (link_id), que pode ser um Táxon ou um Voucher, dependendo da definição da variável, e então o value recebe a contagem numérica.
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#get the trait id from the server (check that trait exists)
odbtraits = odb_get_traits(odb_cfg=cfg)
(m = match(c("taxonCount"),odbtraits$export_name))
#base line
to.odb = data.frame(trait_id = odbtraits$id[m], date = '2014-01-13', stringsAsFactors=F)
#the taxon to link the count value
odbtax = odb_get_taxons(params=list(name='Ocotea guianensis'),odb_cfg=cfg)
to.odb$link_id = odbtax$id
#now add the count value for this trait type
#this is optional for this measurement,
#however, it would make no sense to include such link without a count in this example
to.odb$value = 23
#a note to clarify the measurement (optional)
to.odb$notes = 'No voucher, field identification'
#this measurement will link to a location
to.odb$object_type = "Location"
#get location id from API (must be ID).
#lets add this to a transect
odblocs = odb_get_locations(params=list(adm_level=101,limit=1),odb_cfg=cfg)
to.odb$object_id = odblocs$id
#get dataset id
odbdatasets = odb_get_datasets(params=list(name='Dataset test'),odb_cfg=cfg)
head(odbdatasets)
to.odb$dataset = odbdatasets$id
#person that measured
odbperson = odb_get_persons(params=list(search='ana cristina sega'),odb_cfg=cfg)
to.odb$person = odbperson$id
odb_import_measurements(to.odb,odb_cfg=cfg)

Medições espectrais
value deve ser uma string de valores do espectro separados por “;”. O número de valores concatenados deve corresponder ao atributo value_length da variável, que é extraído da especificação do intervalo de número de onda para a variável. Portanto, você pode verificar isso facilmente antes de importar com odb_get_traits(params=list (fields = 'all', type = 9),cfg)
library(opendatabio)
base_url="https://opendb.inpa.gov.br/api"
token ="GZ1iXcmRvIFQ" #this must be your token not this value
cfg = odb_config(base_url=base_url, token = token)
#read a spectrum
spectrum = read.table("1_Sample_Planta-216736_TAG-924-1103-1_folha-1_abaxial_1.csv",sep=",")
#second column are NIR leaf absorbance values
#the spectrum has 1557 values
nrow(spectrum)
#[1] 1557
#collapse to single string
value = paste(spectrum[,2],collapse = ";")
substr(value,1,100)
#[1] "0.6768057;0.6763237;0.6755353;0.6746023;0.6733549;0.6718447;0.6701176;0.6682984;0.6662288;0.6636459;"
#get the trait id from the server (check that trait exists)
odbtraits = odb_get_traits(odb_cfg=cfg)
(m = match(c("driedLeafNirAbsorbance"),odbtraits$export_name))
#see the trait
odbtraits[m,c("export_name", "unit", "range_min", "range_max", "value_length")]
#export_name unit range_min range_max value_length
#6 driedLeafNirAbsorbance absorbance 3999.64 10001.03 1557
#must be true
odbtraits$value_length[m]==nrow(spectrum)
#[1] TRUE
#base line
to.odb = data.frame(trait_id = odbtraits$id[m], value=value, date = '2014-01-13', stringsAsFactors=F)
#this links to a voucher
to.odb$object_type = "Voucher"
#get voucher id from API (must be ID).
#search for a collection number
odbspecs = odb_get_vouchers(params=list(number="3456-A"),odb_cfg=cfg)
to.odb$object_id = odbspecs$id[1]
#get dataset id
odbdatasets = odb_get_datasets(params=list(name='Dataset test'),odb_cfg=cfg)
to.odb$dataset = odbdatasets$id
#person that measured
odbperson = odb_get_persons(params=list(search='adolpho ducke'),odb_cfg=cfg)
to.odb$person = odbperson$id
#import
odb_import_measurements(to.odb,odb_cfg=cfg)
Texto e notas
Basta adicionar o texto ao campo value e proceder como para os outros tipos de variável.